摘 要:本研究以亚麻(Linum usitatissimum)种质‘84-3’为材料克隆LuFAD7基因,利用生物信息学方法对该基因序列特征进行分析,通过定量逆转录聚合酶链反应(quantitative reverse transcription polymerase chain reaction,qRT-PCR)技术对其表达模式进行分析,并比较油用亚麻种质‘84-3’和纤用亚麻种质‘87-3’中LuFAD7氨基酸序列差异。结果表明,LuFAD7编码序列(coding sequence,CDS)全长为1179bp,共编码392个氨基酸,分子进化树显示亚麻与红花亚麻(Linum grandiflorum)亲缘关系最近,qRT-PCR得出LuFAD7基因在开花后20d的种子中高表达。此外,LuFAD7氨基酸序列在2个不同种质中存在18处氨基酸位点差异,其中有10处氨基酸差异位点位于脂肪酸脱饱和酶结构域。本研究结果促进了对LuFAD7功能及亚麻酸合成机理的认识,为进一步研究提供了理论指导。
关键词:亚麻;LuFAD7;基因克隆;生物信息学分析;表达分析
亚麻(Linum usitatissimum L.),亚麻科(Linaceae)亚麻属(Linum),自花授粉的一年生二倍体草本植物,具有耐寒、耐旱、耐贫瘠等特点,是西北、华北地区的特色作物,是一种优良的纤维作物和油料作物,因其富含纤维素的茎纤维(纤维亚麻)和其种子油(油用亚麻)而在世界范围内广泛种植。随着社会肥胖人群和其他慢性疾病增加,亚麻中因含有高不饱和脂肪酸含量而受到人类关注,有益于人类健康。亚麻籽含油率大约在35%~45%(王维义等,2020),棕榈酸(palmitic acid,PAL)(C16:0)、硬脂酸(stearic acid,STE)(C18:0)、油酸(oleic acid,OLE)(C18:1)、亚油酸(linoleic acid,LIO)(C18:2)和亚麻酸(linolenic acid,LIN)(C18:3)是亚麻籽油中含有的五种主要脂肪酸,其中α-亚麻酸含量高达50%以上(王树彦和李心文,2010),在降血脂血压、降胆固醇、预防糖病、防治癌症以及保护冠状动脉心脏疾病等方面具有重要疗效(赵利等,2006;吴素萍,2010),近年来随着人民生活水平的提升,越来越重视对营养素的摄入,亚麻籽油作为开发α-亚麻酸产品的理想材料,对亚麻油脂品质遗传改良具有生产意义,具有高α-亚麻酸含量的亚麻籽油具有治疗和营养保健品应用,为具有特定脂肪酸组成的油创造了新的市场,对亚麻功能性食品、药品等方面的开发及利用具有一定的经济和社会效益,在现代社会中具有非常大的食用价值。
在调控亚油酸合成亚麻酸途径中,ω-3脂肪酸脱氢酶发挥重要作用(Xue et al.,2018),分为定位于内质网中的FAD3和定位于质体的FAD7和FAD8,而FAD7是其重要组成部分(Lee et al.,2016)。目前已经从芝麻(Sesamum indicum) (Chellamuthu et al.,2022)、小麦(Triticum aestivum)叶片(Horiguchi et al.,1996)等物种中克隆出FAD7基因。在转基因烟草植物中,芝麻SiFAD7的表达导致α-亚麻酸含量增加,亚油酸水平降低(Bhunia et al.,2015)。已有研究表明,在烟草(Nicotiana tabacum)和番茄(Solanum lycopersicum)中分别过量表达CsFAD7和LeFAD7基因,均会导致α-亚麻酸含量增加,而亚油酸含量降低(刘训言,2006;刘春香等,2012)。由此可见,FAD7基因调控植物的脂肪酸组成。
目前FAD3在亚麻中有一定研究,但FAD7和FAD8在亚麻中的研究尚未见报道。研究学者通过基因克隆得到3个LuFAD3基因(LuFAD3A和LuFAD3B)(Vrinten et al.,2005)和LuFAD3C(Banik et al.,2011),LuFAD3A和LuFAD3B表达与亚麻酸积累相关。Urla等(2017)发现在水稻(Oryza sativa)中过表达LuFAD3,导致亚麻酸积累显著增加。本研究基于亚麻参考基因组数据,筛选出α-亚麻酸生物合成的关键基因LuFAD7,克隆其编码序列(coding sequence,CDS)全长,并对LuFAD7基因进行定量逆转录聚合酶链反应(quantitative reverse transcription polymerase chain reaction, qRT-PCR)和生物信息学分析,为LuFAD7在亚麻不饱和脂肪酸合成过程中的调控机制提供理论依据,为培育高α-亚麻酸含量的亚麻良种提供科学依据。
1结果与分析
1.1亚麻LuFAD7基因克隆
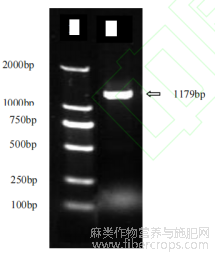
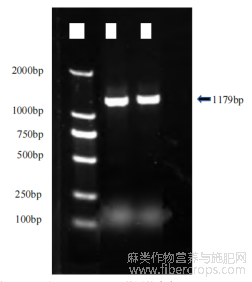
PCR扩增‘84-3’叶片cDNA后得到1179bp目的片段(图1),将该片段进行克隆测序并比对序列后显示,一致性为100%,表明克隆成功(图2)。
图1 亚麻‘84-3’品种LuFAD7基因的克隆
注:M:DL2000 DNA Marker;84:‘84-3’基因
图2 亚麻‘84-3’品种与LuFAD7氨基酸比对
1.2亚麻LuFAD7基因的生物信息学分析
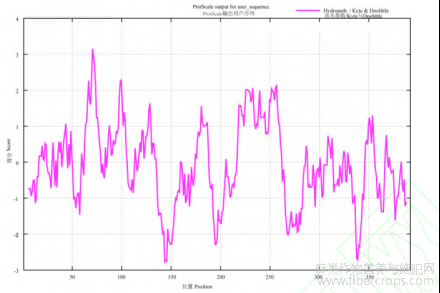
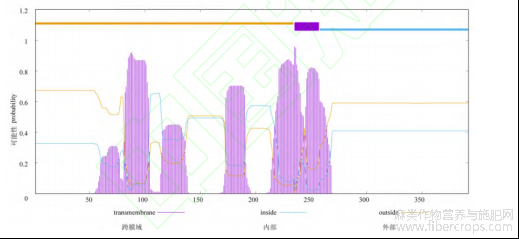
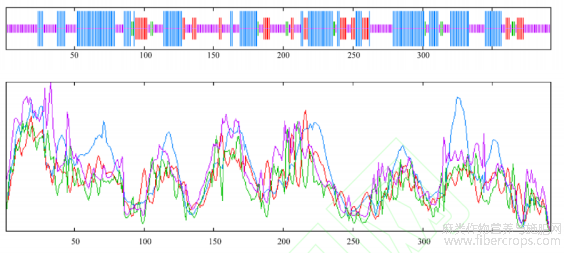
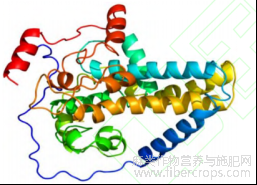
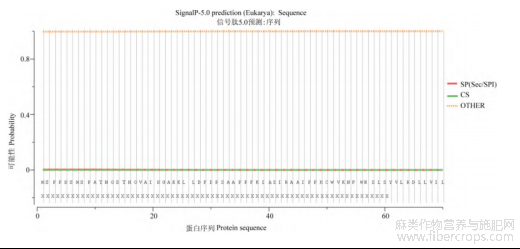
通过生物信息学对LuFAD7基因进行分析(表1),得出该基因CDS全长为1179bp,编码的氨基酸数为392氨基酸,原子总数6258,等电点为9.09,不稳定性指数为37.40,属于稳定蛋白质。此外,LuFAD7蛋白在第143位氨基酸出现最低值-2.800,亲水性最强,在第70位氨基酸出现最高值3.122,疏水性最强。整体看疏水氨基酸和亲水氨基酸分布较均匀,偏向为亲水蛋白(图3),在氨基酸235~257位存在1个跨膜结构域(图4),该蛋白含有Delta12-FADS-like特殊结构域,属于FADS家族蛋白(图5),二级结构预测发现,LuFAD7蛋白二级结构以无规则卷曲为主,占44.13%,其次是α-螺旋,占40.05%,还有11.99%的延伸链和3.83%的β-转角(图6),三级结构经预测得到的结果与二级结构预测结果相一致,都含有α-螺旋、β-转角、无规则卷曲和延伸链(图7)。该蛋白N端前70位氨基酸残基不存在信号肽,属于非分泌蛋白(图8),亚细胞定位于叶绿体和内质网。
表1 LuFAD7基因生物信息学分析
图3 LuFAD7蛋白亲疏水性分析
图4 LuFAD7蛋白跨膜结构域分析
图5 亚麻LuFAD7蛋白保守结构域预测
图6 亚麻LuFAD7蛋白的二级结构预测
注:蛋白序列位点,蓝色:α-螺旋;红色:延伸链;绿色:β-转角;紫色:无规则卷曲
图7 亚麻LuFAD7蛋白的三级结构预测
图8 亚麻LuFAD7蛋白的信号肽分析
注:SP(Sec/SPI):信号肽(由Sec易位子转运并被信号肽酶I切割);CS:剪切位置;OTHER:其它
1.3亚麻LuFAD7蛋白与其它物种进化树构建及蛋白保守基序分析
在NCBI中FAD7氨基酸序列与红花亚麻(Linum grandiflorum)、蓖麻(Ricinus communis)、拟南芥(Arabidopsis thaliana)等比对后发现(李永华等,2015),其序列一致性为73.71%(图9)。
图9 亚麻LuFAD7蛋白与其他植物FAD7蛋白的氨基酸序列同源比对
注:LuFAD7:亚麻;LgFAD7:红花亚麻;RcFAD7:蓖麻;AtFAD7:拟南芥;SlFAD7:番茄;CsFAD7:黄瓜;GmFAD7:大豆;HaFAD7:向日葵;BnFAD7:油菜;NtFAD7:烟草;PlFAD7:芍药;MdFAD7:苹果;CxmFAD7:菊花;CtFAD7:红花;StFAD7:马铃薯;图中不同的颜色:不同氨基酸残基的保守性;白色:氨基酸的保守性不到33%;青色:氨基酸的保守性为50%以上;粉红色:氨基酸的保守性为75%以上;深蓝色:氨基酸完全保守
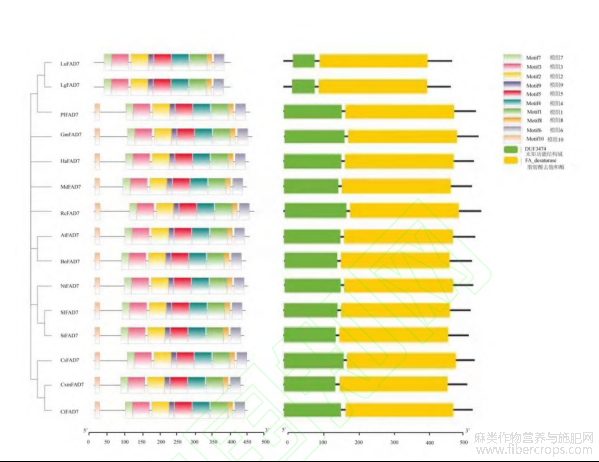
利用MEGA7.0软件,采用邻近法对亚麻LuFAD7蛋白与其他植物FAD7蛋白构建进化树,并整合了domain和10个Motif信息(图10),结果显示,亚麻LuFAD7蛋白与红花亚麻LgFAD7蛋白亲缘关系最近。通过MEME发现,LuFAD7蛋白10个Motif里面,Motif1~9是亚麻LuFAD7蛋白与各物种FAD7蛋白都含有的相同的保守基序,表明这九个基序是FAD7蛋白序列的重要组成部分,推测它们也是亚麻LuFAD7蛋白特有的功能基序,同时,亚麻LuFAD7蛋白与各物种FAD7蛋白结构相同,都含有DUF3474和FA_desaturase(图11)。
图10 亚麻LuFAD7蛋白进化树、保守基序及蛋白结构
注:LuFAD7:亚麻;LgFAD7:红花亚麻;RcFAD7:蓖麻;AtFAD7:拟南芥;SlFAD7:番茄;CsFAD7:黄瓜;GmFAD7:大豆;HaFAD7:向日葵;BnFAD7:油菜;NtFAD7:烟草;PlFAD7:芍药;MdFAD7:苹果;CxmFAD7:菊花;CtFAD7:红花;StFAD7:马铃薯;Motif1~9:模组1~9;DUF3474:未知功能结构域;FA_desaturase:脂肪酸去饱和酶
图11 亚麻LuFAD7蛋白预测的10个模组LOGO
1.4亚麻LuFAD7基因的表达分析
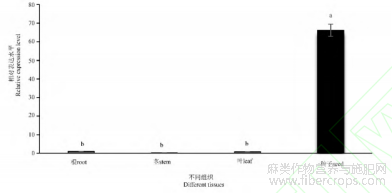
采用qRT-PCR对油用亚麻种质‘84-3’的根、茎、叶、种子进行LuFAD7基因表达量分析,基因表达量变化为:种子>根>叶>茎(图12)。由此可知,亚麻LuFAD7基因在种子中表达量较高,可能与油脂合成相关。
图12 亚麻LuFAD7基因组织特异性表达分析
注:所有实验数据均以三个独立生物学重复的平均值表示,采用SPSS软件对数据进行单因素方差分析;不同的字母表示同一组内显著不同的均值(P<0.05,Duncan's多重范围检验)。
1.5LuFAD7在不同发育时期表达特征分析
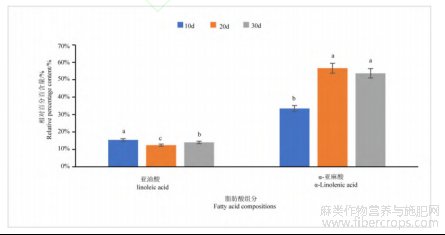
为了研究LuFAD7在亚麻种子不同发育时期的表达量,首先对亚麻‘84-3’授粉后10、20、30d的不同发育时期亚麻籽油进行亚油酸和α-亚麻酸含量测定,研究发现,授粉后10~20d亚麻酸含量从33.51%迅速增加到56.64%,30d又下降到53.72%(图13)。而授粉后10~20d亚油酸含量从15.39%下降到12.41%,30d缓慢上升到13.98%。
图13 ‘84-3’不同发育时期种子亚油酸和α-亚麻酸的含量
注:所有实验数据均以三个独立生物学重复的平均值表示,采用SPSS软件对数据进行单因素方差分析,不同的字母表示同一组内显著不同的均值(P<0.05,Duncan's多重范围检验)
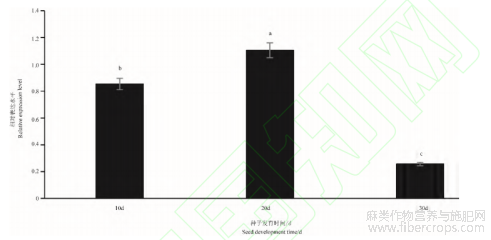
为进一步研究LuFAD7基因的表达量与脂肪酸积累的关系,对‘84-3’进行授粉后种子发育不同时期(10,20,30d)LuFAD7基因在种子中表达情况的研究。根据授粉后10、20、30d不同天数对种子进行取样,通过不同时期qRT-PCR可得出,LuFAD7基因在‘84-3’不同发育时期,其表达量先升高并于20d达到顶峰,随后下降(图14),该表达趋势与亚麻授粉后α-亚麻酸表达趋势相一致(图13),说明LuFAD7基因可能参与α-亚麻酸合成。
图14 LuFAD7在‘84-3’种子不同发育阶段的相对表达量
注:所有实验数据均以三个独立生物学重复的平均值表示,采用SPSS软件对数据进行单因素方差分析,不同的字母表示同一组内显著不同的均值(P<0.05,Duncan's多重范围检验)
1.6不同亚麻种质LuFAD7氨基酸差异分析
选用油用亚麻种质‘84-3’(母本,含油率45.77%),纤用亚麻种质‘87-3’(父本,含油率35.94%),PCR扩增‘84-3’和‘87-3’的叶片cDNA后均得到1179bp目的片段(图1),将该片段进行克隆测序并比对序列后显示,氨基酸序列在亚麻不同种质间存在95.42%相似度(图16)。‘87-3’发生氨基酸替换有15处位点,发生氨基酸缺失有3处位点(表2)。利用InterPro软件分析,发现LuFAD7蛋白在2个不同种质中有10处氨基酸差异位点位于脂肪酸脱饱和酶结构域。
表2 LuFAD7氨基酸差异分析
图15 ‘84-3’和‘87-3’LuFAD7基因的克隆
注:M:DL2000 DNA Marker;84:‘84-3’基因;87:‘87-3’基因
图16 ‘84-3’和‘87-3’LuFAD7氨基酸比对
注:红色方框标出:脂肪酸脱饱和酶结构域
2讨论
亚麻籽中富含的α-亚麻酸在调节血脂、降低血压、血糖,预防癌症等方面具有重要作用。因此,研究亚麻α-亚麻酸含量相关调控基因对亚麻分子植物育种以及优良品种的培育具有重要意义。目前不少学者在水稻和烟草中分别过量表达大豆GmFAD7和NtFAD7基因,均会导致α-亚麻酸含量显著上升(Anai et al.,2003;Orlova et al.,2003),与此同时,将FAD7基因在水稻中过表达后,也得到了一致的结论,使其α-亚麻酸含量从3.6×105μg/kg增加到1.006×107μg/kg(Liu et al.,2012)。在拟南芥中过表达SdFAD7基因,导致不饱和脂肪含量提高(宋玉双和隋娜,2019)。相反,在烟草中沉默NtFAD7基因,不饱和脂肪酸含量明显降低(Murakami et al.,2000)。
本研究发现,LuFAD7蛋白是亲水性的不稳定蛋白质,该蛋白含有Delta12-FADS-like保守结构域,属于FADS家族蛋白,分子进化树分析显示亚麻与红花亚麻FAD7蛋白亲缘关系最近。qRT-PCR分析发现,LuFAD7基因在‘84-3’不同组织表达量差异较大,其在种子中高表达,说明ALA合成相关基因多集中在种子中表达。不同时期qRT-PCR表明,LuFAD7基因在开花后20d的种子中高表达,这与前人的研究相一致(Vrinten et al.,2005)。本研究表明,在调控亚油酸合成α-亚麻酸的途径中,LuFAD7发挥重要的功能,LuFAD7的高表达促进亚油酸向α-亚麻酸转化,使α-亚麻酸积累,同时使亚油酸含量下降。进一步序列分析表明,LuFAD7蛋白在‘84-3’与‘87-3’两个品种中存在多处氨基酸差异,分别是15处位点发生氨基酸替换和3处位点发生氨基酸缺失,且发现‘84-3’在84~336氨基酸处、‘87-3’在82~334氨基酸处都含有脂肪酸脱饱和酶(FA_desaturase)结构域,其结构域中有10处氨基酸差异,这与ω-3脂肪酸脱氢酶在高、低油大豆品种中序列差异所含有脂肪酸脱饱和酶结构域相一致(刘文浩等,2008),推测亚麻不同种质间α-亚麻酸含量差异可能是由上述氨基酸残基的变化引起,这些差异是否直接影响亚麻脂肪酸生物合成还有待进一步验证。
3材料与方法
3.1植物材料
本试验所用材料为油用亚麻种质‘84-3’和纤用亚麻种质‘87-3’,播种于温室,光照处理为光照16h,黑暗8h,待植株生长至生殖生长期,取材料‘84-3’不同组织及不同发育时期种子(开花后10,20,30d),使用EASYspinPlus多糖多酚试剂盒提取RNA。
3.2LuFAD7基因的克隆
从亚麻参考基因组数据中筛选出LuFAD7基因(L.us.o.g.scaffold84.251),利用Primer5设计克隆引物(表3)。采用反转录试剂盒GoldenstarTM RT6 cDNA Synthesis Kit Ver.2进行反转录(北京擎科)。以‘84-3’、‘87-3’的叶片cDNA为模板,用Apex HFHSDNA Polymerase FS Master Mix (dyeplus)试剂盒(湖南艾科瑞)进行基因克隆,PCR产物进行回收后连接到pEASY-Blunt载体(北京全式金),转化至Trans1-T1感受态细胞后进行阳性单菌落筛选,经测序后利用DNAMAN进行比对。
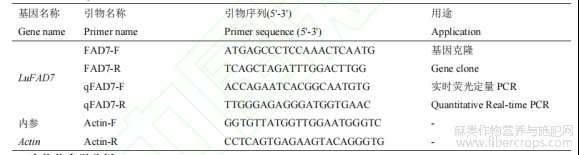
表3 引物序列
3.3生物信息学分析
通过在线工具对LuFAD7进行生物信息学分析(表4),使用MEGA构建进化树,使用DNAMAN软件进行氨基酸序列比对,使用MEME进行基序预测。
表4 生物信息学分析网站
3.4亚油酸和α-亚麻酸含量测定
采用索氏提取法提取开花后10、20、30d种子(45℃,烘至恒重)油脂,亚油酸和α-亚麻酸通过气相色谱-质谱联用仪测定(Zhonget al.,2021)。
3.5实时荧光定量PCR
使用Primer5设计荧光定量引物,以肌动蛋白(Actin)为内参基因(表3)。使用反转录试剂盒对亚麻‘84-3’材料的根、茎、叶、种子及不同发育时期种子(授粉后10,20,30d)的RNA进行反转录,每个组织设置三个生物学重复。荧光定量PCR采用SYBR®Green Premix Pro Taq HS qPCR试剂盒(湖南艾科瑞生物),2-△△Ct为基因相对表达量。
参考文献
Wang W.Y., Xu S.Q., He H.Y., and Wang W., 2020, Progress in nutrients and function of flaxseed, Zhongguo Youzhi (China Oils and Fats),45(4):83-85.(王维义,许帅强,何宏燕,王威,2020,亚麻籽的营养成分及功能研究进展.中国油脂,45(4):83-85.)
Wang S.Y., and Li X.W., 2010, Breeding of new flax varieties Neiyayou 1 and Neiya 7, Zuowu Zazhi (Crops),26(3):128.(王树彦,李心文,2010,胡麻新品种内亚油1号和内亚7号的选育,作物杂志,26(3):128.)
Zhao L., Dang Z.H., Li Y., Zhang J.P., Xie X.L., Wang X.S., and Hu Y.P., 2006, Health function, exploitation and utilization of flaxseed, Zhongguo Youzhi (China Oils and Fats),31(3):71-74.(赵利,党占海,李毅,张建平,谢小龙,王溪森,胡延萍,2006,亚麻籽的保健功能和开发利用,中国油脂,31(3):71-74.)
Wu S.P., 2010, Healthy function and extraction technology of a-linolenic acid in linseed, Zhongguo Niangzao (China Brewing),2:7-11.(吴素萍,2010,亚麻籽中α-亚麻酸的保健功能及提取技术,中国酿造,(2):7-11.)
Xue Y., Chen B., Win A N., Fu C., Lian J., Liu X., Wang R., Zhang X., and Chai Y., 2018, Omega-3 fatty acid desaturase gene family from two ω-3 sources, Salvia hispanica and Perilla frutescens: Cloning, characterization and expression, PLoS One.,13(1):e0191432.
Lee K.R., Lee Y., Kim E.H., Lee S.B., Roh K.H., Kim J.B., Kang H.C., and Kim H.U., 2016, Functional identification of oleate 12-desaturase and ω-3 fatty acid desaturase genes from Perilla frutescens var. frutescens, Plant Cell. Rep.,35(12):2523-2537.
Chellamuthu M., Kumaresan K., Subramanian S., 2022, Increase in alpha-linolenic acid content by simultaneous expression of fatty acid metabolism genes in Sesame (Sesamum indicum L.), Physiol. Mol. Biol. Plants.,28(3):559-572.
Horiguchi G., Iwakawa H., Kodama H., Kawakami N., Nishimura M., and Iba K., 1996, Expression of a gene for plastid ω-3 fatty acid desaturase and changes in lipid and fatty acid compositions in light- and dark- grown wheat leaves, Physiol. Plant,96(2):275-283.
Bhunia R.K., Kaur R., and Maiti M.K., 2015, Metabolic engineering of fatty acid biosynthetic pathway in sesame (Sesamum indicum L.): assembling tools to develop nutritionally desirable sesame seed oil, Phytochem. Rev.,15(5):799-811.
Liu X.Y., 2006, Isolation and functional analysis of LeFAD7 gene in tomato under temperature stress, Dissertation for Ph.D., Shandong Agricultural University, Supervisors: Meng Q.W.,pp.97-98.(刘训言,2006,番茄叶绿体ω-3脂肪酸去饱和酶基因(LeFAD7)的克隆及其在温度逆境下的功能分析,博士学位论文,山东农业大学,导师:孟庆伟.pp.97-98)
Liu C.X., Leng C.Y., and Liu F., 2012, Study on the transformation of CsFAD7 gene into Nicotiana tabacum, Beifang Yuanyi (Northern Horticulture),11:120-123.(刘春香,冷传远,刘福.CsFAD7基因转化烟草的研究,北方园艺,11:120-123.)
Vrinten P., Hu Z., Munchinsky M.A., Rowland G., and Qiu X., 2005, Two FAD3 desaturase genes control the level of linolenic acid in flax seed, Plant Physiol.,139(1):79-87.
Banik M., Duguid S., and Cloutier S., 2011, Transcript profiling and gene characterization of three fatty acid desaturase genes in high, moderate, and low linolenic acid genotypes of flax (Linum usitatissimum L.) and their role in linolenic acid accumulation, Genome.,54(6):471-83.
Urla R., Pavan K.P., Sripadi P., Khareedu V.R., and Vudem D.R., 2017, Cloning of fatty acid desaturase-coding sequence (Lufad3) from flax and its functional validation in rice, Plant Biotechnol. Rep.,11(5):259-270.
Li Y.H., Wang C.L., Li Y., and Yang Q.S., 2015, Cloning and expression analysis of fatty acid desaturase gene CmFAD7 in Chrysanthemum, Yuanyi Xuebao (Acta Horticulturae Sinica), 42(1): 65-74.(李永华,王翠丽,李永,杨秋生,2015,菊花脂肪酸脱饱和酶基因CmFAD7的克隆与表达分析,园艺学报,42(1):65-74.)
Anai T., Koga M., Tanaka H., Kinoshita T., Rahman S.M., and Takagi Y., 2003, Improvement of rice (Oryza sativa L.) seed oil quality through introduction of a soybean microsomal omega-3 fatty acid desaturase gene, Plant Cell. Rep.,21(10):988-992.
Orlova I.V., Serebriiskaya T.S., Popov V., Merkulova N., Nosov A.M., Trunova T.I., Tsydendambaev V.D., and Los D.A., 2003, Transformation of tobacco with a gene for the thermophilic acyl-lipid desaturase enhances the chilling tolerance of plants, Plant Cell. Physiol.,44(4):447-450.
Liu H.L., Yin Z. J., Xiao L., Xu Y. N., and Qu L.Q., 2012, Identification and evaluation of ω-3 fatty acid desaturase genes for hyperfortifying α-linolenic acid in transgenic rice seed, J. Exp. Bot.,63(8):3279-3287.
Song Y. S., and Sui N., 2019, Functional analysis of FAD7 gene in sweet sorghum, Shengwu Jishu Tongbao (Biotechnology Bulletin),35(5):35-41.(宋玉双,隋娜,2019,甜高粱FAD7基因的功能分析,生物技术通报,35(5):35-41.)
Murakami Y., Tsuyama M., Kobayashi Y., Kodama H., and Iba K., 2000, Trienoic fatty acids and plant tolerance of high temperature, Science,287(5452):476-479.
Liu W.H., Li W., Chen X.Y., Dai H.Y., and Zhang L.F., 2008, Cloning and sequence analysis of an ω-3 fatty acid desaturase gene from high-oil and low-oil soybean cultivars, Dadou Kexue (Soybean Science),27(4):569-571.(刘文浩,李伟,陈相艳,戴海英,张礼凤,2008,ω-3脂肪酸脱氢酶在高、低油大豆品种中的克隆与序列分析,大豆科学,27(4):569-571.)
Zhong Y.C., Zhang Z.Q., Chen J., Niu J., Shi Y.L., Wang Y., Chen T.X., Sun Z.M., Chen J.H., and Luan M.B., 2021, Physicochemical properties, content, composition and partial least squares models of A. trifoliata seeds oil, Food. Chem. X.,12:100131.
文章摘自:伍叶娜,姜慧,潘浪浪,常丽,黄思齐,栾明宝,李建军,唐慧娟,潘根,陈安国.亚麻LuFAD7基因的克隆及表达分析[J/OL].分子植物育种:1-15[2023-04-25].http://kns.cnki.net/kcms/detail/46.1068.S.20230310.1627.006.html