摘 要:本发明公开了与苎麻纤维细度关联的SSR标记及其应用,属于农业生产领域。本发明提供一种与苎麻纤维细度相关联的SSR标记及应用,包括与苎麻纤维细度相关联的SSR标记R74、R51、R90。本发明提供了所述苎麻纤维细度相关联的SSR标记在培育苎麻新品种中的应用以及在提高苎麻纤维细度中的应用。本发明的SSR标记能够用于苎麻的分子育种及提高苎麻纤维细度中,对于我国苎麻的育种和生产具有重要意义。同时,本发明也为分子标记辅助育种提供了有益的借鉴。
技术要点
1.一种与苎麻纤维细度相关联的SSR标记,其特征在于,包括SSR标记R74,所述SSR标记R74的序列如SEQIDNO.1和SEQIDNO.2所示。
2.如权利要求1所述的一种与苎麻纤维细度相关联的SSR标记,其特征在于,还包括SSR标记R51、R90,所述SSR标记R51序列如SEQIDNO.3和SEQIDNO.4所示,所述SSR标记R90序列如SEQIDNO.5和SEQIDNO.6所示。
3.如权利要求1所述的一种与苎麻纤维细度相关联的SSR标记,其特征在于,所述SSR标记R74和苎麻头季麻、二季麻和三季麻的苎麻纤维细度相关联。
4.如权利要求2所述的一种与苎麻纤维细度相关的SSR标记,其特征在于,所述SSR标记R51、R90和苎麻二季麻和三季麻的苎麻纤维细度相关联。
5.一种权利要求1或权利要求2任一项所述的SSR标记在培育苎麻新品种中的用途。
6.一种权利要求1或权利要求2任一项所述的SSR标记在提高苎麻纤维细度中的应用。
技术领域
本发明涉及农业生产领域,特别是涉及一种与苎麻纤维细度相关联的SSR标记及其应用。
背景技术
苎麻是具有中国特色的天然纺织原料。苎麻纤维具有拉力强,富弹性,耐水湿,耐热力大,富绝缘性等特性。并且苎麻纤维吸湿散湿快,透气性好,作为优良纺织原料被广泛用于制作服装、装饰用品和产业用纺织品等方面。纤维细度是评定苎麻纤维等级、纤维质量及纺织利用价值的关键性指标。由于纤维细度和产量负相关,高细度品种其产量构成因素整体较低。现有苎麻品种纤维质量难以满足纺织高支、细薄产品的要求,主要原因是缺乏高细度,2500支以上的品种。培育纤维细度高的品种是苎麻育种的现实要求,是突破苎麻产业经济发展瓶颈制约的重大因素,也是苎麻资源优势转变为经济优势的关键所在。
常规的育种方法是借助表型及育种家经验对作物的重要农艺性状进行选育,其效率低,周期长;而基于基因型选择的高效准确的分子辅助技术开启了作物育种的新方向。将分子标记技术用于作物重要农艺性状的辅助选择或者鉴定可大大缩短育种时间和提高选择效率。分子标记在苎麻上的应用主要集中在分子标记开发(Chenetal.2011;Liuetal.2013;Chenetal.2015)、种质资源的遗传多样性分析(揭雨成等,2002;郭安平等,2003;Liuetal.2008;Luanetal.2015b)、分子身份证构建(王晓飞等,2010;Luanetal.2015a)、遗传连锁图谱构建(邹自征,2012;栾明宝等,2013;Liuetal.2014)及QTL定位(Liuetal.2014)。Chenetal.(2015)通过转录组开发了52个可能与纤维品质相关的SSR分子标记。但是到目前从事发掘纤维细度的研究还鲜有报道。
现代育种有必要利用植物遗传多样性平台,并结合最新的基因组技术来发现新基因或等位基因,用于性状的改良。利用目标数量性状连锁分子标记,通过分子标记辅助选择,可有效提高数量性状的遗传改良进程。关联分析(Associationanalysis)是在植物数量性状研究和植物育种中应用的一种较新的分析方法。它以连锁不平衡为基础鉴定某一群体内性状与遗传标记或候选基因间的关系。与重组群体比较,它的显著优点是高通量,即能在全基因组范围有效地检测大量的具有不同遗传背景的种质资源材料的性状控制基因位点或区域;除高通量外,GWAS采用遗传背景丰富的自然群体为材料,不需要花多年时间构建特定的分离群体,具有作图定位精度高、可达到单基因的水平。目前全基因组关联分析已经成功应用于玉米、水稻、拟南芥,油菜、大麦等重要作物。
本发明旨在利用苎麻的自然群体为材料,利用具有多态性的SSR分子标记,分析其遗传多样性、群体结构等;同时对其纤维细度和SSR分子标记进行关联分析,开发并发掘与其相关的SSR位点,从而为苎麻纤维细度性状的QTL定位及其改良等研究提供科学依据;并应用于苎麻新品种育种方面。
发明内容
本发明的目的是提供与苎麻纤维细度相关联的SSR标记及其应用,以解决上述现有技术存在的问题。本发明以苎麻的自然群体为材料,利用具有多态性的SSR分子标记,对其纤维细度和SSR分子标记进行关联分析,开发并发掘了相关的SSR位点,从而为苎麻纤维细度性状的QTL定位及其改良等研究提供科学依据,并能够应用于苎麻新品种的育种。
为实现上述目的,本发明提供了如下方案:
本发明提供一种与苎麻纤维细度相关联的SSR标记,包括SSR标记R74,所述SSR标记R74的序列如SEQIDNO.1和SEQIDNO.2所示。
优选的,所述一种与苎麻纤维细度相关联的SSR标记还包括SSR标记R51、R90,所述SSR标记R51序列如SEQIDNO.3和SEQIDNO.4所示,所述SSR标记R90序列如SEQIDNO.5和SEQIDNO.6所示。
优选的,所述SSR标记R74和苎麻头季麻、二季麻和三季麻的苎麻纤维细度相关联。
优选的,所述SSR标记R51、R90和苎麻二季麻和三季麻的苎麻纤维细度相关联。
本发明还提供所述的SSR标记在培育苎麻新品种中的用途。
本发明还提供所述的SSR标记在提高苎麻纤维细度中的应用。
本发明公开了以下技术效果:
与传统QTL定位方法相比,GWAS采用遗传背景丰富的自然群体为材料,不需要花多年时间构建特定的分离群体,具有作图定位精度高、能同时扫描控制目标性状所有关联位点的优点。
本发明提供的SSR标记与苎麻纤维细度相关联,而培育纤维细度高与苎麻的品质直接相关联,为今后筛选优良种质、基因定位的分子标记辅助育种打下基础,本发明的SSR标记能够用于苎麻的分子育种及提高苎麻产量中。对于我国苎麻的育种和生产具有重要意义。同时,本发明也为分子标记辅助育种提供了有益的借鉴。
附图说明
为了更清楚地说明本发明实施例或现有技术中的技术方案,下面将对实施例中所需要使用的附图作简单地介绍,显而易见地,下面描述中的附图仅仅是本发明的一些实施例,对于本领域普通技术人员来讲,在不付出创造性劳动的前提下,还可以根据这些附图获得其他的附图。
图1为不同种质资源的进化树,图中各分支代表不同的样品;
图2为CV值随K值的变化趋势;
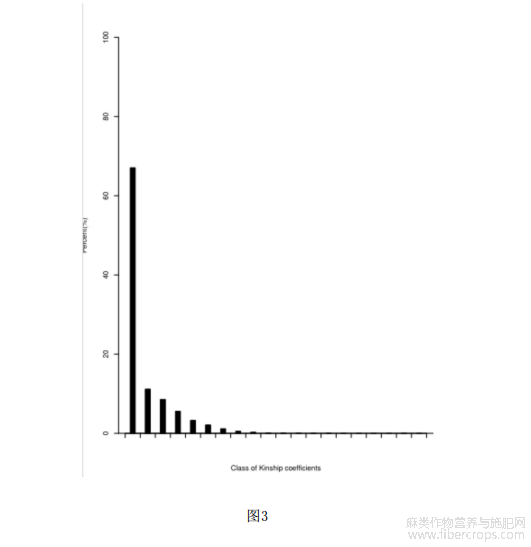
图3为不同种质资源的亲缘关系。
具体实施方式
现详细说明本发明的多种示例性实施方式,该详细说明不应认为是对本发明的限制,而应理解为是对本发明的某些方面、特性和实施方案的更详细的描述。
应理解本发明中所述的术语仅仅是为描述特别的实施方式,并非用于限制本发明。另外,对于本发明中的数值范围,应理解为还具体公开了该范围的上限和下限之间的每个中间值。在任何陈述值或陈述范围内的中间值以及任何其他陈述值或在所述范围内的中间值之间的每个较小的范围也包括在本发明内。这些较小范围的上限和下限可独立地包括或排除在范围内。
除非另有说明,否则本文使用的所有技术和科学术语具有本发明所述领域的常规技术人员通常理解的相同含义。虽然本发明仅描述了优选的方法和材料,但是在本发明的实施或测试中也可以使用与本文所述相似或等同的任何方法和材料。本说明书中提到的所有文献通过引用并入,用以公开和描述与所述文献相关的方法和/或材料。在与任何并入的文献冲突时,以本说明书的内容为准。
在不背离本发明的范围或精神的情况下,可对本发明说明书的具体实施方式做多种改进和变化,这对本领域技术人员而言是显而易见的。由本发明的说明书得到的其他实施方式对技术人员而言是显而易见得的。本申请说明书和实施例仅是示例性的。
关于本文中所使用的“包含”、“包括”、“具有”、“含有”等等,均为开放性的用语,即意指包含但不限于。
实施例1
1、材料与方法
1.1供试材料
以107份苎麻核心种质资源为研究材料。
1.2纤维细度及统计分析
2014年10月(2014年三麻)、2015年8月(2015年二麻)、2015年10月(2015年三麻)在成熟期收获苎麻,调查其纤维细度性状。利用SPSS软件,对107份种质资源的纤维细度和叶片粗蛋白含量进行统计描述分析。
1.3SSR分子标记分析
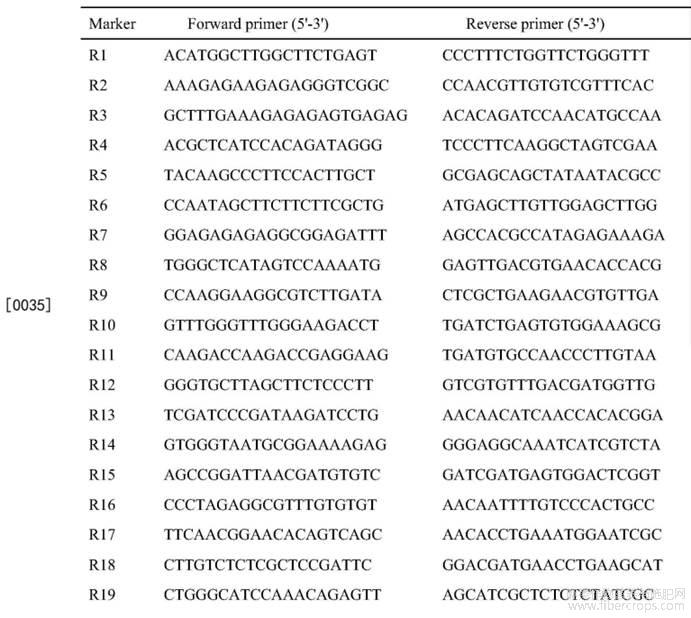
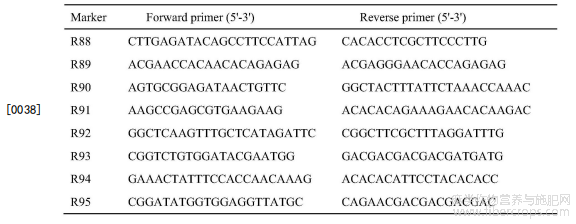
利用CTAB植物基因组DNA快速提取试剂盒提取DNA。用1%琼脂糖凝胶电泳检测纯度与浓度。参照Chen等的方法对实验室的95对SSR多态性引物进行PCR扩增,95对SSR多态性引物序列如表1所示。PCR产物烯酰氨凝胶垂直电泳聚丙烯酰胺凝胶电泳检测扩增产物,银染检测多态性。
表1 公开的95对SSR引物
1.4遗传多样性和聚类分析
利用powerMark3.25,分析其最大等位基因频率、基因型数、等位基因数、基因多样性、杂合度和多态性信息含量(PICvalue);根据NJ聚类法,构建其聚类图。
1.5群体结构分析
通过admixturer软件,分析107个样品的群体结构。分别假设107个样品的分群数(K值)为1-10,进行聚类,根据CV值来确定分群数;CV值最小时对应的K值,即为分群数。
1.6亲缘关系分析
本研究利用95对分子标记,使用软件SPAGeDi计算107份苎麻种质资源材料两两间的Kinship相似系数。运算得到的结果中,所有为负值的亲缘关系相似系数均设置为零,然后将所有的亲缘关系相关系数乘以2,应用于关联分析。
1.7关联分析
利用TASSLE软件的一般线性模型(generallinearmodel,GLM)和混合线性模型(mixedlinearmodel,MLM),将群体结构分析所得的Q值和亲缘关系K作为协变量,进行标记与性状的关联分析。
2、结果与分析
2.1纤维细度表型分析
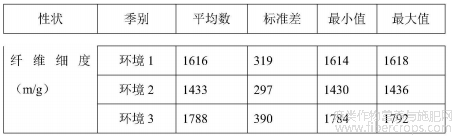
如表1所示。表中环境1指头季麻,环境2指二季麻,环境3指三季麻。
表1 纤维细度表型分析
2.2SSR分子标记分析
2.3遗传多样性分析
利用筛选出的95对SSR多态性引物对107份苎麻进行扩增,共扩增出252个多态性条带。各引物位点的多态信息含量PIC(Simpson多态性指数)平均为0.447969;每一个位点等位基因数平均为2.642105,期望平均值为2.185957;每个位点观察杂合度平均为0.319365,期望平均值为0.519303;基因多样性平均为0.522578;基因型多样性(shannon-wiener多样性指数)平均为0.82431;MAF平均为0.325672。
2.4聚类分析
如图1所示,进化树用来表示物种之间的进化关系,根据各类生物间的亲缘关系的远近,把各类生物安置在有分枝的树状的图表上,简明地表示生物的进化历程和亲缘关系。基于各个种质资源在各SSR位点的基因型,利用powermarker进行NJ聚类,获得各种质资源间的进化树。从种质资源间的进化树上可以看出,107份种质资源主要分为两大类。其中第一类包含87个品种,第二类包含20个品种。
2.5群体结构分析
利用admixture软件分析供试验材料的群体结构,分别假设107个样品的分群数(K值)为1-10,进行聚类,根据CV值来确定分群数。CV值随K值的变化趋势见图2。结果表明,当群体数K=1时其交叉验证误差值为0.87987,此时误差值最小,表明供试材料群体结构简单,可直接分为一个亚群。
2.6亲缘关系分析
如图3所示,自然群体中个体间的亲缘关系是影响关联分析结果的一个重要因素,这种亲缘关系可以用kinship相似系数来衡量。分析结果发现表明,67.704%的两两材料间的kinship值在0到0.05之间表明本研究利用的107苎麻种质资源之间的亲缘关系较远,具有较高的遗传多样性。
2.7SSR位点与苎麻纤维细度的关联分析
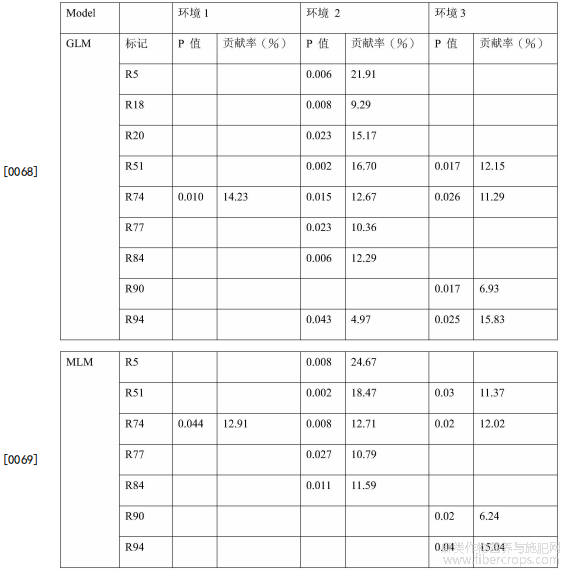
为避免群体结构和亲缘关系的存在影响关联分析的准确性,应用Tassel3.0软件,将核心种质各个体的Q值和Kinship值作为协变量,基于GLM模型和MLM模型进行纤维细度表型变异对标记变异的回归分析,探求相关性状的关联标记,并统计各位点的表型变异解释率。
结果(表2)表明,在基于GLM模型分析中头季麻、二季麻、三季麻分别检测到1个、8个、4个纤维细度关联分子标记(p<0.05)。在两个环境中同时检测到的有3个,其中R74分子标记在三个环境中全被检测到。在二季麻和三季麻中可以被同时检测到的标记有R51、R74、R90。R51、R74在两个环境中表型变异解释率均超过10%。
在基于MLM模型分析中头季麻、二季麻、三季麻分别检测到1个、5个、4个纤维细度关联分子标记(p<0.05);在两个环境中被同时检测到的标记有2个,其中R74分子标记在三个环境中全被检测到。在二季麻和三季麻中可以被同时检测到的标记有R51、R74。R51、R74在两个环境中表型变异解释率均超过10%。
MLM模型检测到的分子标记位点,在GLM模型中均被检测到。在MLM模型下,对于纤维细度而言,3种环境下均检测到与其相关的分子标记位点有1个;2种环境下均检测到的分子标记位点有1个;其余5个分子标记位点仅在一种环境下被检测到。说明苎麻纤维细度虽然存在效应稳定的QTL,但受基因型与环境互作的显著影响。
表2 纤维细度关联分子标记
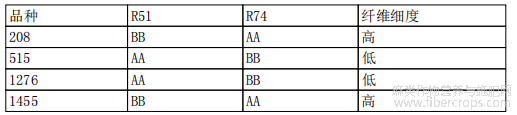
本发明利用分子标记R51和R74,对种质资源圃中的品种进行了快速检测,根据其基因型,成功预测了品种的纤维细度高低情况,下表3为4个品种的基因型和纤维细度高低情况。
以上所述的实施例仅是对本发明的优选方式进行描述,并非对本发明的范围进行限定,在不脱离本发明设计精神的前提下,本领域普通技术人员对本发明的技术方案做出 的各种变形和改进,均应落入本发明权利要求书确定的保护范围内。
摘自国家发明专利,发明人:陈晓蓉,栾明宝,晏文武,申请号202011145396.0,申请日2020.10.23