摘 要:在植物响应紫外线 B(Ultraviolet-B,UV-B)的过程中,UV-B 光受体 UVR8(UV Resistance Locus 8)对植物的光形态建成和生长代谢等过程具有重要调控作用。为探究罗布麻属植物 UV-B 光受体信息,该研究通过罗布麻(Apocynum venetum)和大麻状罗布麻(A. cannabinum)全基因组数据进行 UV-B 光受体 UVR8 的筛选与生物信息学分析,同时利用转录组数据分析 UV-B 胁迫处理下的 UVR8 基因表达模式。结果表明:(1)罗布麻有 6个 UVR8 基因,大麻状罗布麻有 5 个 UVR8 基因,前者分布在 1、7、9 和 11 号染色体上,后者分布在 1、8 和9 号染色体上。(2)UVR8 蛋白为亲水性不稳定蛋白,定位在细胞核,不存在跨膜结构和信号肽,二级结构主要由延伸链、无规则卷曲、α-螺旋和β-转角构成。AvUVR8b 和 AcUVR8a 蛋白三级结构与拟南芥 UVR8(AtUVR8)最为类似,且与小粒咖啡(CaUVR8)和伊德斯种咖啡(CeUVR8)的亲缘关系最近。该研究发现,罗布麻 AvUVR8b和大麻状罗布麻 AcUVR8a 基因和蛋白结构与 AtUVR8 基因及蛋白高度相似。(3)当以一定剂量 UV-B(17.52kJ·m-2·d-1)处理两种罗布麻植株时,AvUVR8b 和 AcUVR8a 的表达量上调。据此推测在响应 UV-B 时,AvUVR8b基因在罗布麻中起主要作用,AcUVR8a 基因在大麻状罗布麻中起主要作用。(4)顺式作用元件分析结果表明,UVR8 的表达受光照、温度、水分、氧气和激素等因素的调控。该研究将为进一步研究罗布麻属 UVR8 的基因功能奠定基础,同时为解析罗布麻属植物适应 UV-B 的分子机制提供线索。
关键词:罗布麻,大麻状罗布麻,UVR8 基因,表达量分析,生物信息学分析
太阳光不仅是光合作用的能量来源,也是调控植物生长发育、昼夜节律、代谢物合成等的重要环境因子(Frohnmeyer et al., 2003;Jenkins, 2014a,b)。紫外线(ultraviolet, UV)作为太阳光的组成部分,按其波长可分为长波紫外线(UV-A, 320~400 nm),中波紫外线(UV-B, 280~320 nm)和短波紫外线(UV-C, 100~280 nm)(刘明雪等,2012;陈慧泽等,2021)。UV-A 可穿过大气层直接到达地表,但不会对生物造成显著影响;UV-C可使大部分植物迅速死亡,但因其波长较短和穿透率差等因素,在到达地表之前就已被大气层吸收;UV-B 大部分可被臭氧层吸收,作为生物有效辐射的 UV-B 具有双重效应。高强度 UV-B 是逆境胁迫因子,破坏细胞的DNA、蛋白质和脂类等生物大分子,甚至导致植物死亡;低强度 UV-B 是细胞信号传导的调控因子,对植物的光形态建成和代谢等生理过程具有重要作用(Frohnmeyer, 2003;Shamala et al., 2020)。
早在2002年筛选对UV-B超敏感的拟南芥突变体(uvr8-1)中鉴定到了光受体UVR8(Kliebenstein et al., 2002),并于 2011 年证实 UVR8 为感受 UV-B 的特异性光受体(Rizzini et al., 2011)。目前,对 UVR8 结构和功能研究在拟南芥中进行的较多,研究结果表明,UVR8 蛋白为盐桥链接的同源二聚体结构,其单体由 7 个片状结构首尾相连的β螺旋纵向排列围成的环形结构(Christie et al., 2012;鲍思元,2016)。高度保守的 UVR8 色氨酸残基(W)具有维持其蛋白结构稳定、接收和传递 UV-B 信号等功能(Jenkins, 2014a;张宏江等,2019;Li et al., 2020),AtUVR8 直接通过其 W233 和 W258 接收 UV-B 而无需借助其他辅助因子作为发色团(Rizzini, 2011;O'Hara et al., 2012;Jenkins, 2014b;Yang et al., 2018)。当植株未照射 UV-B 时,UVR8 以二聚体形式存在于细胞质;照射 UV-B 时,其盐桥断裂形成单体并转移到细胞核(Wu et al., 2012)与解离自 CUL4-DDB1(cullin4damaged DNA binding protein 1) E3 泛素连接酶的 COP1-SPA(constitutively photomorphogenic 1-suppressor of phyA-105)形成 UVR8-COP1-SPA 新复合体(Rizzini, 2011;Huang et al., 2013;Vanesa et al., 2019),从而减少COP1 对 HY5(Long Hypocotyl 5)的降解(Huang, 2013),同时促进 HY5、HYH(HY5 Homolog)和 MYB 等转录因子表达,刺激黄酮类化合物合成过程中相关酶基因的转录(Hartmann et al., 2005;钱崇祯,2019;Shamala, 2020;凌成婷等,2021)。当 UVR8 介导的下游基因过度表达时启动其负反馈机制,如激活 RUP1(repressor of UV-Bphotomorphogenesis 1)/RUP2 和 STO/BBX24(Salt Tolerance/BBX24)等转录(Jenkins, 2014b;Parihar et al., 2015;李国良等,2015)。UVR8 与 RUP1/RUP2 相互作用促进其二聚体化(Cloix et al., 2012;Hideg et al., 2013),使 UVR8 可及时响应 UV-B 光信号。
罗布麻(Apocynum venetum)和大麻状罗布麻(A. cannabinum)为夹竹桃科罗布麻属多年生宿根草本或半灌木植物,具有耐旱、盐碱、贫瘠等强抗逆性(王东清等,2012);拥有“野生纤维之王”的美誉,其纺织物具有透气保暖、抗静电和紫外线防护等功能;作为药用植物,可全株入药,发挥降血压、血脂、血糖和抗衰老等作用(Li et al., 2018),其叶还可制保健茶。黄酮类化合物是罗布麻属植物的主要药用成分(张洋,2021),其合成途径复杂,受内源基因和外界环境的影响。在全球气候变化的背景下,植物面临着越来越严重的紫外线胁迫,探究植物对 UV-B 的响应机制与胁迫反应显得极其重要。在绿藻、苜蓿、大豆和银杏等植物中已展开对UV-B 光受体 UVR8 结构和功能的部分研究。目前,对罗布麻属植物的研究多集中在其抗逆性、药用成分和纤维开发利用等方面,对其 UVR8 的研究尚未有报道。本文基于两种罗布麻的全基因组数据筛选 UVR8 基因,依托生物信息学分析,同时借助转录组数据探究 UVR8 基因表达模式,拟探讨以下问题:(1)UVR8 基因结构、顺式作用元件和染色体定位;(2)UVR8 保守结构域、蛋白结构和理化性质;(3)UVR8 磷酸化位点和系统进化关系等;(4)UV-B 胁迫下的 UVR8 基因表达模式。通过本研究以期进一步深入解析 UVR8 功能及其在罗布麻属 UV-B 响应机制和药用成分合成等方面的研究提供线索。
1材料与方法
1.1材料和数据来源
罗布麻和大麻状罗布麻的 UVR8 基因序列和蛋白序列,均来自本实验室前期全基因组测序工作所获得的数据(宋立肖等,2019;宋立肖,2020),并从拟南芥(Arabidopsis thaliana)数据库 TAIR(https://www.arabidopsis.org/)中下载 AtUVR8 蛋白序列(Protein:AT5G63860.1)。
1.2 方法
1.2.1 两种罗布麻 UVR8 基因筛选
本研究通过罗布麻和大麻状罗布麻全基因组的 IPRSCAN、KEGG、NR 和 Swissport 注释结果,分别筛选出注释到 UV-B 光受体 UVR8 的基因序列和蛋白序列。利用 BioEdit 软件进行分析,以 AtUVR8 蛋白序列为种子序列 进 行 本 地 BLAST 比 对 , E-value<1e-10 , Identity ≥ 30% , 筛 选 最 佳 UVR8 蛋 白 序 列 。 然 后 采 用Pfam(https://pfam.xfam.org/search)和 SMART(http://smart.emblheidelberg.de/)软件进行结构域验证,删除冗余序列,最终分别获得罗布麻和大麻状罗布麻 UVR8 的基因序列和蛋白序列。
1.2.2 两种罗布麻 UVR8 基因染色体定位
基于两种罗布麻全基因组注释文件,获得 UVR8 基因在染色体上的位置信息,并通过软件 MG2C(Chao et al., 2021)(http://mg2c.iask.in/mg2c_v2.1/)在线绘制 UVR8 基因染色体定位图。
1.2.3 两种罗布麻 UVR8 基因结构和保守结构域分析
两种罗布麻 UVR8 基因的外显子和内含子位置信息分别参考基因组注释 gff3 文件,采用 GSDS 2.0(Hu et al., 2015)(http://gsds.gao-lab.org/index.php)工具对 UVR8 基因结构进行分析,UVR8 蛋白保守结构域利用软件 MEME(Bailey et al., 2009)(http://meme-suite.org/tools/meme)进行预测。
1.2.4 两种罗布麻 UVR8 蛋白基本理化性质分析
利用在线软件 ExPASy(Gasteiger et al., 2003)(https://web.expasy.org/cgi-bin/protparam/protpara)进行 UVR8蛋白的氨基酸数、分子量、理论等电点、不稳定指数和脂肪族氨基酸指数等分析。
1.2.5 两种罗布麻 UVR8 蛋白结构分析和亚细胞定位
采用SOPMA(https://npsa-prabi.ibcp.fr/cgi-bin/npsa_automat.pl?page=npsa_sopma.html)工具预测 UVR8 蛋白二级结构,以 AtUVR8 蛋白为模板,利用 Phyre2 ( Kelley et al., 2015 )
(http://www.sbg.bio.ic.ac.uk/phyre2/html/page.cgi?id=index)软件分析蛋白三级结构。通过软件 Cell PLoc 2.0(http://www.csbio.sjtu.edu.cn/bioinf/CellPLoc-2/),选择Euk-PLoc 2.0 进行 UVR8 蛋白核定位分析。
1.2.6 两种罗布麻 UVR8 蛋白跨膜结构预测、信号肽分析和磷酸化位点分析
蛋白跨膜结构利用软件TMHMM 2.0(http://www.cbs.dtu.dk/services/TMHMM/)进行分析,信号肽预测通过SignalP 5.0 (https://services.healthtech.dtu.dk/service.php?SignalP-5.0 ) 进行,并采用软件NetPhots 3.1(http://www.cbs.dtu.dk/services/NetPhos/)统计蛋白磷酸化位点种类和数目。
1.2.7 两种罗布麻 UVR8 基因顺式作用元件分析
利用 TBtools(Chengjie et al., 2020)软件获得 UVR8 基因上游 2 000 bp 序列,作为启动子序列。通过PlantCARE(http://bioinformatics.psd.ugent.be/webtools/plantcare/html/)数据库对所得序列进行预测,采用 TBtools软件绘图,对主要顺式元件的位置和数量进行分析。
1.2.8 两种罗布麻 UVR8 蛋白系统进化分析
采用在线工具 NCBI(https://www.ncbi.nlm.nih.gov/)的 BLASTn 进行同源性搜索,获得两种罗布麻 UVR8基因的同源序列。通过 MEGA 11.0 软件构建系统进化树,利用 Clustal W 进行多重序列比对,采用邻接法(Neighbour-Joining,NJ)构建 UVR8 蛋白系统进化树,Bootstrap method 值设为 1 000,其他参数采用系统默认值。通过工具 ITQ(https://itol.embl.de)进行美化。
1.2.9 两种罗布麻 UVR8 基因表达量分析
本实验室于 2021 年春季在大棚内进行两种罗布麻的盆栽种植(林下土与营养土按 1∶1 混合),田间常规化管理。根据 Gao 等(2019)对 UV-B 辐射剂量的等级划分,结合宁夏银川地区夏季晴天的 UV-B 强度和两种罗布麻的强抗逆性,设自然光照(含 UV-B 强度约 8~11 W·m-2)为对照,在自然光照的基础上增加 UV-B 辐射处理。对生长至 30~40 cm 的两种罗布麻冠层上方 0.5 m 处安装 UV-B 灯管(飞利浦 TL 100W/01),于每天早上10:00 和下午 14:00 开始各处理 4 次,每次处理时长为 10 min,每间隔 10 min 处理一次。可在冠层检测到增加的 UV-B 辐射剂量是 17.52 KJ·m-2·d-1,强度为 3.65 W·m-2(相当于银川地区夏季晴天 UV-B 强度增加33.2%~45.6%左右)。采用 0.1 mm 的醋酸纤维膜覆盖灯管以屏蔽 280 nm 以下的 UV-C,UV-B 强度由 Lutron 公司的 UV-340A 紫外线辐照计测得。期间分别在 UV-B 处理的第 0 天、0.5 天、1 天、4 天、7 天(d0、d0.5、d1、d4、d7)对植株上部成熟叶片进行取样,每个样本均为 3 个生物学重复,-80 ℃保存。本研究借助前期试验的转录组数据(暂未公开)对涉及 UVR8 基因的表达量进行分析,通过 TBtools 软件构建 UVR8 基因表达量热图。
2 结果与分析
2.1 两种罗布麻 UVR8 基因筛选
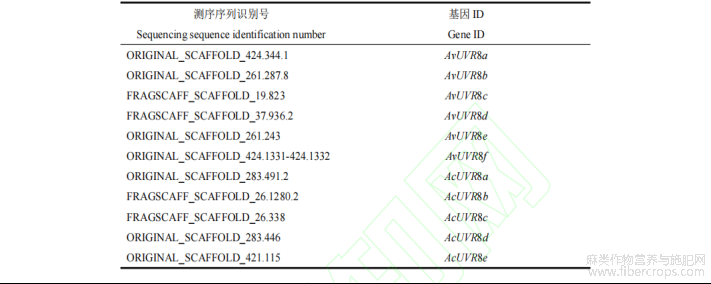
通过罗布麻和大麻状罗布麻的全基因组测序数据,分别筛选到了 10 个罗布麻 UVR8 蛋白序列和 14 个大麻状罗布麻 UVR8 蛋白序列。以拟南芥 AtUVR8 蛋白序列为种子序列,将筛选结果进行序列比对及在线结构域分析。最终获得 6 个罗布麻 UVR8(AvUVR8)基因序列和 5 个大麻状罗布麻 UVR8(AcUVR8)基因序列,分别命名为AvUVR8a、AvUVR8b、AvUVR8c、AvUVR8d、AvUVR8e、AvUVR8f 和 AcUVR8a、AcUVR8b、AcUVR8c、AcUVR8d、AcUVR8e。
表1 两种罗布麻 UVR8 基因测序序列识别号和基因 ID
2.2 两种罗布麻 UVR8 基因染色体定位
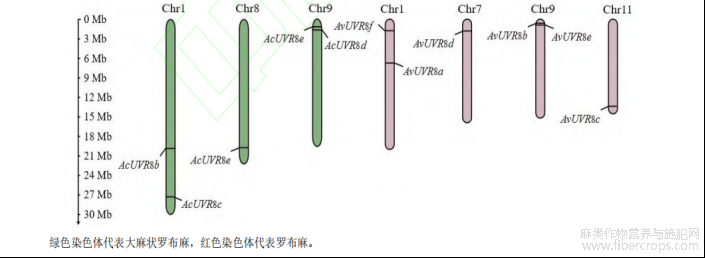
两种罗布麻的全基因组各有 11 条染色体,采用 MG2C 软件绘制 UVR8 基因染色体定位图,分析结果(图 1)显示,UVR8 基因间无串联重复现象,大麻状罗布麻 AcUVR8 基因不均匀地分布在 3 条染色体上,其中 AcUVR8e分布在 8 号染色体,1 和 9 号染色体则各分布有 2 个 AcUVR8 基因,分别是 AcUVR8b 和 AcUVR8c,AcUVR8a和 AcUVR8d;罗布麻 AvUVR8 基因分布在 4 条染色体上,其中 AvUVR8d 和 AvUVR8c 分别分布在 7 和 11 号染色体,1 和 9 号染色体则各分布有 2 个 AvUVR8 基因,分别是 AvUVR8f 和 AvUVR8a,AvUVR8b 和 AvUVR8e。
图1 两种罗布麻 UVR8 基因的染色体定位
2.3 两种罗布麻 UVR8 基因结构和蛋白保守结构域分析
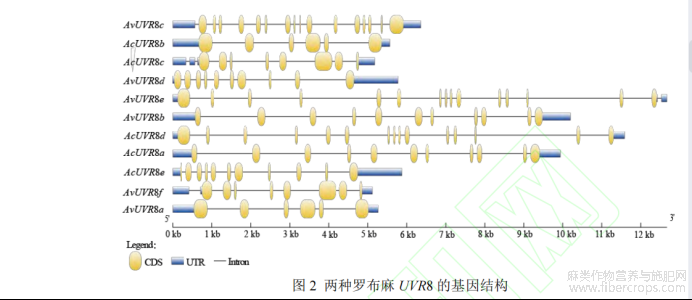
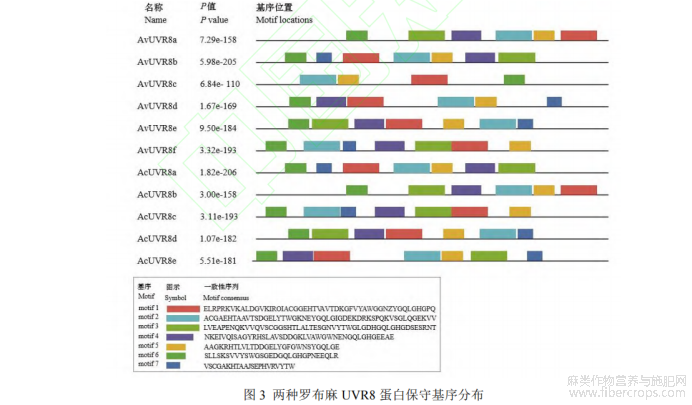
通过两种罗布麻基因组注释 gff3 文件,采用 GSDS 2.0 软件对 UVR8 基因结构进行分析。结果表明,UVR8基因皆含上、下游非编码区、内含子和外显子,但基因长度不同,外显子数也存在差异(图 2)。AvUVR8 基因长度为 5 115~12 672 bp,外显子数在 6~16 个,其中 AvUVR8c 外显子数最高(16 个),AvUVR8a 外显子数最低(6 个);AcUVR8 基因长度为 5 876~11 585 bp,AcUVR8 外显子数在 6~15 个,其中 AcUVR8d 外显子数最高(15个),AcUVR8b 外显子数最低(6 个)。此外,利用 MEME 软件分析 UVR8 蛋白保守结构域(motif),motif设为 7,其他参数为默认值。结果(图 3)显示,AvUVR8 的 motif 数为 4~7 个,其中 AvUVR8b、AvUVR8e 和AvUVR8f 的 motif 数最多(7 个),AvUVR8c motif 数最少(4 个);AcUVR8 的 motif 数为 6~7 个,其中 AcUVR8b的 motif 数最少(6 个),其他 4 个 AcUVR8 则各有 7 个 motif。同种罗布麻的 UVR8 motif 之间存在一定差异,但两种罗布麻的 UVR8 之间具有一定相似性,如 AvUVR8a 与 AcUVR8b,AvUVR8b 与 AcUVR8a,AvUVR8e与 AcUVR8d,AvUVR8f 与 AcUVR8c motif 的数目、位置和氨基酸组成等两两相似。
2.4 两种罗布麻 UVR8 蛋白质理化性质分析
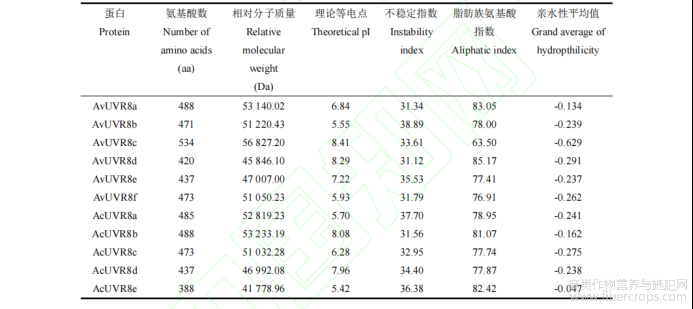
采用 ExPASy 工具对 UVR8 蛋白理化性质进行预测,结果(表 2)表明,AvUVR8 蛋白长度为 420~534 aa,相对分子质量在 45 846.10~56 827.20 Da,理论等电点在 5.55~8.41 之间。其中 AvUVR8c 的氨基酸数、相对分子质量和等电点最高,分别为 534 aa、56 827.20 Da 和 8.41;AvUVR8d 的氨基酸数和相对分子质量最小,分别为420 aa 和 45 846.10 Da。当蛋白不稳定指数>40 时,判定该蛋白为不稳定蛋白;当蛋白不稳定指数<40 时,判定该蛋白为稳定蛋白(夏巧玉,2007)。AvUVR8 蛋白的不稳定指数皆小于 40,为不稳定蛋白,其中 AvUVR8b的不稳定指数最高(38.89),AvUVR8d的不稳定指数最低(31.12)。AvUVR8 蛋白脂肪族氨基酸指数在 63.50~85.17之间,亲水性平均值在-0.629~-0.134 之间且小于 0,为亲水性蛋白。仅 AvUVR8a、AvUVR8b 和 AvUVR8f 等电点<7,为酸性蛋白,其余 3 个为碱性蛋白。
AcUVR8 蛋白长度为 388~488 aa,相对分子质量在 41 778.96~53 233.19 Da,理论等电点在 5.42~8.08 之间。其中 AcUVR8b 的氨基酸数、相对分子质量和等电点最高,分别为 488 aa、53 233.19 Da 和 8.08;AcUVR8e 的氨基酸数、相对分子质量和等电点最小,分别为 388 aa、41 778.96 Da 和 5.42。AcUVR8 蛋白的不稳定指数皆小于 40,为不稳定蛋白,其中 AcUVR8a 的不稳定指数最高(37.70),AvUVR8b 的不稳定指数最低(31.56)。AcUVR8 蛋白脂肪族氨基酸指数在 77.74~82.42 之间,亲水性平均值在-0.047~-0.275 之间且小于 0,为亲水性蛋白。仅 AcUVR8b 和 AcUVR8d 等电点>7,为碱性蛋白,其余 3 个为酸性蛋白。
表2 两种罗布麻 UVR8 蛋白理化性质
2.5 两种罗布麻 UVR8 蛋白结构分析和亚细胞定位
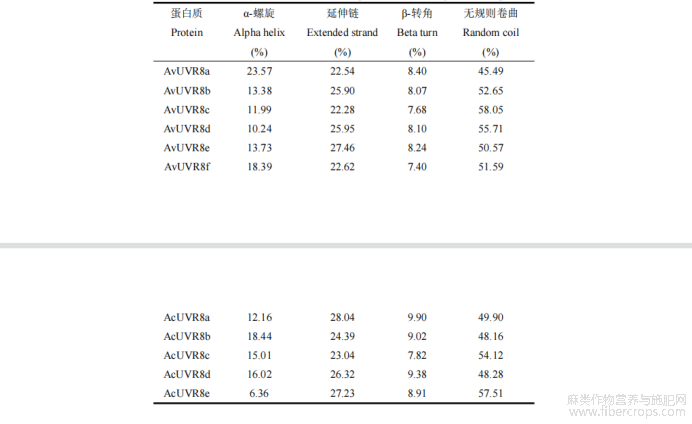
利用 SOPMA 软件分析 UVR8 蛋白二级结构,结果表明,UVR8 蛋白二级结构由α-螺旋(alpha helix)、β-转角(beta turn)、延伸链(extended strand)和无规则卷曲(random coil)4 部分组成(表 3,图 4)。其中无规则卷曲占比最高(45.49%~58.05%),其次为延伸链(22.28%~28.04%),β-转角占比最低(7.40%~9.90%),可见延伸链、无规则卷曲和α-螺旋结构为 UVR8 蛋白二级结构的主要组成部分。另外,本研究通过 Cell PLoc 2.0在线软件,选择 Euk-PLoc 2.0 对 UVR8 蛋白进行核定位预测,结果显示 UVR8 蛋白皆定位在细胞核。
表3 两种罗布麻 UVR8 蛋白二级结构组成
以 AtUVR8 蛋白为模板,采用软件 Phyre2 预测 UVR8 蛋白三级结构。发现仅 AvUVR8b 和 AcUVR8a 三级结构同 AtUVR8 相似,单体由 7 个完整的 RCC1 保守基序形成七叶β-折叠结构,而其他 UVR8 蛋白结构因 RCC1保守基序不完整或缺少,无法形成完整的七叶β-折叠结构,表明 AvUVR8b 和 AcUVR8a 在植株响应 UV-B 时发挥重要作用(图 5)。
2.6 两种罗布麻 UVR8 蛋白跨膜结构预测、信号肽分析和磷酸化位点分析
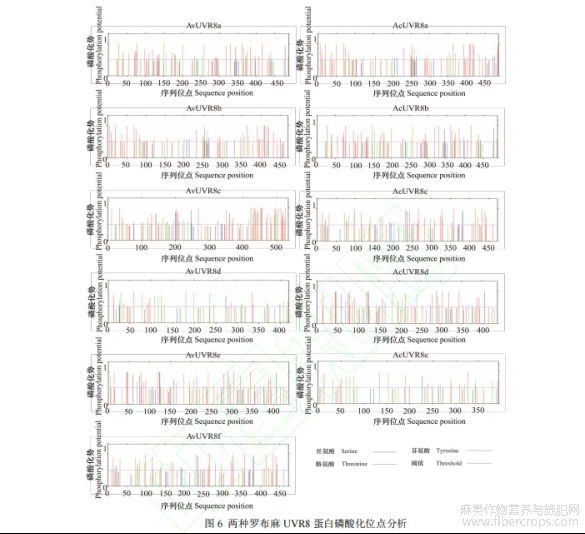
UVR8 蛋白跨膜结构利用软件 TMHMM 2.0 进行分析,信号肽预测采用 SignalP 5.0 软件,并通过工具NetPhots 3.1 分析蛋白磷酸化位点。UVR8 磷酸化位点预测结果显示,AvUVR8 存在 35~50 个磷酸化位点,其中有 13~31 个丝氨酸(serine)位点,10~19 个苏氨酸(threonine)位点和 4~8 个酪氨酸(tyrosine)位点(图 6)。其中 AvUVR8a 磷酸化位点数最高(50 个),AvUVR8d 磷酸化位点数最低(35 个)。AcUVR8 存在 31~51 个磷酸化位点,其中有 12~31 个丝氨酸位点,10~17 个苏氨酸位点和 2~8 个酪氨酸位点。其中 AcUVR8 磷酸化位点数最高(51 个),AvUVR8d 磷酸化位点数最低(31 个)。另外,预测结果表明两种罗布麻 UVR8 蛋白不存在跨膜结构和信号肽。
2.7 两种罗布麻 UVR8 基因顺式作用元件分析
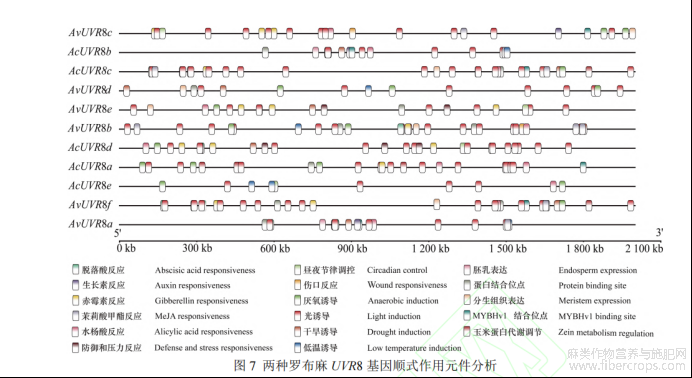
本研究通过 PlantCARE 软件对 UVR8 基因编码区上游 2 000 bp 序列进行顺式作用元件分析,结果(图 7)显示,除了基础性元件(TATA-box 和 CAAT-box 等)之外,UVR8 基因顺式作用元件主要涉及光诱导、激素反应、逆境胁迫响应、生长发育响应等。其中,光诱导元件数量最多(117 个),主要包括 ATC-motif、Box 4、I-box、TCT-motif、GA-motif、GT1-motif、G-box、AT1-motif、ACE 等元件;其次为激素反应元件(44 个),主要有参与赤霉素反应的 TATC-box、GARE-motif 和 P-box,响应茉莉酸甲酯的 CGTCA-motif 和 TGACG-motif,介导水杨酸反应的 TCA-element,涉及脱落酸反应的 ABRE 及与生长素反应相关的 TGA-element;响应逆境胁迫元件(38 个)主要有响应厌氧反应的 ARE,涉及伤口反应的 WUN-motif,参与干旱反应的 MBS,有关低温胁迫的 LTR 及参与防御和压力反应的 TC-rich repeats;生长发育响应元件数量最少(18 个),主要涉及昼夜节律调控元件 circadian,分生组织表达元件 CAT-box,胚乳表达元件 GCN4-motif 和调节玉米蛋白代谢的 O2-site元件。以上说明两种罗布麻 UVR8 基因的表达不仅受光照的影响,还受到温度、水分、氧气和内源激素等因素的调控。
2.8 两种罗布麻 UVR8 蛋白系统进化分析
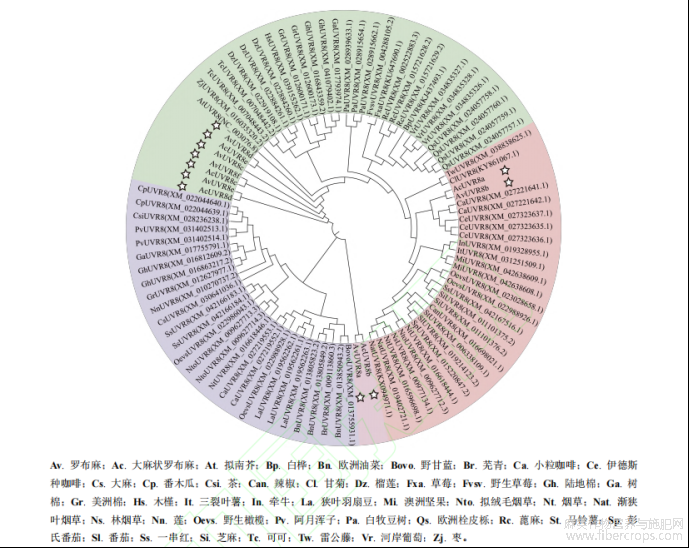
UV-B 光受体 UVR8 最早发现于拟南芥(Kliebenstein, 2002),并陆续在其他植物中发现。为进一步探究两种罗布麻 UVR8 蛋白系统进化关系,本研究通过 NCBI 软件的 BLASTn 进行同源性搜索并下载了 42 种植物的UVR8 基因序列,涉及 94 条 UVR8 基因。借助软件 MEGA 11.0 的 Clustalw 进行多重序列比对,并采用邻接法构建 UVR8 蛋白系统进化树。分析结果显示,聚类树分为 3 个亚族,两种罗布麻的 UVR8 蛋白分布在两个亚族上,其中 AvUVR8a 和 AcUVR8b 未与 AtUVR8 共处同一亚族且单独占一亚族,说明其与 AtUVR8 的亲缘关系存在一定差距;AvUVR8b 和 AcUVR8a 聚集在一处且与小粒咖啡(CaUVR8)和伊德斯种咖啡(CeUVR8)的亲缘关系最近,其次与甘菊(ClUVR8)、雷公藤(TwUVR8)、澳洲坚果(MiUVR8)、三裂叶薯(ItUVR8)和牵牛(InUVR8)的亲缘关系较近;在两种罗布麻 UVR8 蛋白主要聚集处,其与拟南芥(AtUVR8)和枣(ZjUVR8)的亲缘关系最近,其次与可可(TcUVR8)、榴莲(DzUVR8)、木槿(HsUVR8)、陆地棉(GhUVR8)、树棉(GaUVR8)和美洲棉(GrUVR8)的亲缘关系较近(图 8)。说明在两种罗布麻 UVR8 蛋白主要聚集的亚族上,各物种间 UVR8 蛋白存在明显的同源关系。
. 图8 UVR8 蛋白系统进化树
2.9 两种罗布麻 UVR8 基因表达量分析
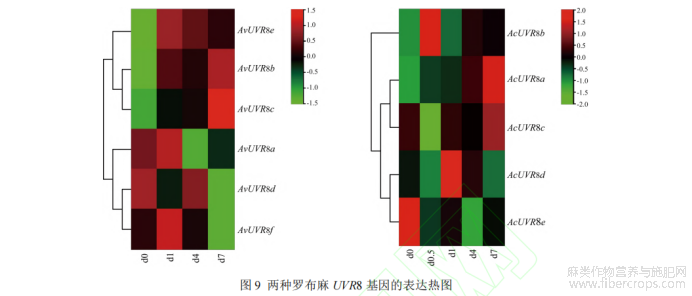
利用 TBtools 软件构建 UV-B 胁迫处理下的 UVR8 基因表达量热图,其中 d0 为对照组,罗布麻苗在 0 和 0.5d 处理后,观测到的表型变化和观测数据不明显,故对两组数据进行合并处理。结果显示,在罗布麻中,AvUVR8b、AvUVR8c 和 AvUVR8e 表达量均上调,其中 AvUVR8b 在 d7 时最高;AvUVR8e 在 d1 时最高,随后下降;AvUVR8c呈上升趋势。AvUVR8a、AvUVR8d 和 AvUVR8f 表达量均下调,其中 AvUVR8a 在 d1 时最高,d4 时最低;AvUVR8d在 d0 时最高,d7 时最低;AvUVR8f 在 d1 时最高,d7 时最低(图 9)。在大麻状罗布麻中,AcUVR8a 和 AcUVR8b表达量均上调,其中 AcUVR8a 呈上升趋势,AcUVR8b 在 d0.5 时最高。AcUVR8c 和 AcUVR8d 表达量波动较大,其中 AcUVR8c 下降,但从 d0.5 开始呈上升趋势,其在 d0.5 时最低,d7 时最高;AcUVR8d 在 d1 时最高,d0.5时最低。AcUVR8e 表达量下调,在 d0 时最高,d4 时最低(图 10)。表明在植物响应 UV-B 胁迫的过程中,AvUVR8b、AvUVR8c 和 AvUVR8e 在罗布麻中起重要作用;AcUVR8a 和 AcUVR8b 在大麻状罗布麻中起重要作用。
3 讨论与结论
随着人类社会的发展,臭氧层变薄导致地表 UV-B 增强(Caldwell et al., 1989),从而影响植物的光合作用速率、代谢和生态等作用(鲍思元,2016),继而对生长发育及作物产量等造成威胁。UVR8 作为 UV-B 的特异性光受体,研究其结构、功能及 UV-B 响应机制等对作物来说是必要的(Wargent et al., 2013)。在 UV-B 下,uvr8-1 突变体对黄酮类化合物和花青素合成关键基因的诱导下降,查尔酮合酶 mRNA 和蛋白表达不再上调(Kliebenstein, 2002)。当 UVR8 过表达时 UV-B 介导的光形态建成更显著,对 UV-B 的适应和耐受能力增强(Favory et al., 2009)。研究发现,UVR8 在响应 UV-B 的过程中通过调控多项生命活动提高植株的适应性和抗逆性(Jenkins, 2014b;Vandenbussche et al., 2014),其中低剂量 UV-B 辐射抑制下胚轴和根的生长(Frohnmeyer, 2003;Wellmann, 1976),同时促进 UV-B“防晒剂”黄酮类化合物合成等以增强适应性(Winkel-Shirley, 2002;Hartmann et al., 2005;Gruber et al., 2010),UV-B 损伤修复主要体现在抗氧化系统和酶修复 DNA 损伤(Jenkins, 2014b)。这为研究罗布麻属 UVR8 功能及 UV-B 调控网络提供线索。
本研究通过罗布麻和大麻状罗布麻全基因组数据筛选 UVR8 蛋白序列,以 AtUVR8 蛋白为种子序列进一步筛选,最终获得 6 个罗布麻 UVR8 基因和 5 个大麻状罗布麻 UVR8 基因,并对其进行生物信息学分析,同时利用 UV-B 胁迫处理数据分析 UVR8 基因表达模式。研究结果显示,UVR8 基因不均匀地分布在多条染色体上,且不存在串联重复现象。并发现同一物种的 UVR8 (AvUVR8 或 AcUVR8)蛋白序列存在一定差异,但 AvUVR8 和AcUVR8 之间具有相似性,如 AvUVR8a 与 AcUVR8b,AvUVR8b 与 AcUVR8a 等蛋白保守基序的数目、位置和种类高度相似。有研究报道,不同物种 UVR8 蛋白的关键氨基酸残基数目和位置高度相似,暗示 UVR8 蛋白在进化上相对保守(Yang et al., 2018),即光合生物的紫外线防护作用具有相似的分子功能(Rizzini, 2011)。在本研究中,UVR8 蛋白二级结构组成相似,但环形三级结构并不完全相同。其中 AvUVR8b 和 AcUVR8a 的三级结构同 AtUVR8 最接近,单体由 7 个完整的 RCC1 保守基序形成七叶β-折叠结构,与报道的 UVR8 结构研究一致(Jenkins, 2014a;鲍思元,2016;张宏江,2019)。而其他 UVR8 蛋白可能在进化过程中逐渐退化,导致 RCC1保守基序不完整或缺少,无法形成完整的七叶β-折叠结构,表明 AvUVR8b 和 AcUVR8a 在响应 UV-B 时可能发挥主要作用。在两种罗布麻 UVR8 蛋白磷酸化中,以丝氨酸修饰为主,并涉及苏氨酸和酪氨酸修饰。且 UVR8蛋白为不稳定性亲水蛋白,不存在信号肽和跨膜结构,与雨生红球藻、水稻等其他物种的 UVR8 研究结果相同(鲍思元,2016;张宏江,2019)。有研究表明,亲水蛋白含有大量亲水氨基酸,过表达时有利于提高植物耐旱、低温和高盐等抗逆性(刘盈盈,2019),说明 UVR8 可能参与植物的抗逆过程。
UVR8蛋白系统进化树主要分为三个亚族,除AvUVR8a和AcUVR8b单独处一亚族外,其余的AvUVR8和AcUVR8与拟南芥聚集于同一亚族。在AvUVR8/AcUVR8主要集聚处其与拟南芥(AtUVR8)和枣(ZjUVR8)的亲缘关系最近,而聚集在该亚族中部的AvUVR8b和AcUVR8a与小粒咖啡(CaUVR8)和伊德斯种咖啡(CeUVR8)的亲缘关系最近。表明在两种罗布麻UVR8主要聚集的亚族上,物种间具有明显的同源关系。顺式作用元件序列位于基因上游通过与转录因子结合的方式调控基因表达,涉及启动子、增强子、调控元件和诱导元件等(刘贺等,2022)。本研究通过UVR8基因顺式作用元件分析,发现UVR8基因的表达不仅受光照影响,还受到温度、水分、氧气和内源激素等因素的调控,说明UVR8基因参与调控植物的生长发育和抗逆过程。研究发现,适当的UV-B辐射剂量对植物生长发育、品质改善、增强保鲜和抵抗逆境胁迫等存在积极调控作用(Jenkins, 2009;刘一诺等,2020)。在UV- B预处理后,番茄中超氧化物歧化酶(SOD)和过氧化氢酶(CAT) 的基因表达和酶活性显著升高。当UVR8功能沉默时UV-B激活的SOD和CAT基因表达下调,抑制UV-B缓解的氧化应激和冷害。表明UVR8参与UV-B诱导的耐寒性,抗氧化酶活性依赖于UVR8(Jiang et al., 2022)。在UV-B胁迫下的基因表达模式分析中,我们发现AvUVR8b、AvUVR8c、AvUVR8e、AcUVR8a和AcUVR8b基因表达量上调,其中AvUVR8c和AcUVR8a表达量随处理时长的增加呈递增趋势,表明这些基因参与植物响应UV-B的过程。
综上所述,AvUVR8b和AcUVR8a蛋白结构、保守基序等与AtUVR8的相关信息最接近,且在UV-B胁迫下其基因表达量上调。推测在植物响应UV-B的过程中,AvUVR8b在罗布麻中起主要作用,AcUVR8a在大麻状罗布麻中起主要作用。以AvUVR8b和AcUVR8a作为重点研究对象,将展开后续UVR8基因功能分析和UV-B胁迫反应研究,为今后深入探究罗布麻属UVR8响应UV-B的分子机制和调控网络等研究提供线索。
参考文献
BAILEY TL, BODEN M, BUSKE FA, et al., 2009. MEME SUITE: tools for motif discovery and searching [J]. NuclAcid Res, 37(Web Server issue): W202-W208.
BAO SY, 2016. In silico cloning and bioinformatics analysis of OsUVR8 gene form rice [J]. Biotechnolgy, 26(2):169-175. [鲍思元,2016. 水稻OsUVR8基因电子克隆及生物信息学分析[J]. 生物技术,26(2): 169-175.]
CALDWELL MM, TERAMURA AH, TEVINI M, 1989. The changing solar ultraviolet climate and the ecologicalconsequences for higher plants [J]. Trend Ecol Evol, 4(12): 363-367.
CHAO JT, LI ZY, SUN YH, et al., 2021. MG2C: a user-friendly online tool for drawing genetic maps [J]. Mol Hortic, 1(1): 1-16.
CHEN CJ, CHEN H, ZHANG Y, et al., 2020. TBtools: An integrative toolkit developed for interactive analyses of big biological data [J]. Mol Plant, 13(8): 1194-1202.
CHEN HZ, NIU JR, HAN R, 2021. Signal transduction pathways of plant ultraviolet B receptor UVR8 [J]. PlantPhysiol J, 57(6): 1179-1188. [陈慧泽,牛靖蓉,韩榕,2021. 植物紫外光B受体UVR8的信号转导途径[J]. 植物生理学报,57(6): 1179-1188.]
CHRISTIE JM, ARVAI AS, BAXTER KJ, et al., 2012. Plant UVR8 photoreceptor senses UV-B bytryptophan-mediated disruption of cross-dimer salt bridges [J]. Science, 335(6075): 1492-1496.
CLOIX C, KAISERLI E, HEILMANN M, et al., 2012. C-terminal region of the UV-B photoreceptor UVR8 initiatessignaling through interaction with the COP1 protein [J]. Proc Natl Acad Sci USA, 109(40): 16366-16370.
FAVORY JJ, STEC A, GRUBER H, et al., 2009. Interaction of COP1 and UVR8 regulates UV-B-inducedphotomorphogenesis and stress acclimation in Arabidopsis [J]. EMBO J, 28(5): 591-601.
FROHNMEYER H, STAIGER D, 2003. Ultraviolet-B radiation-mediated responses in plants. Balancing damage andprotection [J]. Plant Physiol, 133(4): 1420-1428.
GAO LM, LIU Y, WANG XF, et al., 2019. Lower levels of UV-B light trigger the adaptive responses by inducing plantantioxidant metabolism and flavonoid biosynthesis in Medicago sativa seedlings [J]. Funct Plant Biol, 46(10): 896-906.
GASTEIGER E, GATTIKER A, HOOGLAND C, et al., 2003. ExPASy: The proteomics server for in-depth proteinknowledge and analysis [J]. Nucl Acid Res, 31(13): 3784-3788.
GRUBER H, HEIJDE M, HELLER W, et al., 2010. Negative feedback regulation of UV-B-inducedphotomorphogenesis and stress acclimation in Arabidopsis [J]. Proc Natl Acad Sci USA, 107(46): 20132-20137.
HARTMANN U, SAGASSER M, MEHRTENS F, et al., 2005. Differential combinatorial interactions of cis-acting elements recognized by R2R3-MYB, BZIP, and BHLH factors control light-responsive and tissue-specific activation of phenylpropanoid biosynthesis genes [J]. Plant Mol Biol, 57(2): 155-171.
HIDEG E, JANSEN MA, STRID A, 2013. UV-B exposure, ROS, and stress: inseparable companions or loosely linked associates [J]. Trends Plant Sci, 18(2): 107-115.
HU B, JIN JP, GUO AY, et al., 2015. GSDS 2.0: an upgraded gene feature visualization server [J]. Bioinformatics, 31(8): 1296-1297.
HUANG X, OUYANG XH, YANG PY, et al., 2013. Conversion from CUL4-based COP1-SPA E3 apparatus to UVR8-COP1-SPA complexes underlies a distinct biochemical function of COP1 under UV-B [J]. Proc Natl AcadSci USA, 110(41): 16669-16674.
JENKINS GI, 2009. Signal transduction in responses to UV-B radiation [J]. Ann Rev Plant Biol, 60(1): 407-431.
JENKINS GI, 2014a. Structure and function of the UV-B photoreceptor UVR8 [J]. Curr Opin Struct Biol, 29: 52-57.
JENKINS GI, 2014b. The UV-B photoreceptor UVR8: from structure to physiology [J]. Plant Cell, 26(1): 21-37.
JIANG ZF, XU MF, DONG JF, et al., 2022. UV-B pre-irradiation induces cold tolerance in tomato fruit bySIUVR8-mediated upregulation of superoxide dismutase and catalase [J]. Postharvest Biol Technol: 185.
KELLEY LA, MEZULIS S, YATES CM, et al., 2015. The Phyre2 web portal for protein modeling, prediction andanalysis [J]. Nat Protoc, 10(6): 845-858.
KLIEBENSTEIN DJ, LIM JE, LANDRY LG, et al., 2002. Arabidopsis UVR8 regulates ultraviolet-B signal transduction and tolerance and contains sequence similarity to human regulator of chromatin condensation 1 [J]. Plant Physiol, 130(1): 234-243.
LI GL, ZHANG H, XU YQ, et al., 2015. Research progress in plant photoreceptor UVR8 [J]. Plant Physiol J, 51(11):1809-1814. [李国良,张鸿,许泳清,等,2015. 植物紫外光受体UVR8的研究进展[J]. 植物生理学报,51(11):1809-1814.]
LI XK, LIU ZY, REN HS, et al., 2020. Dynamics and mechanism of light harvesting in UV photoreceptor UVR8 [J]. Chem Sci, 11(46): 12553-12569.
LI XT, WU T, YU ZH, et al., 2018. Apocynum venetum leaf extract reverses depressive-like behaviors in chronically stressed rats by inhibiting oxidative stress and apoptosis [J]. Biomed Pharmacother, 100: 394-406.
LING CT, LI X, ZHOU YY, et al., 2021. Phytohormone pathway and molecular response regulated by UV-B radiation[J]. Plant Physiol J, 57(10): 1839-1851. [凌成婷,李想,周应嫄,等,2021. UV-B辐射调控的植物激素路径和分子响应[J]. 植物生理学报,57(10):1839-1851.]
LIU H, HUANG QM, LIU YJ, et al., 2022. Genome-wide identification and bioinformatics analysis of Chrysanthemum indicum bZIP transcription factor [J]. Mol Plant Breed, 20(14): 4586-4600. [刘贺,黄沁梅,刘颖婕,等,2022. 野菊bZIP转录因子全基因组鉴定及生物信息学分析[J]. 分子植物育种,20(14): 4586-4600.]
LIU MX, SUN M, WANG Y, et al., 2012. Arabidopsis UV-B photoreceptor and its light signal transduction in plants[J]. Chin Bull Bot, 47(6): 661-669. [刘明雪,孙梅,王宇,等,2012. 植物UV-B受体及其介导的光信号转导[J]. 植物学报,47(6):661-669.]
LIU YN, AO M, LI B, et al., 2020. Effect of ultraviolet-B(UV-B) radiation on plant growth and development and its application value [J]. Soils Crops J, 9(2): 191-202. [刘一诺,敖曼,李波,等,2020. UV-B辐射对植物生长发育的影响及其应用价值[J]. 土壤与作物,9(2): 191-202.]
LIU YY, 2019. Chaperone-like and catalytic functions in Deinococcus radiodurans of hydrophilic protein DohL involved in protective against oxidative stress [D]. Beijing: Chinese Academy of Agricultural Sciences. [刘盈盈,2019. 耐辐射异常球菌亲水蛋白DohL具有类分子伴侣和核酸内切酶功能并参与氧化胁迫保护[D]. 北京:中国农业科学院.]
O'HARA A, JENKINS GI, 2012. In vivo function of tryptophans in the Arabidopsis UV-B photoreceptor UVR8 [J]. Plant Cell, 24(9): 3755-3766.
PARIHAR P, SINGH S, SINGH R, et al., 2015. Changing scenario in plant UV-B research: UV-B from a generic stressor to a specific regulator [J]. J Photochem Photobiol B-Biol, 153: 334-343.
QIAN CZ, 2019. Molecular mechanism of the subcellular localization and activity of Arabidopsis UV-B photoreceptor [D]. Xiamen: Xiamen University. [钱崇祯,2019. 拟南芥UV-B光受体亚细胞定位与功能研究[D]. 厦门:厦门大学.]
RIZZINI L, FAVORY JJ, CLOIX C, et al., 2011. Perception of UV-B by the Arabidopsis UVR8 protein [J]. Science,332(6025): 103-106.
SHAMALA LF, ZHOU HC, HAN ZX, et al., 2020. UV-B induces distinct transcriptional re-programing in UVR8-signal transduction, flavonoid, and terpenoids pathways in Camellia sinensis [J]. Front Plant Sci, 11: 234.
SONG LX, 2020. Whole genome sequencing and gene family evolution analysis of Apocynum venetum L.[D]. Yinchuan: Ningxia University. [宋立肖,2020. 罗布麻全基因组测序及基因家族进化分析[D]. 银川:宁夏大学.]
SONG LX, LI GQ, JIN CQ, et al., 2019. Whole genome sequencing and development of SSR markers in Apocynum cannabinum [J]. J Plant Genet Resour, 20(5): 1309-1316. [宋立肖,李国旗,靳长青,等,2019. 大麻状罗布麻的全基因组分析和SSR标记开发[J]. 植物遗传资源学报,20(5):1309-1316.]
TOSSI VE, REGALADO JJ, IANNICELLI J, et al., 2019. Beyond Arabidopsis: differential UV-B response mediated by UVR8 in diverse species [J]. Front Plant Sci, 10: 780.
VANDENBUSSCHE F, TILBROOK K, FIERRO AC, et al., 2014. Photoreceptor-mediated bending towards UV-B in Arabidopsis [J]. Mol Plant, 7(6): 1041-1052.
WANG DQ, LI GQ, SU DX, 2012. Effect of drought stress on osmotic adjustment substances and activity of protective enzymes in two species of Apocynum [J]. J Arid Land Resour Environ, 26(12):177-181. [王东清,李国旗,苏德喜,2012. 干旱胁迫对两种罗布麻渗透调节物质积累和保护酶活性的影响[J]. 干旱区资源与环境,26(12):177-181.]
WARGENT JJ, JORDAN BR, 2013. From ozone depletion to agriculture: understanding the role of UV radiation in sustainable crop production [J]. New Phytol, 197(4): 1058-1076.
WINKEL-SHIRLEY B, 2002. Biosynthesis of flavonoids and effects of stress [J]. Curr Opin Plant Biol, 5(3): 218-223. WU D, HU Q, YAN Z, et al., 2012. Structural basis of ultraviolet-B perception by UVR8[J]. Nature, 484(7393):214-219.
XIA QY, 2007. Cloning and expression of venom allergen Soli1 and Soli4 genes in the Solenopsis invicta [D]. Chongqing: Southwest University. [夏巧玉,2007. 红火蚁毒素致敏原Soli1、Soli4基因的克隆及表达[D]. 重庆:西南大学.]
YANG YJ, YANG XL, JANG ZF, et al., 2018. UV RESISTANCE LOCUS 8 from Chrysanthemum morifolium Ramat (CmUVR8) plays important roles in UV-B signal transduction and UV-B-induced accumulation of flavonoids [J]. Front Plant Sci, 9: 955.
ZHANG HJ, HANG W, MA HT, et al., 2019. Gene cloning and bioinformatics analysis of novel ultraviolet-B photoreceptor UV Resistance Locus 8 (UVR8) from green alga Haematococus pluvialis [J]. SW Chin J Agric Sci, 32(9): 2025-2032. [张宏江,杭伟,马浩天,等,2019. 雨生红球藻UVR8的基因克隆和生物信息学分析[J]. 西南农业学报,32(9):2025-2032.]
ZHANG Y, 2021. Protective effect and mechanism of Apocynum venetum leaves flavonoids and isoquercitrin on pirarubicin-induced cardiac injury [D]. Changchun: Jilin University. [张洋,2021. 罗布麻叶总黄酮及异槲皮苷对吡柔比星致心脏损伤的保护作用和机制[D]. 长春:吉林大学.]
文章摘自:车金凤,张庆,李国旗,谢博勋,解盛,赵长海,张柯雨,刘星.罗布麻和大麻状罗布麻UV-B光受体UVR8基因的鉴定及表达分析[J/OL].广西植物:1-15[2022-12-06].http://kns.cnki.net/kcms/detail/45.1134.Q.20221122.2005.002.html