摘 要:本发明公开了一种红麻铅胁迫响应基因EST?SSR引物组及试剂盒。本发明提供22个红麻EST?SSR标记的引物序列。本发明的22个EST?SSR标记在138份红麻品种/品系中具有多态性,可以将红麻种质完全区分开,并将138份材料划分为3个群体,说明红麻种质抵抗铅胁迫能力存在着明显的分群,为筛选耐重金属铅胁迫红麻种质资源、耐重金属铅分子标记辅助育种提供了候选标记。
技术要点
1 .一种红麻铅胁迫响应基因EST?SSR引物组,其特征在于,
所述引物共有22对,具体如下:
第1对引物,其序列如 SEQ ID NO:1和SEQ ID NO:2所示;
第2对引物,其序列如 SEQ ID NO:3和SEQ ID NO:4所示;
第3对引物,其序列如 SEQ ID NO:5和SEQ ID NO:6所示;
第4对引物,其序列如 SEQ ID NO:7和SEQ ID NO:8所示;
第5对引物,其序列如 SEQ ID NO:9和SEQ ID NO:10所示;
第6对引物,其序列如 SEQ ID NO:11和SEQ ID NO:12所示;
第7对引物,其序列如 SEQ ID NO:13和SEQ ID NO:14所示;
第8对引物,其序列如 SEQ ID NO:15和SEQ ID NO:16所示;
第9对引物,其序列如 SEQ ID NO:17和SEQ ID NO:18所示;
第10对引物,其序列如 SEQ ID NO: 19和SEQ ID NO:20所示;
第11对引物,其序列如 SEQ ID NO:21和SEQ ID NO:22所示;
第12对引物,其序列如 SEQ ID NO:23和SEQ ID NO:24所示;
第13对引物,其序列如 SEQ ID NO:25和SEQ ID NO:26所示;
第14对引物,其序列如 SEQ ID NO:27和SEQ ID NO:28所示;
第15对引物,其序列如 SEQ ID NO:29和SEQ ID NO:30所示;
第16对引物,其序列如 SEQ ID NO:31和SEQ ID NO:32所示;
第17对引物,其序列如 SEQ ID NO:33和SEQ ID NO:34所示;
第18对引物,其序列如 SEQ ID NO:35和SEQ ID NO:36所示;
第19对引物,其序列如 SEQ ID NO:37和SEQ ID NO:38所示;
第20对引物,其序列如 SEQ ID NO:39和SEQ ID NO:40所示;
第21对引物,其序列如 SEQ ID NO:41和SEQ ID NO:42所示;
第22对引物,其序列如 SEQ ID NO:43和SEQ ID NO:44所示。
2.一种鉴定筛选耐重金属铅胁迫红麻种质资源的试剂盒,其特征在于,它包括如权利要求1中所述的引物组。
技术领域
本发明涉及EST?SSR标记,具体而言,是红麻铅胁迫响应基因EST?SSR引物组及试剂盒,属于生物技术领域。
背景技术
红麻是一种重要的韧皮纤维作物、麻纺原料,是绿色环保天然纤维制品的重要原材料之一。红麻可富集土壤中的重金属,对修复酸性土壤起到重要作用。红麻资源的研究还停留在植物学性状、农艺性状、品质性状的描述上,缺乏抗逆性状等在分子层面的研究。开发功能基因的分子标记,可加快耐重金属红麻材料的筛选进程,减少亲本选择的工作量和时间。
简单重复序列多态性(Simple sequence repeats ,SSR)是继形态学标记、同工酶标记之后的第三代标记的代表技术之一。SSR和SNP标记是目前两种最主流的分子标记技术,与SNP标记的二等位、易于自动化高通量分析不同,SSR分子标记与SNP一样,属于共显性标记,但是其多态性远高于SNP,在遗传多样性评价、群体结构分析、亲缘关系鉴定和指纹图谱等领域有着广泛的应用。
目前红麻分子标记的研究和应用也已开展,主要应用于遗传多样性研究、遗传连锁图谱构建和重要农艺性状QTL定位,使用的分子标记技术为包括SRAP,RAPD,AFLP,ISSR和SSR。总体来说,红麻的分子标记数量仍然非常少,尤其是目前主流的分子标记技术SNP和SSR分子标记与其他作物相比落后许多。开发新的分子标记尤其是SSR和SNP分子标记有利于加快红麻遗传改良进程。
随着二代高通量测序成本的降低,转录组测序成为SSR分子标记开发的重要途径之一,这种开发SSR分子标记的策略,在红麻中也有报道。EST?SSR分子标记是外显子区的简单重复序列差异,会导致基因编码的蛋白氨基酸产生剧烈变异,例如氨基酸插入/缺失,移码突变或提前终止。因此,表达序列标签简单序列重复多态性(EST?SSR)分子标记可能会直接引起基因功能缺陷,如果其所在基因是控制重要农艺性状的基因,其基因外显子区的SSR序列变异可能导致基因功能缺失,从而引起性状的变异。基于此,使用EST?SSR标记应用于数量性状定位,可以加速QTL定位确定候选基因。
基于转录组的分子标记开发,根据转录组测序的实验目的和采样方式不同,比如不同生物、非生物胁迫,不同生长发育阶段,不同的组织器官等,所得到的表达基因及差异表达基因DEGs也有所不同。
发明内容
为了克服现有技术的不足,本发明提供了一组红麻铅胁迫响应基因EST?SSR引物及试剂盒。从铅胁迫转录组测序拼接unigene序列中,开发EST?SSR分子标记,并分析了转录组中SSR的分布规律,包含SSR的基因潜在功能,选择蛋白功能互作网络(PPI)基因DEGs设计了57对新的EST?SSR标记。利用30个红麻品种/品系中进行多态性标记筛选,经过琼脂糖凝胶电泳和ABI3730xl毛细管电泳两轮筛选,最终得到了22对检出率高,多态性好的EST?SSR标记。这些红麻铅胁迫差异表达基因的EST序列尚未提交到NCBI数据库,是新的EST序列,开发得到的22个新EST?SSR标记也不同于已报道的红麻SSR标记。
一种红麻铅胁迫响应基因EST?SSR引物组,所述引物共有22对,具体如下:
第1对引物,其序列如SEQ ID NO:1和SEQ ID NO:2所示;
第2对引物,其序列如SEQ ID NO:3和SEQ ID NO:4所示;
第3对引物,其序列如SEQ ID NO:5和SEQ ID NO:6所示;
第4对引物,其序列如SEQ ID NO:7和SEQ ID NO:8所示;
第5对引物,其序列如SEQ ID NO:9和SEQ ID NO:10所示;
第6对引物,其序列如SEQ ID NO:11和SEQ ID NO:12所示;
第7对引物,其序列如SEQ ID NO:13和SEQ ID NO:14所示;
第8对引物,其序列如SEQ ID NO:15和SEQ ID NO:16所示;
第9对引物,其序列如SEQ ID NO:17和SEQ ID NO:18所示;
第10对引物,其序列如SEQ ID NO:19和SEQ ID NO:20所示;
第11对引物,其序列如SEQ ID NO:21和SEQ ID NO:22所示。
第12对引物,其序列如SEQ ID NO:23和SEQ ID NO:24所示;
第13对引物,其序列如SEQ ID NO:25和SEQ ID NO:26所示;
第14对引物,其序列如SEQ ID NO:27和SEQ ID NO:28所示;
第15对引物,其序列如SEQ ID NO:29和SEQ ID NO:30所示;
第16对引物,其序列如SEQ ID NO:31和SEQ ID NO:32所示;
第17对引物,其序列如SEQ ID NO:33和SEQ ID NO:34所示;
第18对引物,其序列如SEQ ID NO:35和SEQ ID NO:36所示;
第19对引物,其序列如SEQ ID NO:37和SEQ ID NO:38所示;
第20对引物,其序列如SEQ ID NO:39和SEQ ID NO:40所示;
第21对引物,其序列如SEQ ID NO:41和SEQ ID NO:42所示;
第22对引物,其序列如SEQ ID NO:43和SEQ ID NO:44所示。
一种鉴定筛选耐重金属铅胁迫红麻种质资源的试剂盒,包括所述的引物组。
本发明的有益效果是:
本发明从铅胁迫转录组测序拼接unigene序列中,开发EST?SSR分子标记,并分析了转录组中SSR的分布规律,包含SSR的基因潜在功能,选择蛋白功能互作网络(PPI)基因DEGs设计了57对新的EST?SSR标记。我们利用30个红麻品种/品系中进行多态性标记筛选,经过琼脂糖凝胶电泳和ABI3730xl毛细管电泳两轮筛选,最终得到了22对检出率高,多态性好的EST?SSR标记。用138个红麻种质资源验证了这些新EST?SSR标记在红麻种质资源遗传多样性和群体结构分析的适用性。发现这22个EST?SSR标记遗传多样性丰富,可以将红麻种质完全区分开,并将138份材料划分为3个群体,红麻种质抵抗铅胁迫能力存在着明显的分群。这为今后研究红麻种群结构,筛选耐重金属胁迫种质提供了重要的信息。这些红麻铅胁迫差异表达基因的EST序列尚未提交到NCBI数据库,是新的EST序列,开发得到的22个新EST?SSR标记也不同于已报道的红麻SSR标记。
附图说明
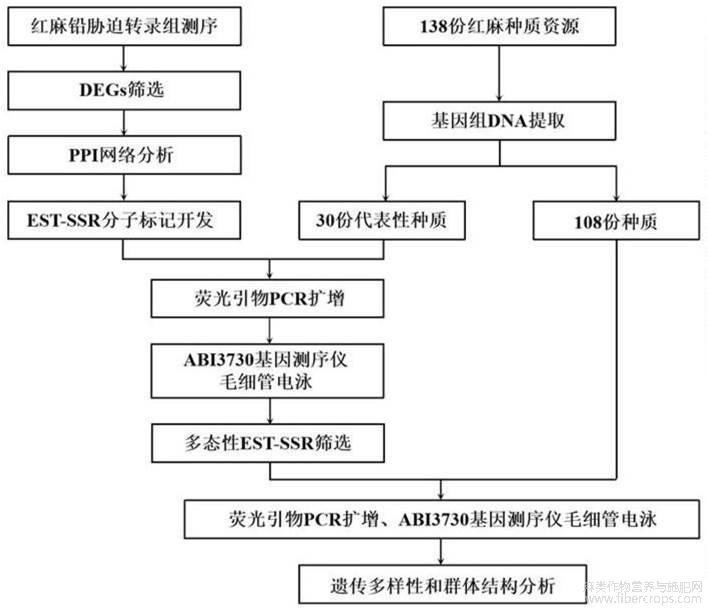
图1是红麻响应铅胁迫关键基因EST?SSR标记开发流程图。
图2是最大PPI成员基因富集分析。
图3是红麻种质资源群体结构分析。
具体实施方式
红麻响应铅胁迫关键基因的EST?SSR标记,是对红麻转录组测序数据和红麻材料搜集、SSR标记开发、GO和KEGG富集分析、差异基因蛋白互作网络分析、基因组DNA提取、SSR分型、遗传多样性分析、遗传结构分析等过程得到的(图1)。
本发明利用红麻转录组测序数据得到铅胁迫关键基因开发红麻EST?SSR引物的具体方法是:
一、红麻转录组测序数据和材料搜集
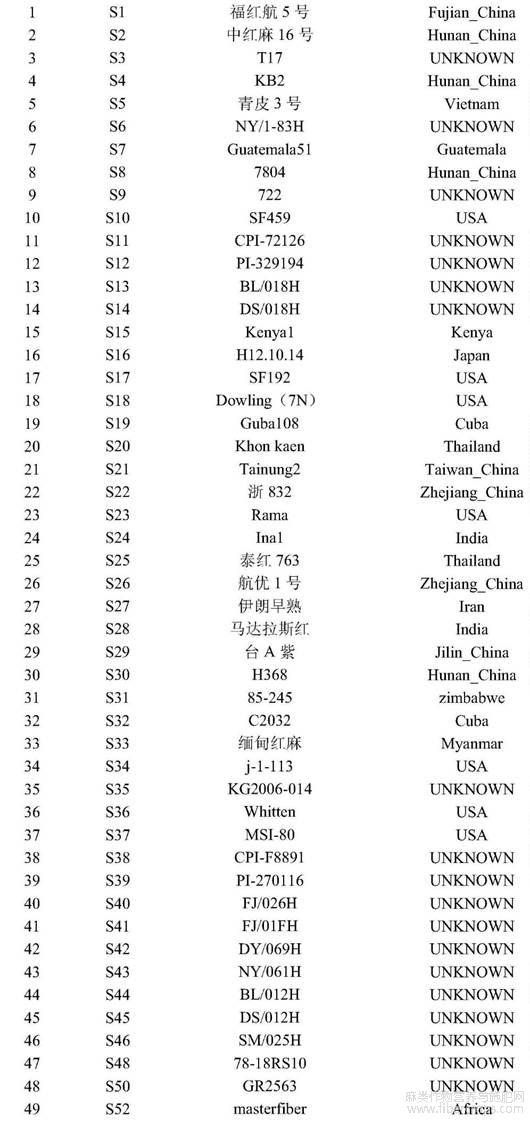
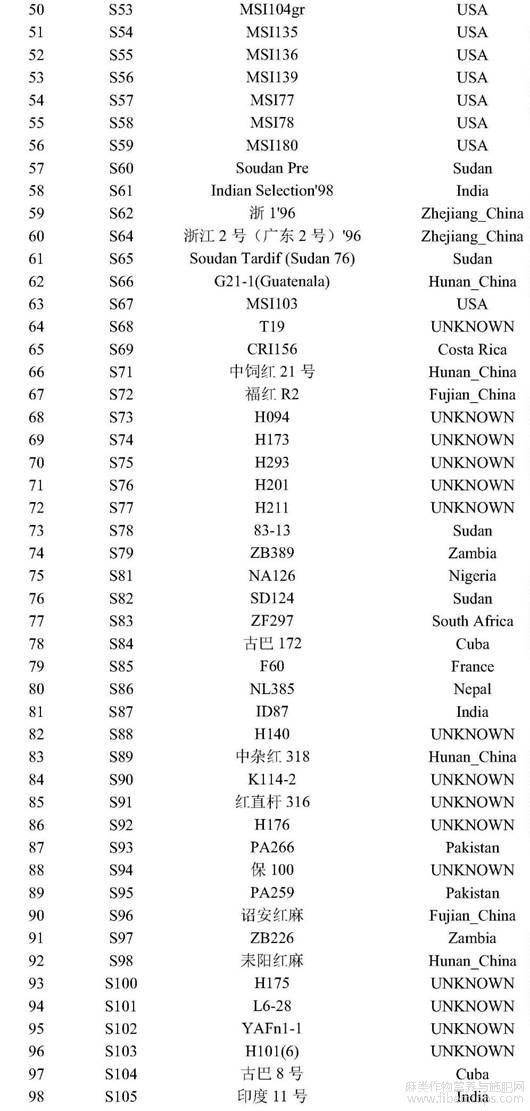
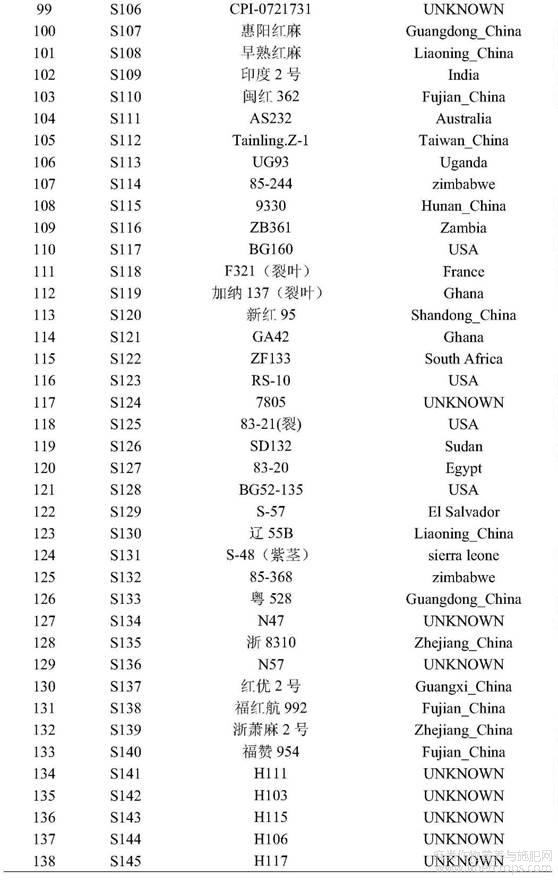
转录组的数据在NCBI SRA:SRR9163842 .to SRR9163845 .本研究所用红麻种质资源及其地域来源如表1所示,总计138份红麻种质,包括来自中国9个省的30份、Pakistan2份,France2份,Cuba4份,Ghana2份,zimbabwe3份,USA19份,South Africa2份,Sudan5份,Thailand2份,India6份,Zambia3份,其他15个国家Egypt、Australia、Africa、Costa Rica、Kenya、Myanmar、Nepal、Nigeria、Japan、El Salvador、sierra leone、Guatemala、Uganda、Iran、Vietnam等各1份种质,地域来源未知的43份种质。其中编号S1?S30共30份种质用于EST?SSR分子标记的初步筛选和验证;总体138份红麻种质应用于22对高质量EST?SSR分子标记(表2)的应用评价。
表1 红麻种质资源及其地域来源
表2 麻EST?SSR标记特征
二、SSR标记开发
转录组测序得到的Unigene序列,导入MISA软件进行SSR分析,统计SSR motif的类型,频数分布等。选择SSR位点的重复基元,以单核苷酸为重复单位时,其重复次数≥10,二核苷酸重复次数≥6次,三、四、五、六核苷酸重复次数≥5次进行SSR检测。找出序列中简单重复序列元件后,以Unigene的序列为参考,利用Primer3批量设计SSR引物,PCR产物长度在100?400bp之间。
三、GO和KEGG富集分析
使用TBTools软件对包含SSR的Unigene和包含SSR的PPI网络中DEGs进行GO和KEGG富集分析,解析Unigene和PPI中包含SSR的基因的共性,其参与的主要生物学过程,分子生物学功能,细胞组分,以及参与的代谢途径特征。
四、差异基因蛋白互作网络分析
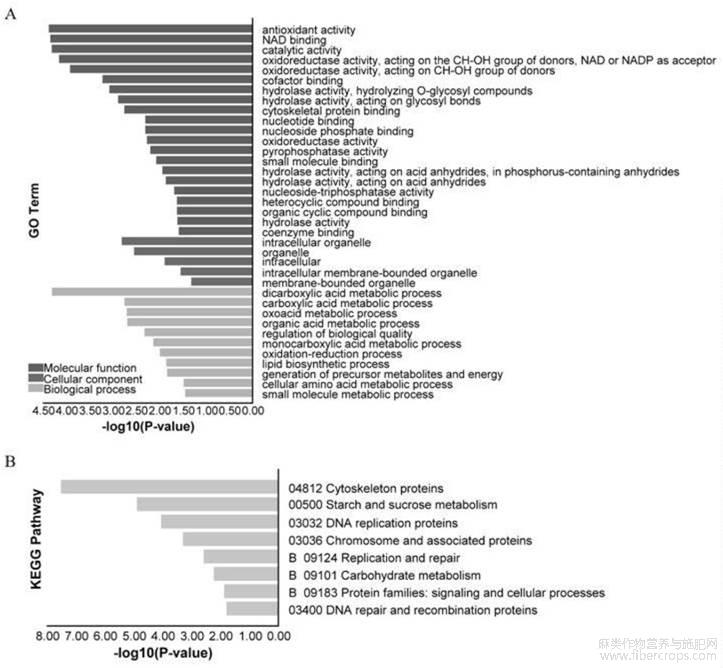
使用String(https://string?db.org/)分析差异表达基因的蛋白功能互作网络,并利用Cytoscape软件绘制和编辑互作网络图。最大的PPI中有91个基因,富集分析发现(图2A部分),主要参与dicarboxylic acid metabolic process,carboxyic acid metabolicprocess和oxoacid metabolic process等生物学过程,主要富集在antioxidantacitivty、NAD binding和catalytic activity等分子功能 ,GO_CC富集主要是intracellular organelle、organelle和intracellulsr等。KEGG富集分析发现,这些PPI基因主要富集在cytoskeleton proteins,starch and sucrose metabolism和DNAreplication proteins等代谢途径(图2B部分)。
五、基因组DNA抽提
取200mg嫩叶,采用Tiangen DNAsecure新型植物基因组DNA提取试剂盒(货号DP320?02)提取红麻基因组DNA,具体操作步骤参考试剂盒说明书。提取完成后,取2μl使用Thermo微量紫外分光光度计NanoDrop(型号ND1000)测定DNA浓度及A260/280,4μl体积在1%浓度的琼脂糖凝胶上进行电泳,检测DNA完整性,主带清晰且不拖尾的DNA用于后续SSR分型实验。
六、SSR分型
根据引物序列,合成荧光SSR引物,在引物5’端添加FAM荧光基团。DNA聚合酶使用Takara DNA聚合酶(TransGen Phi29 DNA Polymerase,货号LP101?01)。PCR体系总体积为20μl,DNA模板2μl,Buffer 2μl,TransTaq 0 .3μl,dNTP1 .6μl,ddH2O 12.1μl,正反向引物各1μl(浓度2μmol/μl)。PCR扩增程序设置为预变性94℃4min,变性94℃30s→退火56℃90s→延伸72℃1min,这3个阶段循环35次,延伸72℃5min,4℃保存。PCR扩增结束后,取1μl PCR产物,在ABI3730xl毛细管电泳仪上进行电泳,电泳结束后,GeneMapper4 .0软件读数,导出SSR基因分型信息。
七、遗传多样性分析
SSR分型数据根据各软件格式要求进行转换。将基因型格式文件导入POPGENE1 .32软件,选择二倍体共显性数据格式,分析单个SSR位点在样本整体的等位基因数(Na)、有效等位基因数(Ne)、观测杂合度(Ho)、期望杂合度(He)、Shannon’s多样性指数(I)。Powermarker3.25软件计算各位点的多态性信息含量(PIC)。利用NTSYSPC2.10e软件计算样本两两间的Jaccard遗传相似系数,导出样本两两间遗传相似系数矩阵。
八、遗传结构分析
使用Powermarker3.25软件计算基于等位基因频率的Nei’s遗传距离,使用邻位相连聚类法(Neighbor?Joining clustering)绘制聚类树,并使用MEGA7.0软件对聚类树进行编辑美化。将SSR分型数据导入Structure2.0软件进行分析,K设为从1到20,MCMC(Markov Chain Monie Carfo)和不作数迭代值(length of burn?in period)分别设为10000和100000,每个K值运行重复运行3次。将Structure运行的results文件上传至Structure Harvester在线工具进行分析,绘制ΔK值随K值的变化曲线,根据峰值判断最佳K值,将最佳K值对应的3次重复运行结果indfile下载并导入Clumpp2.0软件将3次结果合并为1个Q值矩阵,利用Structure2.0软件绘制Q plot。对比NJ聚类和Structure的分析模型,解析群体遗传结构。基于这22个SSR位点的分型数据,我们对这138份红麻材料的群体遗传结构进行了分析。Strucuture软件的先验模型推测每个材料划分入特定亚群的概率,结果导入structure harvester分析ΔK值随着K值的变化曲线如图3A所示,K=3时,ΔK为最大值的拐点,这138份材料最佳亚群数目为3个。我们将最佳K值对应的3次重复的Q矩阵使用Clumpp合并,绘制Q plot(图3B)。
最后,还需要注意的是,以上所述仅是本发明的若干个具体实施例而已,本发明不限于以上实施例,还可以有各种改动和变形。
序列表
<110> 浙江省萧山棉麻研究所
<120> 红麻铅胁迫响应基因EST?SSR引物组及试剂盒
<141> 2020?12?31
<160> 44
<170> SIPOSequenceListing 1 .0
<210> 1
<211> 20
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 1
gtcgaccggg tctacgaatc 20
<210> 2
<211> 20
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 2
tcaactacgg tagggcagga 20
<210> 3
<211> 20
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 3
cctccgttct taccaccacc 20
<210> 4
<211> 20
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 4
tcccaaagga atgcccaaca 20
<210> 5
<211> 22
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 5
agaaaaggcc attcctgatc ca 22
<210> 6
<211> 20
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 6
attggtggac tgcagtacgg 20
<210> 7
<211> 20
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 7
tgtaacccac ggtggctttt 20
<210> 8
<211> 27
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 8
cacattacaa atctaaacct tccctga 27
<210> 9
<211> 20
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 9
cgtcgaatca gtcatgctgc 20
<210> 10
<211> 24
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 10
tgctcattgc tcaatagatc aaga 24
<210> 11
<211> 20
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 11
ctcttccaac gcagccaaac 20
<210> 12
<211> 20
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 12
aatgggtttt ccgacaccga 20
<210> 13
<211> 20
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 13
acggccgact ttcagaatgt 20
<210> 14
<211> 20
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 14
tccaggctcc agctatctca 20
<210> 15
<211> 20
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 15
aggctttcgt tgctcaccat 20
<210> 16
<211> 20
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 16
tttggaggca cgggagattc 20
<210> 17
<211> 20
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 17
ttccggtgca gatagggaga 20
<210> 18
<211> 20
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 18
acaaggaaac agaggcagca 20
<210> 19
<211> 20
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 19
ccaccgtatt ctcatcggca 20
<210> 20
<211> 20
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 20
ctccaatcca tcggagcctc 20
<210> 21
<211> 20
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 21
cacatcccat ttgaccccca 20
<210> 22
<211> 20
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 22
tctgtgccaa tggaagagca 20
<210> 23
<211> 21
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 23
cccgtctcaa attctcagcc a 21
<210> 24
<211> 20
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 24
cgaaatgcca gcgttgttca 20
<210> 25
<211> 20
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 25
acctctctct gtgtttccgc 20
<210> 26
<211> 20
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 26
ccagtcacca gcacgtactt 20
<210> 27
<211> 20
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 27
cattgcctgc cagaccaaac 20
<210> 28
<211> 21
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 28
ggtgtccaca tggtattcgg t 21
<210> 29
<211> 20
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 29
tgaggccctc tgaggctaat 20
<210> 30
<211> 20
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 30
cgacgactct aacaagcggt 20
<210> 31
<211> 20
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 31
gcagactgca gaagaccctt 20
<210> 32
<211> 21
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 32
tctttctcac cacagttgac a 21
<210> 33
<211> 20
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 33
aaacgatgtc aggcgtcagt 20
<210> 34
<211> 20
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 34
cactgtgtgg cgtgtttcag 20
<210> 35
<211> 20
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 35
tgtgctctgt actggcaagt 20
<210> 36
<211> 20
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 36
gcatgaactc aactccccga 20
<210> 37
<211> 20
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 37
gcaggggtgg ggttgttatt 20
<210> 38
<211> 20
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 38
acaggagagt gtggggagaa 20
<210> 39
<211> 20
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 39
tgttggcagc ctatgaagca 20
<210> 40
<211> 20
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 40
cggtttgggg gagcaaagta 20
<210> 41
<211> 20
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 41
caaccagtac tctaccggcc 20
<210> 42
<211> 20
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 42
gaacagttgg gaggactgca 20
<210> 43
<211> 20
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 43
tgttggcagc ctatgaagca 20
<210> 44
<211> 20
<212> DNA
<213> 人工序列(Artificial Sequence)
<400> 44
cggtttgggg gagcaaagta 20
图1
图2
图3
摘自国家发明专利,发明人:安霞,金关荣,柳婷婷,李文略,骆霞虹,陈常理,朱关林,申请号202011641431.8,申请日2021.03.26