摘 要:CsMIXTA在调控大麻雌性花器官中腺毛的发生和发育中发挥重要作用。为了研究CsMIXTA基因的表达调控,对CsMIXTA基因上游2199bp启动子序列进行了克隆。通过PlantCARE预测发现,该启动子序列具有多个胁迫响应元件与光响应元件。根据预测的响应元件分布情况,扩增获得5个5′端缺失的启动子片段。用克隆的启动子全长和缺失片段分别构建融合GUS基因的表达载体,并将其瞬时转化到烟草叶片和工业大麻糖叶中。通过GUS染色观察发现,CsMIXTA启动子的核心区域位于-393~-99bp之间,包含赤霉素响应元件TATC-box和转录起始元件TATA-box。通过LUC荧光素酶表达发现,CsMIXTA启动子的核心区域具有转录活性。并且研究还发现,CsMIXTA基因在工业大麻糖叶的腺毛中特异性表达。对启动子进行胁迫响应分析的结果显示,低温、脱落酸(ABA)和赤霉素(GA3)都能增强该启动子的活性。这些结果为CsMIXTA基因调控机制研究奠定了重要基础。
关键词:工业大麻;CsMIXTA启动子;瞬时表达;GA3、ABA处理
大麻(Cannabis sativa L.)是大麻科大麻属的一年生草本植物,也被称为工业大麻、火麻、汉麻[1]。大麻可以产生丰富的次级代谢产物,比如萜烯类、大麻素等,其中包括具有重要医用潜力的大麻二酚(Cannabidiol,CBD)[2]。大麻中的腺毛是一种特殊的表皮毛结构,是大麻次生代谢物产生的重要工厂[3]。研究发现通过增加光照或改善营养条件,能增加大麻中这些次级代谢物的含量[4]。此外,使用包括植物激素在内的多种植物生长调节剂也可以增加次级代谢物的含量[5]。
研究表明,R2R3-MYB家族的一些成员可以调控表皮细胞发育[6]。例如,在金鱼草中发现MIXTA基因控制着锥形表皮细胞的分化[7]。青蒿中的AaMYB1和AaMIXTA1[8-9],番茄中的SlMX1[10]被确定为腺毛启动的正调控因子。过表达AaMYB1基因可以增加青蒿植株的腺毛密度[8-9],而AaMIXTA1和SlMX1的下调则会降低腺毛密度[10]。大麻R2R3-MYB家族基因同样对腺毛发育起重要作用[11]。研究发现大麻R2R3-MYB转录因子CsMIXTA在雌花中显著表达。CsMIXTA在烟草中过表达后,烟草叶片的腺毛大小与密度明显增加,且腺毛分支也显著增加。这些结果表明CsMIXTA可能调控大麻雌性花器官中腺毛的发生和发育[12]。然而,目前该基因的调控机制与组织表达特性尚不明确。
为了深入探究大麻CsMIXTA基因的功能和调控机制,本研究克隆了CsMIXTA基因2199bp启动子序列,并分析了该启动子序列的顺式作用元件。通过5′端启动子缺失融合GUS基因在烟草和工业大麻中进行瞬时表达,发现了该启动子的核心启动区和组织表达特异性,并初步验证了其顺式作用元件的功能。这些结果为进一步研究CsMIXTA基因的功能和调控机制提供了参考。
1材料与方法
1.1材料与试剂
本研究使用中国农业科学院麻类研究所保存的高CBD工业大麻种质‘DMG265’为试验材料,培养条件为光强471μmolm-2s-1,营养期光周期为18h光照/6h黑暗,花期光周期为12h光照/12h黑暗,湿度50%~60%,温度(25±2)℃。本氏烟草(Nicotianabenthamiana)种子由中国农业科学院麻类研究所特色蔬菜团队提供,培养条件为光强471μmolm-2s-1、光周期18h光照/6h黑暗、湿度50%~60%、温度(25±2)℃。采集成熟期雌花的糖叶,并立即用液氮暂存,随后放入-80℃冰箱保存,用于提取总DNA。
胶回收试剂盒购自OMEGA公司;高保真酶ApexHFHSDNA聚合酶混合液FS、PCR酶AccurateTaqDNA预混液、DL2199Marker、DL10000Marker购自艾科瑞生物公司;限制性内切酶BamHⅠ购自NewEnglandBiolabs公司;DNA提取试剂盒购自TIANGEN公司;质粒提取试剂盒、同源重组试剂盒购自诺唯赞公司;Trelief5αChemicallyCompetentCell感受态细胞、农杆菌GV3101、pClone007购自北京擎科生物科技股份有限公司;GUS染色液、SilwettL-77购自北京酷来搏科技有限公司;RNA提取试剂盒购自北京艾德莱生物公司;MES、MgCl2、葡萄糖、乙酰丁香酮、抗坏血酸购自上海麦克林生化科技股份有限公司;PluronicF-68购自赛默飞世尔科技有限公司;MS培养基购自青岛高科技工业园海博生物技术有限公司;由北京擎科生物科技股份有限公司进行引物合成及测序。植物启动子表达载体pCAMBIA1391、pNC-Green-LUC由本实验室保存。
1.2CsMIXTA启动子克隆与分析
按DNA提取试剂盒说明书提取保存工业大麻‘DMG265’糖叶的总DNA。利用NCBI上的大麻基因组数据库(Cs10:GCA_900626175.2)获取CsMIXTA基因的基因组信息(LOC115701410),分析提取其起始ATG上游的启动子序列,使用Snapgene软件设计启动子克隆引物,命名为MT-F与MT-R(表1)。以总DNA为模板对CsMIXTA基因起始ATG上游2199bp进行克隆。通过胶回收试剂盒回收克隆的片段,并通过pClone007试剂盒进行T载连接。将连接产物转化到Trelief5α感受态细胞中,通过菌落PCR鉴定获得阳性克隆。阳性克隆菌液质粒送北京擎科生物科技股份有限公司测序,并将测序结果与已知序列在Snapgene软件上进行比对确认。
1.3CsMIXTA启动子生物信息学分析
通过PlantCARE数据库(https://bioinformatics.psb.ugent.be/webtools/plantcare/html/)预测上述克隆获得的CsMIXTA基因启动子序列的顺式作用元件的功能与位点。通过网站Softberry(http://www.softberry.com)对CsMIXTA启动子序列进行分析,预测其转录起始位点。
1.4CsMIXTA启动子5′端缺失克隆
根据预测获得的顺式作用元件的功能与位点,以已测序的2199bp启动子片段为模板进行5′端缺失克隆。以已测序的启动子全长质粒为模板进行PCR扩增(引物序列见表1),通过胶回收试剂盒回收克隆的片段,并通过pClone007试剂盒进行T载连接。将连接产物转化到Trelief5α感受态细胞中,平板培养后挑取单菌落并进行PCR鉴定,获得阳性克隆后进行测序确认。
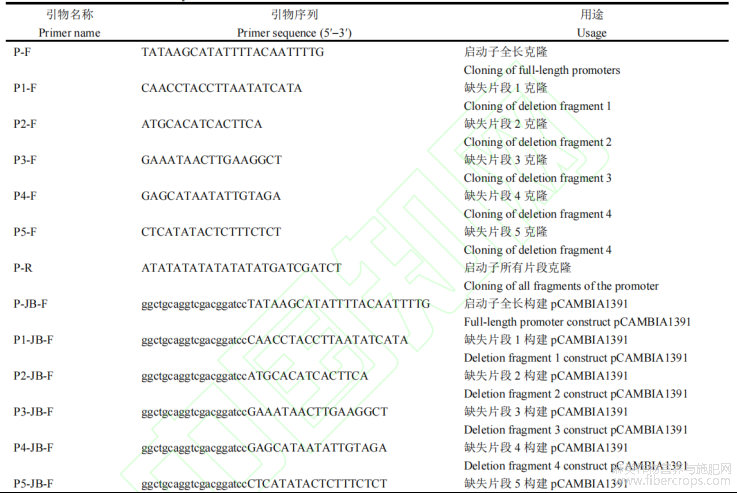
表1本研究所用引物
1.5CsMIXTA植物启动子表达载体构建
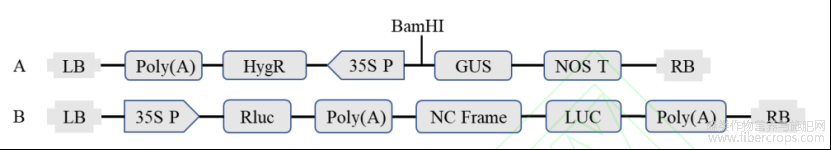
使用植物启动子表达载体pCAMBIA1391(图1)对CsMIXTA基因启动子的表达活性进行分析。依据载体上的多克隆位点信息,选用BamHⅠ单个限制性内切酶,对启动子全长以及缺失片段设计同源重组加臂引物(表1)。以启动子全长以及缺失片段质粒为模板进行PCR克隆,纯化扩增产物后使用同源重组试剂盒将其与经过BamHⅠ单酶切的pCAMBIA1391载体进行同源重组连接。将同源重组液转化到Trelief5α感受态细胞中,平板培养后挑取单菌落并进行PCR鉴定,获得阳性克隆后进行测序确认。
使用荧光素酶表达载体pNC-Green-LUC载体对CsMIXTA基因核心启动子区的表达活性进行分析。该载体为NC克隆载体,使用NC克隆通用接头对CsMIXTA基因核心启动子片段进行加臂(表1)。以核心启动子片段为模板进行PCR克隆,纯化扩增产物后使用NC克隆试剂盒将其与pNC-Green-LUC载体进行连接。将同源重组液转化到Trelief5α感受态细胞中,平板培养后挑取单菌落并进行PCR鉴定,获得阳性克隆后进行测序确认。
图1植物表达载体构建示意图
A:携带GUS的pCAMBIA1391;B:携带LUC的pNC-Green-LUC;Poly(A):花椰菜花叶病毒多聚腺苷酸化信号;HygR:潮霉素抗性基因;35SP:花椰菜花叶病毒35S启动子;NOST:胭脂碱合酶基因终止子;Rluc:海肾荧光素酶;NCFrame:NC克隆框架;GUS:葡糖苷酸酶;LUC:萤火虫荧光素酶。
1.6农杆菌介导的烟草瞬时转化
将构建好的植物启动子表达载体质粒转化到农杆菌GV3101感受态细胞中,平板培养后挑取单菌落并进行PCR鉴定,获得阳性后将单菌落接入LB液体培养基中,28℃培养至OD600为2,4000×g离心5min集菌,接着加入含有10mmolL−1MES、10mmolL−1MgCl2和200mmolL−1乙酰丁香酮的重悬液(pH5.6),调节农杆菌浓度使其OD600为1,并在室温避光环境中静置2~6h。选用移栽后30~40d的本氏烟草,将含有启动子表达质粒的农杆菌注射至完全展开的上部2片叶片中,并使用pCAMBIA1391空载体作为为阴性对照。
1.7农杆菌介导的工业大麻瞬时转化
将1.2.5中使用的全长启动子表达载体质粒阳性单菌落农杆菌培养物重悬于含10mmolL−1MES、1×MS、2%葡萄糖、0.015%SilwettL-77、0.05%PluronicF-68、5mmolL−1抗坏血酸和200µmolL−1乙酰丁香酮的重悬液中,pH为5.6。将进入花期1周后的工业大麻植株倒置浸泡在重悬液中,并放入真空室,打开真空泵降低压力,待压力值达到80mbar后计算渗透时间。在低压下5~15min后,缓慢打开释放阀,使农杆菌进入植物组织间隙。
1.8烟草与工业大麻GUS组织化学染色
在农杆菌注射后的烟草叶片中打孔,获得直径为5mm的注射烟草叶盘。将叶盘放入GUS染液中,室温避光条件下静置过夜。取出染色后的叶片,进行3~4次70%乙醇脱水,直到阴性对照呈白色。最后,在体式显微镜下观察并拍摄样品。
将侵染后的工业大麻糖叶摘下,放入GUS染液中,室温避光条件下静置过夜。取出染色后的叶片,进行3~4次70%乙醇脱水,直到阴性对照呈白色。最后,在体式显微镜下观察并拍摄样品。
1.9烟草荧光素染色
在农杆菌注射后的烟草叶背上喷施1mmolL−1荧光素酶底物,随后室温避光处理7min,放入植物活体成像仪中观察并拍摄样品。
1.10不同胁迫环境下工业大麻糖叶中CsMIXTA表达分析
分别使用100μmolL-1赤霉素和100μmolL-1脱落酸喷施于开花期的工业大麻‘DMG265’糖叶。处理24h后,检测糖叶中的CsMIXTA表达活性。
将工业大麻‘DMG265’置入4℃植物培养箱中,处理24h后检测糖叶中的CsMIXTA表达活性。
本研究使用Bio-RadCFX进行qPCR分析。qPCR反应内参为BUQ5[13],CsMIXTA引物见表1。试验采用4个生物重复。
2结果与分析
2.1CsMIXTA启动子克隆
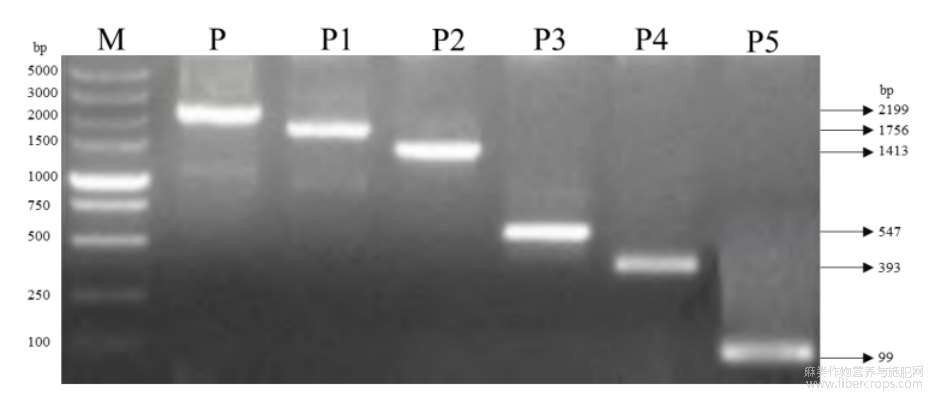
以工业大麻‘DMG265’糖叶的DNA为模板,经过PCR克隆获得2199bp的产物(图2),构建T载测序比对显示该片段与基因组Cs10中的CsMIXTA基因的启动子区序列一致,成功获得CsMIXTA基因启动子片端。
图2CsMIXTA启动子的克隆
M:DNA分子量标准DL2000;P:全长启动子。
2.2CsMIXTA启动子序列分析
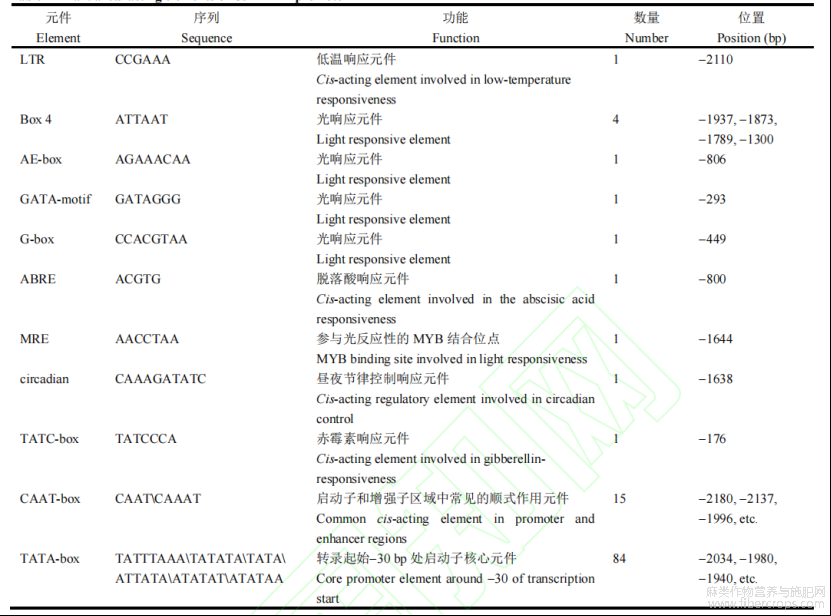
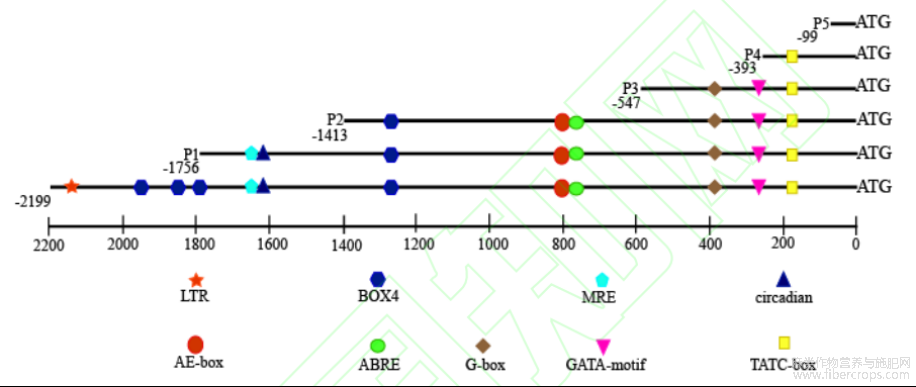
利用在线网站PlantCARE对克隆的CsMIXTA基因启动子序列进行顺式作用元件预测发现,除具有启动子的基本元件84个TATA-box和15个CAAT-box外,该启动子序列还具有光响应元件(Box4、AE-box、GATA-motif、G-box)、参与光反应性的MYB结合位点(MRE)、昼夜节律控制元件(circadian)、低温响应元件(LTR)、赤霉素响应元件(TATC-box)、脱落酸响应元件(ABRE)等(图3和表2)。
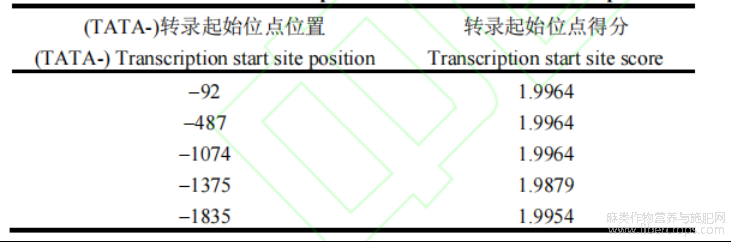
通过网站Softberry中的TTSplantTool在线程序对克隆的CsMIXTA基因启动子序列的转录起始点进行预测,结果得到5个评分较高的转录起始点分别位于-92bp、-487bp、-1074bp、-1375bp、-1835bp(表3)。
图3CsMIXTA启动子序列分析
起始密码按照ATG的“G”为+1位点,红色下画线标注的“G”为转录起始位点。
表2 CsMIXTA启动子部分顺式作用元件
表3 CsMIXTA启动子转录起始位点预测
2.3CsMIXTA启动子缺失片段克隆
根据PlantCARE预测的顺式作用元件将克隆获得的CsMIXTA基因上游2199bp启动子片段分为5段启动子缺失片段。经过克隆获得了长度分别为1756、1413、547、393、99bp的5个5′端缺失启动子片段,分别命名为P1、P2、P3、P4、P5(图4)。与全长启动子序列P相比,P1片段缺失了低温响应元件LTR;P2片段缺失了昼夜节律控制元件circadian;P3片段缺失了光响应元件G-Box和脱落酸响应元件ABRE;P4片段缺失了光响应元件GATA-motif和G-box;P5片段缺失了赤霉素响应元件TATC-box,只含有TATA-box元件(图5)。
图4CsMIXTA启动子缺失片段克隆
M:DNA分子量标准DL2000;P:全长启动子;P1~P5:启动子缺失片段1~5。
图5CsMIXTA启动子全长及缺失片段示意图
LTR:低温响应元件;Box4:光响应元件;MRE:参与光反应性的MYB结合位点;circadian:昼夜节律控制响应元件;AE-box:光响应元件;
ABRE:脱落酸响应元件;G-box:光响应元件;GATA-motif:光响应元件;TATC-box:赤霉素响应元件;图中数值单位为bp。
2.4CsMIXTA全长及不同缺失启动子作用下烟草瞬时表达分析
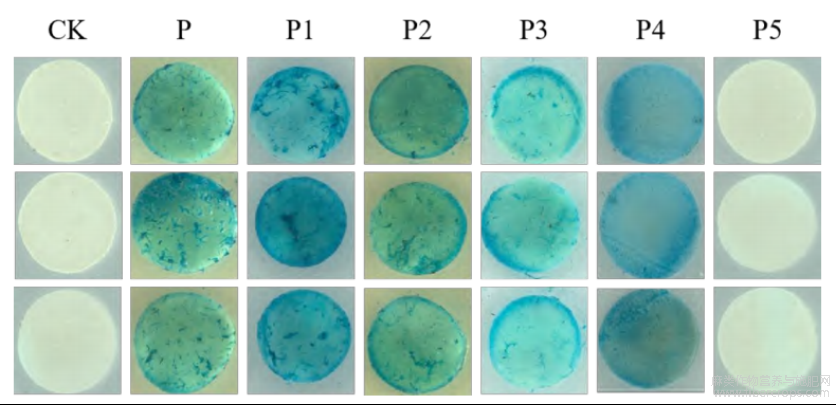
为了验证CsMIXTA启动子的核心启动区与表达活性,将全长及缺失启动子片段同源重组到GUS表达质粒中,瞬时转化到本氏烟草中,2d后取样进行GUS染色。由图6可知,阴性对照(CK)为注射了pCAMBIA1391载体的烟草叶片,呈现无色,证明该载体不具备启动活性。全长启动子片段P,缺失片段P1、P2、P3和P4注射的烟草叶片被染成不同成程度的蓝色,证明CsMIXTA基因启动子全长以及前4个缺失序列都具备启动GUS基因表达的能力。而启动子缺失片段P5注射的烟草叶片呈现无色,说明该缺失片段不具备启动GUS基因的能力,由此确定启动子核心区域位于P4和P5之间,即-393~-99bp。此外,除P5外的启动子缺失片段以及全长片段注射的烟草叶片上的大量表皮毛被上了较深的蓝色。
图6CsMIXTA启动子全长及缺失片段作用下的烟草GUS染色
CK:阴性对照;P:全长启动子;P1~P5:启动子缺失片段1~5。
2.5CsMIXTA核心启动子作用下烟草瞬时表达分析
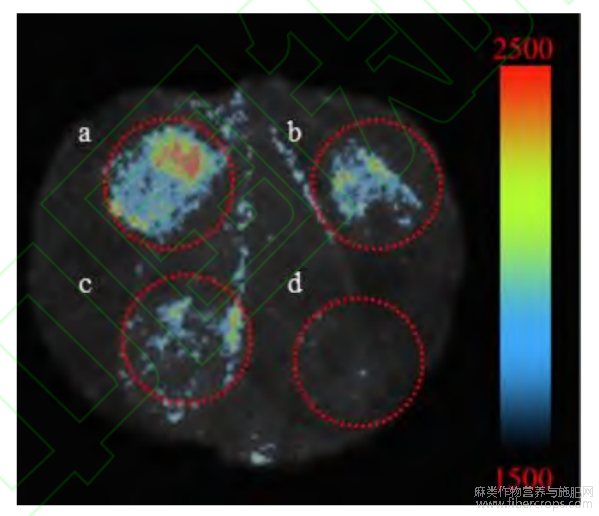
为了验证CsMIXTA启动子核心启动区的表达活性,将核心启动区P4(393bp)片段通过NC克隆到LUC表达质粒中,瞬时转化到本氏烟草中,2d后取样进行荧光素酶检测。由图7可知,CsMIXTA的核心启动子区具备转录活性。
图7CsMIXTA启动子核心区域P4作用下的LUC荧光表达
a~c:P4-LUC注射区域;d:pNC-Green-LUC空载注射区域(阴性对照)。
2.6 CsMIXTA全长启动子作用下工业大麻糖叶瞬时表达分析
通过农杆菌介导的瞬时转化方法,CsMIXTA启动子驱动GUS基因在工业大麻糖叶中的表达如图8所示,在糖叶中的不具备分泌能力的单细胞表皮毛均未被染上色,而大量头状无柄腺毛被染成蓝色;部分头状有柄腺毛的腺毛柄被染成蓝色。
图8CsMIXTA全长启动子作用下的工业大麻GUS染色
a:单细胞表皮毛;b:头状无柄腺毛;c:头状有柄腺毛。
2.7不同胁迫处理下工业大麻糖叶中CsMIXTA表达分析
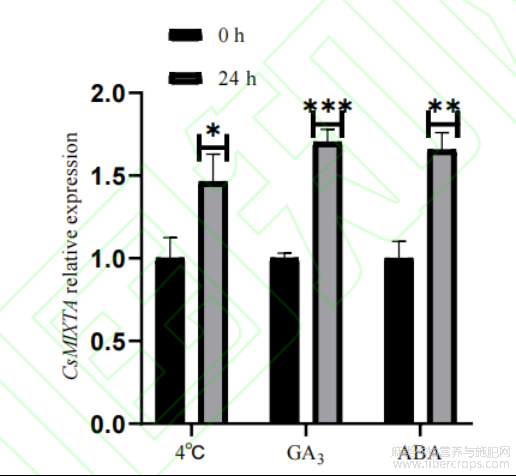
分析发现CsMIXTA启动子区含有低温诱导、ABA和GA响应元件,本研究利用实时定量PCR检测4℃、ABA和GA3处理下CsMIXTA在工业大麻糖叶中的表达量。由图9可知,4℃处理24h,CsMIXTA的表达量较正常培养条件下表达水平上调46%;ABA处理下CsMIXTA基因的表达量上调70%;GA3处理下CsMIXTA基因的表达量上调65%。说明,低温、GA3及ABA均能增强启动子的活性。
图9 3种胁迫处理对CsMIXTA表达模式的影响
*、**、***分别表示在0.05、0.01和0.001水平上差异显著。
3讨论
目前工业大麻腺毛分子机制研究较少,只有3个相关的转录因子被报道。bHLH转录因子CsMYC4使番茄腺毛密度增加,且其受茉莉酸甲酯正向调控[14]。HD-ZIP转录因子CsHDG5可能正向调控腺毛发育[15]。本研究中R2R3-MYB转录因子CsMIXTA在烟草中过表达提高了烟草腺毛密度[12],因此CsMIXTA启动子研究具有重要意义。
启动子具有多种顺式作用元件,对基因的表达调控具有重要的作用,能够影响基因的表达水平和空间特异性,有助于研究基因的表达调控与组织表达特异性[16-18]。本研究克隆了CsMIXTA基因上游的2199bp启动子序列,预测分析其顺式作用元件,发现该启动子序列除了具有启动子基本元件TATA-box和CAAT-box外,还存在低温响应元件、光响应元件、脱落酸响应元件、昼夜节律控制响应元件、赤霉素响应元件等顺式作用元件,这与拟南芥、玉米等MYB基因家族启动子顺式作用元件的预测结果相似[19,20]。我们的研究表明CsMIXTA基因可能受到多个因素的诱导表达。
启动子的核心区域在基因表达水平上起到关键的调控作用,启动子缺失法常用于研究启动子的核心区域[21]。为了探索CsMIXTA基因启动子的核心区域,本研究将启动子全长和5个5′端缺失片段融合进了具有GUS基因的植物表达载体并瞬时转化烟草,对各个片段转化的烟草进行GUS染色,发现只有99bp的缺失片段P5没有被染色,不具备启动子活性。因此我们推测CsMIXTA基因启动子的核心区域位于P4与P5(-393~-99bp)之间,此外本研究还将P4片段连接了LUC基因在烟草中进行表达,验证发现该核心启动区具有转录活性。依据此核心区域我们了结合Softberry预测的转录起始位点,确定CsMIXTA基因的启动子转录起始位点为鸟嘌呤(G),位于ATG上游196bp处。除此之外,在除P5外各个片段侵染的烟草叶片的表皮毛都被染上了较深的蓝色,表明CsMIXTA启动子具有组织特异性,可能控制着表皮毛的发育。
在植物腺毛中发挥功能的R2R3MYB家族基因,其启动子大部分能在腺毛中特异表达。青蒿中能够促进腺毛发育和青蒿素合成的AaMIXTA1基因的启动子在腺毛中的表达比在其它区域强[9]。棉花中与表皮毛发育相关的基因GaMYB2的启动子在棉花、拟南芥、烟草的腺毛中都具有特异表达[22]。前人研究发现CsMIXTA可能调控大麻雌性花器官中腺毛的发生和发育[12]。为验证CsMIXTA启动子在工业大麻的组织表达模式,本研究将CsMIXTA启动子全长融合进了具有GUS基因的植物表达载体并侵染工业大麻糖叶。结果显示在工业大麻糖叶中没有分泌能力的单细胞表皮毛都没有被染成蓝色,叶表面、头状无柄腺毛和部分头状有柄腺毛的腺毛柄被染成了蓝色,且头状无柄腺毛被染得更深。由于在大麻中头状有柄腺毛是从头状无柄腺毛发育而来的[23],因此CsMIXTA可能在工业大麻腺毛的生长发育中起到重要得作用。
在植物表皮毛发育过程中受到GA等植物激素的调控。GA主要是通过诱导DELLA降解,协同促进毛状体的形成,如拟南芥[24]、棉花[25]。而R2R3-MYB中的部分转录因子可以调控赤霉素的含量,如青蒿中AaMYB1和AtMYB61通过积极影响酶的表达,将GA9转化为生物活性的GA4[9],菊花中CmMYB2通过与CmBBX24的相互作用改变细胞GA含量[26]。本研究中使用GA3对工业大麻进行胁迫处理,CsMIXTA基因显著上调,表明该基因对赤霉素能够产生应答,可能间接调控表皮毛发育。
研究表明低温胁迫下番茄SlMYB96表达量升高,揭示了SlMYB96能够响应低温胁迫,将其沉默后番茄植株抗寒性降低。青海茄参MYB转录因子McMYB-86在低温处理的24h内显著上调表达[27]。水稻OsMYBR1的启动子区域含有ABRE,并且OsMYBR1过表达水稻对ABA的抗性更强,揭示了OsMYBR1过表达水稻通过ABA介导的途径上调胁迫响应基因增强耐旱性[28]。本研究4℃、ABA对工业大麻进行胁迫处理,并进行qRT-PCR分析,结果显示CsMIXTA基因的相对表达量在这2个处理条件下都显著上调,表明CsMIXTA基因能对低温与ABA处理产生应答。
4结论
本研究克隆了工业大麻CsMIXTA基因启动子,利用生物信息学的方法预测了其可能存在的顺式作用元件;通过GUS染色找到了CsMIXTA启动子的核心区域位于-393~-99bp;通过启动子瞬时转化工业大麻糖叶,发现该启动子在腺毛中特异表达;对工业大麻糖叶进行非生物胁迫处理,发现低温、GA3及ABA均增强启动子的活性,这为深入探究CsMIXTA基因的功能及调控机制提供了理论和试验依据。
References
[1] 李秋实, 孟莹, 陈士林. 药用大麻种质资源分类与研究策略. 中国中药杂志, 2019, 44: 4309–4316.
Li Q S, Meng Y, Chen S L. A new Cannabis germplasm classification system and research strategies of non-psychoactive medicinal cannabis. China J Chin MaterMed, 2019, 44: 4309–4316 (in Chinese with English abstract).
[2] Goncalves J, Rosado T, Soares S, SimaoAY, Caramelo D, LuisA, Fernandez N, Barroso M, Gallardo E, Duarte A P. Cannabis and its secondary metabolites: their use as therapeutic drugs, toxicological aspects, and analytical determination. Medicines (Basel), 2019, 6: 31.
[3] Tanney CA S, BackerR, GeitmannA, Smith D L L. Cannabis glandular trichomes: a cellular metabolite factory. Front Plant Sci, 2021, 12: 1923.
[4] BackerR, Schwinghamer T, Rosenbaum P, McCarty V, Bilodeau S E, LyuD, Ahmed M B, Robinson G, Lefsrud M, Wilkins O, Smith D L. Closing the yield gap for cannabis: a meta-analysis of factors determining cannabis yield. Front Plant Sci, 2019, 10: 495.
[5] Burgel L, Hartung J, Schibano D, Graeff-Hoenninger S. Impact of different phytohormones on morphology, yield and cannabinoid content of Cannabis sativa L. Plants (Basel), 2020, 9: 725.
[6] Chezem W R, Clay N K. Regulation of plant secondary metabolism and associated specialized cell development by MYBs and bHLHs.Phytochemistry, 2016, 131: 26–43.
[7] Brockington S F, Alvarez-Fernandez R, Landis J B, Alcorn K, Walker R H, Thomas M M, Hileman L C, Glover B J. Evolutionary analysis of the MIXTA gene family highlights potential targets for the study of cellular differentiation. Mol BiolEvol, 2013, 30: 526–540.
[8] Matias-Hernandez L, Jiang W, Yang K, Tang K, Brodelius P E, Pelaz S. AaMYB1 and its orthologue AtMYB61 affect terpene metabolism and trichome development in Artemisia annua and Arabidopsis thaliana. Plant J, 2017, 90: 520–534.
[9] ShiP, Fu X, Shen Q, Liu M, Tang K. The roles of AaMIXTA1 in regulating the initiation of glandular trichomes and cuticle biosynthesis in Artemisia annua. New Phytol, 2017, 217: 261–276.
[10] Ewas M, Gao Y, Wang S, Liu X, Zhang H, Nishawy E M E, Ali F, ShahzadR, ZiafK, Subthain H. Manipulation of SlMXl for enhanced carotenoids accumulation and drought resistance in tomato. Sci Bull, 2016, 61: 1413–1418.
[11] Chalvin C, Drevensek S, Dron M, Bendahmane A, Boualem A. Genetic control of glandular trichome development. Trends Plant Sci, 2020, 25:477–487.
[12] Haiden S R, Apicella P V, Ma Y, Berkowitz G A. Overexpression of CsMIXTA, a transcription factor from Cannabis sativa, increases glandular trichome density in tobacco leaves. Plants (Basel), 2022, 11: 1519.
[13] Alter H, Peer R, DombrovskyA, Flaishman M, Spitzer-Rimon B. Tobacco rattle virus as a tool for rapid reverse genetics screens and analysis of gene function in Cannabis sativa L. Plants (Basel), 2022, 11: 327.
[14] Huang X, Chen W, Zhao Y, Chen J, Ouyang Y, Li M, Gu Y, Wu Q, Cai S, Guo F, Zhu P, Ao D, You S, Vasseur L, Liu Y. Deep learning-based quantification and transcriptomic profiling reveal a methyl jasmonate-mediated glandular trichome formation pathway in Cannabis sativa. Plant J,2024.
[15] Ma G, Zelman AK, Apicella PV, Berkowitz G. Genome-wide identification and expression analysis of homeodomain leucine zipper subfamily IV(HD-ZIP IV) gene family in Cannabis sativa L. Plants (Basel), 2022, 11: 1307.
[16] 张春晓, 王文棋, 蒋湘宁, 陈雪梅. 植物基因启动子研究进展. 遗传学报, 2004, 31: 1455–1464.
Zhang CX, Wang WQ, Jiang XN, Chen XM. Review on plant gene promoters. J Genet Genomics, 2004, 31: 1455–1464 (in Chinese with English abstract).
[17] 马倩, 马宝月, 穆波, 马慧. 植物基因启动子的克隆及分析的研究进展. 中国农业文摘-农业工程, 2018, 30(3): 23–29.
Ma Q, Ma B Y, Mu B, Ma H. Research progress on cloning and analysis of plant gene promoter. Agric Sci Eng China, 2018, 30(3): 23–29 (in Chinese with English abstract).
[18] 毛燕, 郑名敏, 牟成香, 谢吴兵, 唐琦. 渗透胁迫下玉米自然反义转录本cis-NATZmNAC48启动子的功能分析. 作物学报, 2024, 50: 354–362.
Mao Y, Zheng M M, Mou C X, Xie W B, Tang Q. Function analysis of the promoter of natural antisense transcript cis-NATZmNAC48 in maize under osmotic stress. Acta Agron Sin, 2024, 50: 354–362 (in Chinese with English abstract).
[19] Ambawat S, Sharma P, YadavN R, Yadav R C J P. MYB transcription factor genes as regulators for plant responses: an overview. PhysiolMol Biol Plants, 2013, 19: 307–321.
[20] Du H, Feng B R, Yang S S, Huang YB, TangY X. The R2R3-MYB transcription factor gene family in maize. PLoS One, 2012, 7: e37463.
[21] 周悦, 赵志华, 张宏宁, 孔佑宾. 大豆紫色酸性磷酸酶基因GmPAP14启动子克隆与功能分析. 作物学报, 2022, 48: 590–596.
Zhou Y, Zhao Z H, Zhang HN, Kong YB. Cloning and functional analysis of the promoter of purple acid phosphatase gene GmPAP14 in soybean. ActaAgron Sin, 2022, 48: 590–596 (in Chinese with English abstract).
[22] Shangguan X X, Xu B, Yu Z X, Wang L J, Chen XY. Promoter of a cotton fibre MYB gene functional in trichomes of Arabidopsis and glandular trichomes of tobacco. J Exp Bot, 2008, 59: 3533–3542.
[23] Livingston S J, Quilichini T D, Booth J K, Wong D C J, Rensing K H, Laflamme-Yonkman J, Castellarin S D, Bohlmann J, Page J E, Samuels A L. Cannabis glandular trichomes alter morphology and metabolite content during flower maturation. Plant J, 2020, 101: 37–56.
[24] Maes L, Inze? D, Goossens A. Functional specialization of the TRANSPARENT TESTA GLABRA1 network allows differential hormonal control of laminal and marginal trichome initiation in Arabidopsis rosette leaves. Plant Physiol, 2008, 148: 1453–1464.
[25] Xia X C, Hu Q Q, Li W, Chen Y, Han LH, Tao M, Wu WY, LiX B, Huang GQ. Cotton (Gossypium hirsutum) AZ3 and SLR1 function in jasmonate and gibberellin mediated epidermal cell differentiation and elongation. Plant Cell Tissue Organ Cult, 2018, 133: 249–262.
[26] Zhu L, GuanY, Liu Y, Zhang Z, Jaffar MA, Song A, Chen S, Jiang J, Chen F. Regulation of flowering time in chrysanthemum by the R2R3 MYB transcription factor CmMYB2 is associated with changes in gibberellin metabolism. HorticRes, 2020, 7: 96.
[27] 赵悦, 赵艳艳, 李强锋, 段红俊. 青海茄参MYB转录因子家族生物信息学及应答低温胁迫分析. 分子植物育种, 网络首发[2023-12-06],https://link.cnki.net/urlid/46.1068.s.20231205.1916.027.
Zhao Y, Zhao Y Y, Li Q F, Duan H J. Bioinformatics analysis of MYB transcription factor family in mandragora chinghaiensis under low temperature induction. Mol Plant Breed, Published online [2023-12-06], https://link.cnki.net/urlid/46.1068.s.20231205.1916.027 (in Chinese with English abstract).
[28] Yin X, Cui Y, Wang M, Xia X. Overexpression of a novel MYB-related transcription factor, OsMYBR1, confers improved drought tolerance and decreased ABA sensitivity in rice. Biochem Biophys Res Commun, 2017, 490: 1355–1361.
文章摘自:周智满,张小雨,高峰,等.工业大麻腺毛发育基因CsMIXTA启动子克隆及功能分析[J/OL].作物学报,1-13[2024-08-19].http://kns.cnki.net/kcms/detail/11.1809.S.20240709.1425.004.html.