摘 要:剑麻是热带地区重要的纤维作物,生产中易受到寒害威胁而造成减产。超氧化物歧化酶(SOD)是一种重要的保护酶,在植物应答低温等逆境条件胁迫过程中起重要作用。前人研究已初步明确超氧化物歧化酶在剑麻低温处理后起重要作用,但其在剑麻中的基因序列及分子调控机制尚未明确。在已发表的剑麻转录组数据库中鉴定出5个SOD基因,遗传进化分析显示其在不同植物物种中进化较保守。实时定量PCR结果显示低温胁迫后5个基因表达模式均呈先上升后下降趋势,且均与SOD活性变化显著正相关(P<0.05)。其中2个CSD基因和1个FSD基因与SOD活性极显著正相关(P<0.01),表明这3个基因在应答低温胁迫过程中起重要作用。研究鉴定了剑麻SOD基因并初步明确其应答低温胁迫表达模式,为深入开展剑麻抗寒机制研究提供了重要基础。
关键词:剑麻;超氧化物歧化酶基因;低温胁迫;基因表达
剑麻是热带亚热带地区广泛种植的硬质纤维作物,在我国主要种植于广西、广东、海南等地[1]。剑麻纤维具有强度高、质地坚韧、耐腐蚀等特性,在航海、渔业、造纸等行业用途广泛[2]。此外,剑麻纤维复合材料的高强度、耐腐蚀等特性在环保再生型建筑材料方面存在巨大的应用前景[3]。近年来随着全球气候的变化,极端温度(高温和低温)出现频次日益增多,已成为制约全球农业进一步发展的重要因素[4]。低温作为一种非生物胁迫,可造成植物组织冻伤,引起芽萎缩、叶片发黄萎蔫、组织死亡等寒害症状[5]。剑麻作为典型的热带作物之一,对低温寒害耐受力较差。我国大部分剑麻种植区处于热带亚热带地区,在早春易受“倒春寒”影响,严重时可造成大面积减产[6]。目前拟南芥、水稻等模式植物中已开展了大量的低温胁迫调控机制相关研究,而剑麻相关研究报道较少。张学财等[7]测定了低温胁迫后剑麻叶片中保护酶等生理指标的变化规律,黄显雅等和田夏红等分别开展了剑麻种质资源抗寒性田间调查[4,8]。杨欣丽开展了剑麻应答低温胁迫机制研究,明确了8种生理生化指标及CBF、psaA和psaB等基因的变化规律[6]。超氧化物歧化酶(Superoxide Dismutase, SOD)作为一种重要的保护酶,可有效清除植物体内过量的活性氧,在植物生长发育、逆境胁迫应答等过程中起重要作用[9]。剑麻中已初步明确低温胁迫后SOD活性变化,但相关基因及表达模式尚未报道[6,7]。本研究根据已发表的剑麻转录组,通过同源序列比对鉴定SOD基因,并开展系统进化及表达分析,阐明剑麻SOD基因的遗传进化规律及其应答低温胁迫表达模式,为探明其生物学功能及作用机制提供基础。
1 材料与方法
1.1 材料种植与处理
本研究选取的试验材料为剑麻主栽品种‘H11648’株芽苗,在中国热带农业科学院环境与植物保护研究所(19.99°N,110.33°E)盆栽种植2个月后放置于智能人工气候培养箱(DRXM-150-2,上海川昱)进行低温处理。6°C处理0h、6h、12h、18h、24h时剪取叶片取样[6],每个处理均在不同植株重复取样3次,随后置于液氮速冻,放置于-80°C超低温冰箱保存备用。
1.2 材料种植与处理
以7个拟南芥和7个水稻SOD基因为参考序列,通过TBlastx方法在剑麻转录组数据库检索SOD基因同源序列[10-13]。通过ORF-FINDER软件分析检索获得的剑麻基因序列,将包含完整编码序列的SOD基因进行后续分析[14]。通过ProtParam软件在线预测剑麻SOD蛋白长度、分子量和理论等电点等理化性质[15]。选取拟南芥(7个)、水稻(7个)、芦笋(9个)和剑麻(5个)SOD蛋白序列在ClustalX2.0软件中进行多重序列比对,进而构建Neighbor-Joining系统进化树,Bootstrap分析重复1000次[16]。
1.3 剑麻SOD基因表达分析及SOD活性测定
采用天根植物总RNA提取试剂盒(天根生化科技,中国)提取样品中的总RNA,并通过Go Script Reverse Transcription System(Promega,美国)反转录为cDNA备用。通过QuantStudio6实时荧光定量PCR系统(赛默飞,美国)进行实时定量PCR分析。PCR反应程序为94℃反应30s;94℃反应3min,94℃反应10s,58℃反应30s,共重复40个循环;溶解曲线循环。PCR扩增体系最终体积为20μL,其中包含1μLc DNA模板、10μL Trans Start Tip Green qPCR Super mix(全式金,中国)、0.4μL Passive Reference Dye(全式金,中国)、0.5μL正向引物、0.5μL反向引物和7.6μ双蒸水。根据5个剑麻SOD基因及内参基因PP2A(proteinphosphatase2A)的编码序列,在Primer3软件中设计定量PCR引物,其序列见表1[17]。每个样品重复3次作为技术重复,通过2-ΔΔCt法计算每个基因的相对表达量[18]。采用羟胺法测定剑麻样品中SOD活性[6]。
2 结果与分析
2.1 剑麻SOD基因的鉴定
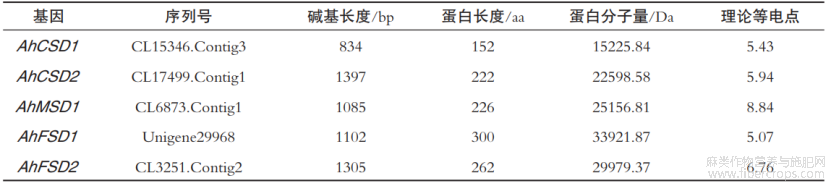
在剑麻转录组中进行同源序列检索共获得5个含完整编码序列的SOD基因(表2),将其命名为AhCSD1、AhCSD2、AhMSD1、AhFSD1和AhFSD2,其序列长度为834~1397bp,编码的蛋白质长度为152~300aa。蛋白质分子量为15225.84~33921.87Da,理论等电点为5.07~8.84。
表1 实时定量PCR引物
表2 剑麻SOD基因及其蛋白理化性质
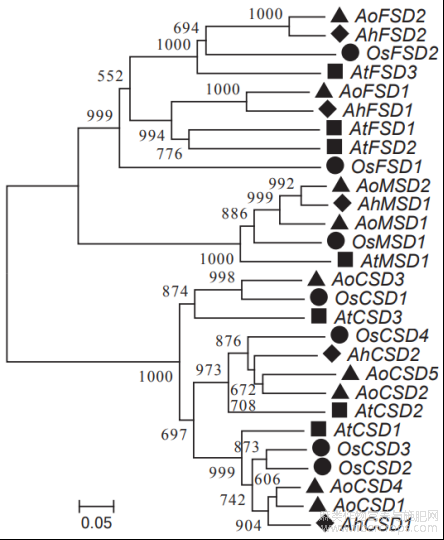
图1 拟南芥、水稻、芦笋及剑麻SOD基因遗传进化分析
注:At表示拟南芥;Ah表示剑麻;Ao表示芦笋;Os表示水稻
2.2 剑麻SOD基因系统进化分析
以拟南芥、水稻SOD基因为参照,选取天门冬目作物芦笋、剑麻SOD基因进行遗传进化分析。利用ClustalX2.0软件进行氨基酸序列的多重比对并构建Neighbor-Joining进化树(Bootstrap分析采用1000次重复),结果如图1所示。28个蛋白序列被分成3个分支,即FSD、MSD和CSD分支。FSD和MSD分支中各物种序列数相近,拟南芥和芦笋序列数分别在两分支多于其他物种。CSD分支中水稻和芦笋序列数多于拟南芥和剑麻,其中CSD3亚分支中未聚类到剑麻序列。此外,剑麻和芦笋序列均聚类在相同分支。
2.3 剑麻SOD基因应答低温胁迫表达分析
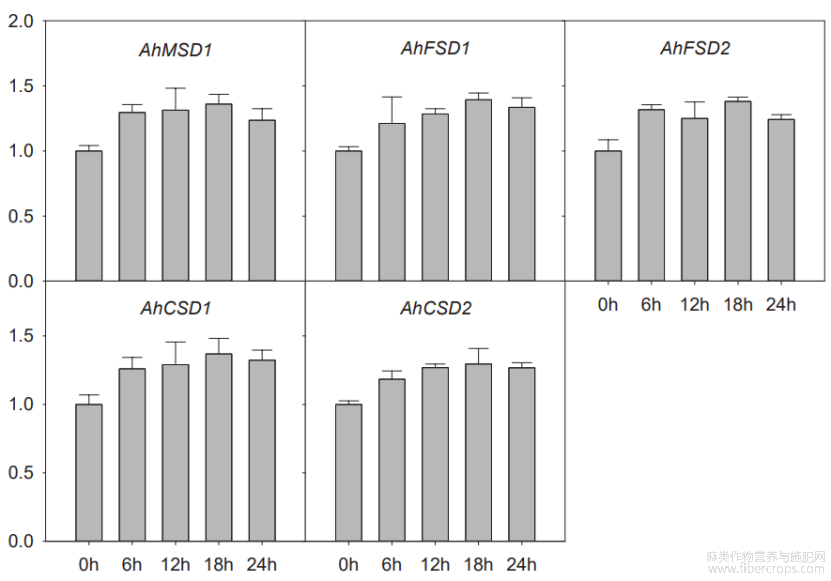
通过实时定量PCR检测低温胁迫后剑麻SOD基因表达模式,结果显示5个基因表达水平均呈先上升后下降趋势,但未达到显著水平(图2)。其中AhFSD2表达水平在处理后12h和24h时分别较6h和18h时略有下降,其余4个基因表达水平仅在处理后24h略有下降。
2.4 剑麻低温胁迫SOD活性与基因表达相关性分析
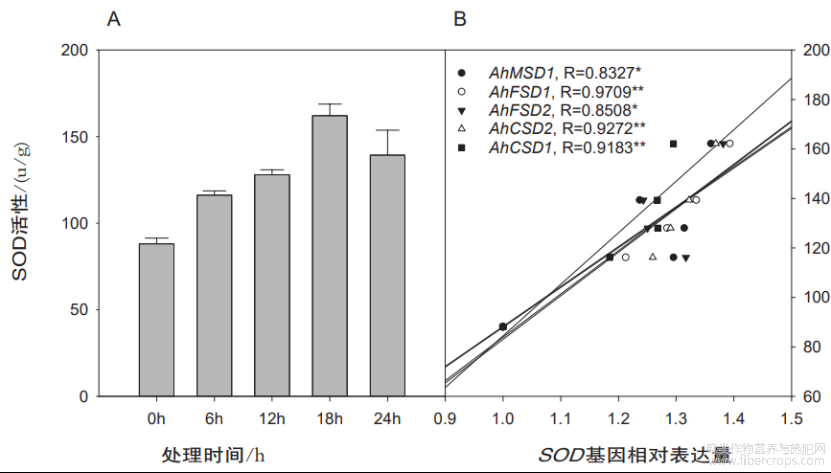
进一步测定了低温胁迫后剑麻叶片中SOD活性,结果显示其呈先上升后下降趋势,处理后18h时SOD活性达到峰值后下降(图3A)。将SOD活性与5个基因的表达模式进行相关性分析(图3B),结果显示AhFSD1、AhFSD2和AhCSD1表达模式与SOD活性呈极显著正相关(P<0.01),其余2个基因呈显著正相关(P<0.05)。
图2 剑麻SOD基因低温胁迫表达模式
A:样品叶片中SOD活性图 B:SOD与基因表达相关性分析图
图3 剑麻SOD基因低温胁迫表达模式
注:B中R为相关系数,*和**分别表示显著(P<0.05)和极显著(P<0.01)
3 讨论
SOD在植物抗氧化防御系统中起重要作用,可将超氧阴离子自由基歧化为H2O2和O2以清除活性氧[19]。剑麻中已初步明确SOD在应答低温胁迫过程中起重要作用,但其蛋白类型及基因序列尚未明确[6]。植物中主要存在3种类型SOD,即CSD(Cu/Zn-SOD)、MSD(Mn-SOD)和FSD(Fe-SOD),高等植物中以CSD为主[20]。根据同源序列检索发现剑麻中存在上述3种类型SOD基因,在不同植物物种中进化较保守,与模式植物相比CSD基因数量较少,可能与SOD基因的组织特异性表达有关[11]。目前剑麻基因组序列尚未公布,叶片转录组数据中可能并未完全反映基因组水平SOD基因家族的表达模式。实时定量PCR结果显示低温处理后5个剑麻SOD基因表达水平均上升,在24h时开始下降,与SOD活性变化规律一致,且均呈显著正相关,表明5个基因均参与应答低温胁迫。其中2个CSD基因和1个FSD基因与SOD活性极显著正相关,表明这3个基因在应答低温胁迫过程中起重要作用。后续将进一步探究剑麻CSD基因应答低温胁迫机制,提升对剑麻抗寒机制的理解,对选育抗寒剑麻新品种具有重要意义。
[1] 陈莉莎.我国剑麻种质资源纤维强力性能研究[J].中国麻业科学,2023,45(1):33-40,48.
[2] 黄兴,习金根,陈涛,等.剑麻苯丙氨酸裂解酶基因的鉴定及表达分析[J].作物学报,2021,47(6):1082-1089.
[3] 杨芳,于淑娟,朱永飞,等.剑麻纤维/聚合物复合材料研究进展[J].化工技术与开发,2015,44(4):28-31.
[4] 田夏红,刘青,刘建荣,等.剑麻种质资源表型多样性分析与抗寒性调查[J].中国麻业科学,2020,42(3):97-103.
[5] Guo X, Liu D, Chong K. Cold signaling in plants: insights into mechanisms and regulation [J]. J Integr Plant Biol,2018, 60:745-756.
[6] 杨欣丽.剑麻应答低温胁迫机制及抗寒性比较研究[D].海口:海南大学,2023.
[7] 张学财,周文钊,李俊峰.低温胁迫下4个剑麻品种的SOD、POD、CAT变化[J].中国热带农业,2009(4):47-50.
[8] 黄显雅,陈涛,金刚,等.2017年南宁龙舌兰种质寒害调查及养护[J].热带农业科学,2018,38(9):65-68,90.
[9] 杨林林,韩敏琦,高嘉,等.小麦超氧化物歧化酶基因家族鉴定及盐胁迫下响应锌钾的表达分析[J].山东农业科学, 2023,55(8):11-20.
[10] Altschul SF, Gish W, Miller W, et al. Basic local alignment search tool[J]. J Mol Biol,1990,215:403-410.
[11] Huang X, Xiao M, Xi J, et al. De novo transcriptome assembly of Agave H11648 by Illumina sequencing and identification of cellulose synthase genes in Agave species[J]. Genes,2019,10:103.
[12] Kaminaka H, Morita S, Tokumoto M, et al. Differential gene expressions of rice superoxide dismutase isoforms to oxidative and environmental stresses[J]. Free Radic Res,1999,31:219-225.
[13] Zhang G, Ding Q, Wei B. Genome-wide identification of superoxide dismutase gene families and their expression patterns under low-temperature, salt and osmotic stresses in watermelon and melon[J]. 3 Biotech,2021,11(4):194.
[14] Rombel IT, Sykes KF, Rayner S, et al. ORF-FINDER: A vector for high-throughput gene identification[J]. Gene,2002,282:33-41.
[15] Wilkins MR, Gasteiger E, Bairoch A, et al. Protein identification and analysis tools in the ExPASy server[J]. Methods Mol.Biol.,1999,112:531-552.
[16] Larkin MA, Blackshields G, Brown NP, et al. Clustal W and Clustal X version 2.0[J]. Bioinformatics,2007,23:2947-2948.
[17] Untergasser A, Cutcutache I, Koressaar T, et al. Primer 3- new capabilities and interfaces[J]. Nucleic Acids Res,2012, 40:115.
[18] Livak KJ, Schmittgen TD. Analysis of relative gene expression data using real-time quantitative PCR and the 2(-Delta Delta C(T)) method[J]. Methods,2001,25: 402-408.
[19] 魏婧,徐畅,李可欣,等.超氧化物歧化酶的研究进展与植物抗逆性[J].植物生理学报,2020,56(12):2571-2584.
[20] Zang Y, Chen J, Li R, et al. Genome-wide analysis of the superoxide dismutase (SOD) gene family in Zostera marina and expression profile analysis under temperature stress[J]. PeerJ,2020,8:9063.
文章摘自: 陈莉莎,李玉波,胡晓丽,杨欣丽,易克贤,黄兴. 剑麻超氧化物歧化酶基因鉴定及低温胁迫表达分析[J]. 中国热带农业,2024,116(01):23-27.