摘 要:工业大麻是重要的纤维作物,其纤维具有多种独特优势和广泛应用前景,WOX基因家族在调控工业大麻纤维发育与植株生长抗逆过程中具有潜在作用。目前,工业大麻中CsWOX基因家族的功能及其在稳产、增产等关键生物学过程中的作用尚未明晰。因此,本研究旨在系统鉴定工业大麻CsWOX基因家族成员,揭示其在生长发育和干旱胁迫响应中的分子功能,初步阐明钩状木霉通过调控WOX基因网络提升植株抗逆性的分子机制。结果表明:(1)从工业大麻中鉴定出11个CsWOX基因家族成员,编码氨基酸长度为202~435 aa;相对分子量为25 398.3~48 429.45 Da,该基因家族蛋白多数呈酸性且多数定位于细胞核。(2)CsWOX蛋白成员可分为3个分支(古代支、中间支、现代支),现代支有含有6个亚支,中间支含有2个亚支,古代支只有1个亚支,同一亚支的WOX转录因子保守基序种类及组织形式存在一定保守性,每个家族成员均含有1个螺旋-环-螺旋-转角-螺旋结构的保守结构域。(3)启动子预测分析发现光相关响应、赤霉素响应、脱落酸响应相关的顺式元件数量最多,其他元件如生长素响应、水杨酸响应、厌氧响应、昼夜节律响应、低温和防御响应等顺式元件不均匀分布于启动子中。(4)CsWOX4、CsWOX13a/b基因在根、茎、叶、花等组织均有较高的表达量;并且与对照组相比,CsWOX4和CsWOX13a/b在干旱处理、钩状木霉处理及干旱和钩状木霉共处理的表达量发生变化,推测其可能在工业大麻生长发育以及植株抗逆过程中发挥潜在作用。该研究为解析CsWOX基因家族在工业大麻生长发育及抗逆中的功能提供了重要线索。
关键词:工业大麻,WOX基因家族,生物信息学分析
植物特异性WOX(WUSCHEL-related homeobox,WOX)基因家族编码的转录因子是调控植物发育的关键因子(Van et al.,2009),其成员均具有保守的同源盒结构域(Homeobox,HB),该结构域通过特异性DNA结合调控下游基因表达(Gehring et al.,1990;Gehring et al.,1994)。根据进化关系可将WOX基因家族被划分为现代支、中间支和古老支三个分支(Wu et al.,2019)。在拟南芥中已经鉴定出15个WOX基因家族成员,分别是AtWUS和AtWOX1-AtWOX14。每个WOX基因在植物体内具有不同的功能(Zhang et al.,2019)。例如,AtWUS调控着植物芽干细胞的稳态(Xu,2019)。AtWOX2在拟南芥中调控着子叶边界的建成(Lie et al.,2012),而AtWOX3参与着花生萼片和雄蕊的发育(Nardmann et al.,2004)。AtWOX4则通过信号转导调控着植物形成层干细胞的增殖(Hirakawa et al.,2010)。另外,AtWOX5对根分生组织附近的静止中心的建立起着至关重要的作用(Gonzali et al.,2005)。AtWOX9和AtWOX8通过移动受精卵中细胞器位置的来建立受精卵极性,从而实现不对称细胞分裂,在植物胚胎发育和受精卵极性建立中发挥着关键的作用(Ueda et al.,2011),同时AtWOX9还能维持着分生组织的生长(Wu et al.,2005)。然而,Romera等(2013)采用反向遗传学方法发现了AtWOX13基因通过负向调控中央区JAG/FIL基因的表达来调控拟南芥果实果实模式建成。综上,WOX基因家族在植物生长发育中扮演重要的角色。
工业大麻(Cannabis sativa)作为一种多用途植物经济植株,其花可用于提取次生代谢物,种子可以用于榨取油脂,其韧皮纤维具有韧皮纤维因具备高强度、耐腐蚀等特性,已广泛应用于纺织品和建筑材料领域(Andre et al.,2016)。在建筑材料领域,工业大麻纤维与石灰复合形成的生物基建筑材料(Hempcrete)因其轻质、隔热和吸湿特性,已成为可持续建筑的研究热点(Shahzad,2012)。近年来,随着基因组学技术的快速发展,工业大麻的基因组研究取得了显著进展。2011年,van Bakel等(2011)首次发布了工业大麻的基因组草图,为后续的功能基因组学研究奠定了基础。然而,近年来随着极端气候频繁发生致使工业大麻减产,其中其中干旱胁迫被确认为主要限制因子(Amaducci et al.,2008;?ydelis et al.,2022),而研究表明合理施用植物促生微生物会增强植物抗旱性并提高植株产量(Stamenkovi?et al.,2018),钩状木霉作为一种促生微生物,能增强多酚氧化酶(PPO)、超氧化物歧化酶(SOD)等保护性清除酶的活性,降低过氧化氢(H2O2)等非酶类胁迫标志物的水平,从而增强植株整体抗性(Abdelkhalek et al.,2022)。Bae等(2009)研究发现,钩状木霉(Trichoderma hamatum)通过分泌吲哚乙酸(IAA),显著促进可可幼苗根系生物量和侧根数量的增加,并能延迟植物对干旱胁迫的响应;还有研究表明,促生微生物增加植株产量及抗性的主要机理是通过合成吲哚类似物诱导WOX5基因的表达,调控细胞周期(K?pczyńska&Karczyński,2020),然而,钩状木霉通过WOX基因家族调控工业大麻生长发育与抗旱性的分子互作机制尚未解析,阐明其生长发育与抗逆响应的作用模式,将为突破纤维品质与抗逆性协同提升的育种瓶颈提供关键基因编辑靶点。
目前,WOX基因家族在烟草,毛竹、生菜等植物中已有报道(李晨曦等,2016;李晓旭等,2021;罗康胜等,2024),但工业大麻相关研究仍属空白。WOX基因家族通过调控植物生长发育和组织分化,可能直接影响生物量积累(Zhou et al.,2024),因此,研究WOX基因家族有助于改良大麻的生物质生产,提高经济效益。例如,在棉花中,WOX4基因通过调控纤维细胞的伸长和分化,影响纤维的长度和强度(Sajjad et al.,2021),类似地,在工业大麻中,WOX基因也可能通过调控纤维细胞的发育,影响其纤维质量和产量。此外,工业大麻在生长过程中可能遭遇多种逆境胁迫,WOX基因在植物逆境胁迫过程中的作用也逐渐受到关注。例如,干旱胁迫可以上调或下调大豆中某些WOX基因的转录丰度(Hao et al.,2019),水稻中OsWOX11通过控制根系生长来提高水稻的耐旱性(Cheng et al.,2016),马尾松中的PmWOX1基因被ABA、MeJA、H2O2和干旱胁迫显著诱导表达,并对植物抵御逆境有一定影响(Wang et al.,2024)。因此,研究工业大麻WOX基因家族的功能,还可为培育高产抗逆品种开发提供新的思路。以期为我国工业大麻的发展及育种提供理论依据。
本研究以工业大麻为研究材料,利用生物信息学技术手段对工业大麻CsWOX基因家族开展其理化性质、保守结构域以及表达模式分析,以探讨以下问题:(1)工业大麻中CsWOX基因家族成员数量及理化性质;(2)对CsWOX基因家族成员保守结构域、系统进化树、保守基序、顺式作用元件等进行分析;(3)工业大麻CsWOX基因家族成员在不同部位及干旱胁迫和钩状木霉处理下的表达变化情况。本研究旨在筛选工业大麻的WOX基因,探究其协同提升纤维产量与植株抗性的分子机制,为多性状协同育种提供靶点,对于进一步解析工业大麻中WOX基因的功能及其机制具有一定参考意义。
1 材料与方法
1.1 转录因子鉴定和理化性质分析
从大麻数据库中(http://gdb.supercann.net/)下载大麻基因组的相关数据(品种为Finola);从拟南芥TAIR网站上下载拟南芥WOX蛋白序列,以e<10-5为阈值进行同源比对进行初步筛选,利用Pfam数据库中下载中下载隐马科夫模型(PF00046),用HMMER程序进一步剔除不含PF00046结构域的序列,筛选工业大麻CsWOX基因家族成员。将工业大麻CsWOX蛋白序列提交到在线工具ProtParam(Expasy-ProtParam tool)和wolfpsort(WoLF PSORT:Protein Subcellular Localization Prediction(hgc.jp)),预测氨基酸数量、分子量、等电点和亚细胞定位。
1.2 构建系统发育树、多序列比对和共线性分析及蛋白互作预测
以拟南芥、工业大麻和马铃薯的WOX蛋白序列在MEGA X软件上,使用邻接法建树,(参数设置:模型为p-distance,缺失数据方法为Partial deletion,cutoff为50%,Bootstrap设置为1 000)参数为默认参数,利用在线工具网站Evolview(https://www.evolgenius.info/evolview/#/)进行进化树美化。利用软件jalviewg进行保守结构域多序列比对,利用PS软件对比对结果进行美化作图。利用TBtools软件的“One Step MCScanX-Super Fast”等功能对拟南芥、工业大麻和马铃薯的WOX基因家族进行物种间的共线性分析并作图。利用String数据库(https://cn.string-db.org/)并以拟南芥为参考植物,将相关蛋白序列提交String数据库中进行预测蛋白互作(参数设置:最小置信度为0.40,最大蛋白预测数量为10)。
1.3 基因家族蛋白保守基序、启动子元件预测
利用在线工具MEME(https://meme-suite.org/meme/tools/meme)分析蛋白的保守基序(保守基序寻找数量为10),使用TBtools工具软件进行可视化制图。利用TBtools提取工业大麻CsWOX基因的上游2000bp区域的启动子序列,利用在线工具PlantCARE(https://bioinformatics.psb.ugent.be/webtools/plantcare/html/)来预测CsWOX启动子区域的作用元件,在启动子图谱中标记和筛选数量排名前25的结合位点,然后采用TBtools可视化作图。
1.4 CsWOX基因家族GO富集分析
富集气泡图利用TBtools软件对CsWOX基因生物过程富集分析,以P值小于0.05作为为显著性富集的阈值,选取最显著的10个Term,利用R语言程序对富集结果作图并美化输出。气泡网络图利用Cytoscape软件的Gluego插件对CsWOX基因家族的所有GO生物过程进行可视化,绘图参数设置如下:比对品种选用为工业大麻(Cannabis sativa),启用GO项目融合(GO Term Fusion),仅显示置信度大于95%的节点,卡帕参数(Kappa Score)设置为0.12,Go Tree Interval设置在3到5之间。
1.5 工业大麻的CsWOX基因家族的转录组数据数据分析
植物材料为纤维品种工业大麻扦插苗,品种为云麻7号,在温室中生长,光周期为16/8,光照强度为1 500μmol/m2/s,材料生长周期为6个月,对植物的花,茎,根,茎侧枝,叶柄,叶组织取样后送测,每个组织或器官设置3个生物学重复。干旱胁迫处理下的工业大麻对钩状木霉(Trichoderma hamatum)的转录组的相关表达矩阵从NCBI的GEO数据库(https://www.ncbi.nlm.nih.gov/geo/)下载,登陆号为GSE266916,该表达矩阵设置4个处理,分别为对照组,钩状木霉接种处理,干旱胁迫处理,干旱胁迫和钩状木霉接种处理,转录组取样部位为植物叶片,每个处理设置3个生物学重复,转录组表达矩阵使用TBtools语言工具软件对CsWOX基因家族在对不同部位的表达量进行log2FC函数计算与聚类分析,并将结果绘制表格与图片。
2 结果与分析
2.1 工业大麻CsWOX基因家族理化性质预测
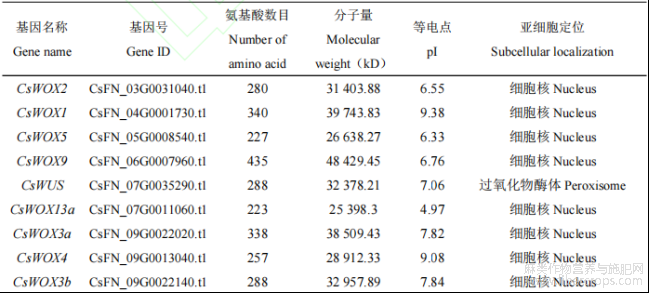
筛选得到的11个工业大麻CsWOX基因家族成员,新鉴定的工业大麻CsWOX基因家族成员的命名参考系统发育邻接树和染色体位置(表1)。进一步分析其蛋白理化性质以及亚细胞位置,结果表明,工业大麻WOX转录因子蛋白氨基酸数目分布在223(CsWOX13a)至435(CsWOX9)aa之间;相对分子质量分布在25398.3 Da(CsWOX13a)至48429.45 Da(CsWOX9)之间;等电点分布在4.97(CsWOX13a)至9.38(CsWOX1)之间,该基因家族蛋白多数呈酸性(pI<7),其中6个呈酸性蛋白,5种为碱性蛋白。多数蛋白定位于细胞核中(表1),仅有CsWOX6以及CsWOX12定位于细胞核外,表明该基因族主要定位于细胞核中进行功能行使。综合研究结果表明,所筛选得到的工业大麻WOX转录因子成员蛋白理化性质存在差异。
表1 工业大麻WOX转录因子家族成员信息
2.2 多序列比对
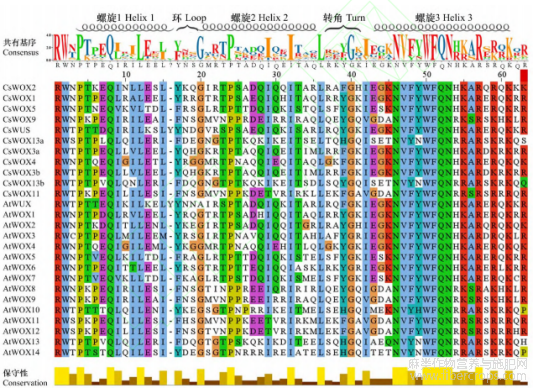
根据现有报道的拟南芥WOX转录因子成员蛋白序列和工业大麻WOX转录因子成员蛋白多重序列对比结果(图1)显示,工业大麻和拟南芥WOX转录因子成员均含有由61个或62个氨基酸残基构成的螺旋-环-螺旋-转角-螺旋(helix-loop-helix-turn-helix)同源异型保守结构域,这表明该结构域对植物WOX转录因子功能的完整性起着关键作用。另外,该结构域包含了已报道的保守位点(Mukherjee et al.,2009),螺旋1中的Q8和L12位置,以及螺旋3中的V47、W50、F51、Q52位置,这些位点在工业大麻和拟南芥的WOX转录因子家族成员中同样表现出高度的保守性。另外,在拟南芥的AtWUS和工业大麻的CsWUS的同源异型结构域中,16号位出现一个酪氨酸残基插入突变,这提示该残基可能在WUS类转录因子特异性识别下游顺式作用元件过程中发挥一定的作用。
图1 WOX蛋白保守结构域的多序列比对
2.3 构建及分析系统进化树
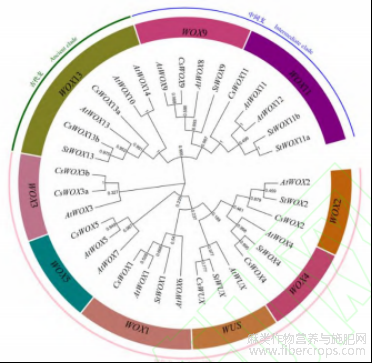
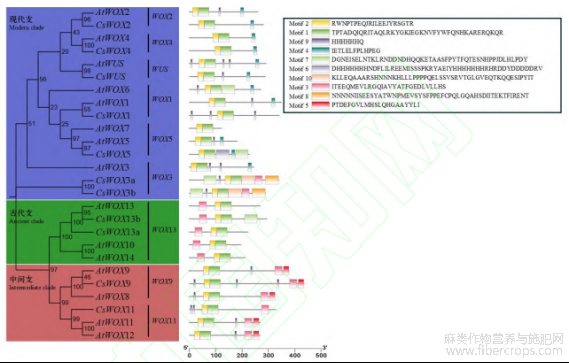
为了分析不同物种WOX转录因子之间的进化关系,下载15条拟南芥、11条工业大麻和8条马铃薯的WOX转录因子蛋白全长序列采用邻接法构建系统进化树(图2)。拟南芥、工业大麻和马铃薯的WOX基因家族成员均可分为3个进化支:古代支(Ancient clade)、中间支(Intermediate clade)以及现代支(Modern clade),它们还可以分为9个进化亚支。现代进化支有WUS、WOX1、WOX2、WOX3、WOX4、WOX5,共计6个亚支;中间进化支有WOX9、WOX11,共计2个亚支;古代支只有WOX13亚支。此外,各亚支中的工业大麻、拟南芥和马铃薯WOX基因家族的成员数量亦有差别。在WOX3与WOX5亚支中,马铃薯的WOX基因家族成员未找到对应成员;在WOX13亚支中,工业大麻有2个成员(CsWOX13a和CsWOX13b),马铃薯有1个成员StWOX13,拟南芥有3个成员AtWOX10、AtWOX13和AtWOX14。这些情况说明工业大麻、拟南芥和马铃薯在分化以后,WOX13、WOX3和WOX5等亚支经历的进化事件不同。
图2 工业大麻和拟南芥及马铃薯 WOX 基因家族的系统发育树
2.4 工业大麻CsWOX基因共线性分析
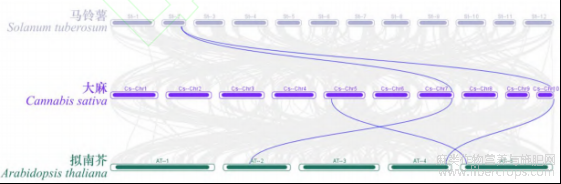
为进一步了解工业大麻大麻CsWOX基因的进化机制,体用TBtools软件构建了工业大麻、马铃薯(Solanum tuberosum)、拟南芥(Arabidopsis thaliana)的WOX转录因子家族的共线性关系图谱(图3),St表示马铃薯的染色体,Cs-Chr表示工业大麻的染色体,AT表示拟南芥的染色体,结果表明,工业大麻于马铃薯有2对同源基因,与拟南芥存在3对同源基因,进一步说明所鉴定的相关工业大麻CsWOX基因与部分拟南芥及马铃薯相关的WOX基因直系同源。
图3 种间共线性分析
2.5 蛋白质保守基序分析
分析蛋白结构有助于揭示基因家族的进化规律。利用在线软件MEME对拟南芥及工业大麻WOX家族成员的蛋白保守基序进行分析发现(图4),11个CsWOX转录因子蛋白序列中共预测出10个保守的蛋白质基序(Motif),所有家族成员均含有Motif 1和Motif 2,Motif 1和Motif 2为组合在一起的同源结构域,侧面反映了该预测分析的可靠性。此外,同一亚支的WOX转录因子保守基序种类及组织形式存在一定保守性,古代支的WOX13亚支中除了包含Motif 1和Motif 2这一同源结构域外,其蛋白N端还均包含保守基序Motif 3。此外,保守基序Motif 5只分布在中间支的WOX11及WOX9亚支中,只有CsWOX11不包含Motif 5,暗示Motif 5在中间支中发挥独特作用。然而,Motif 4只分布于现代支中且普遍靠近蛋白序列C端。
图4 WOX 转录因子保守基序分析
2.6 启动子区域的顺式元件分析
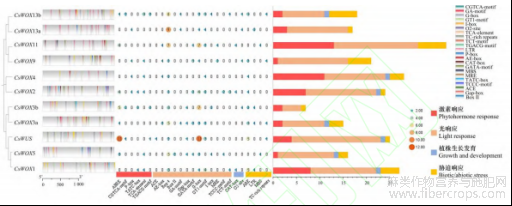
通过PlantCARE在线工具预测工业大麻CsWOX基因家族启动子区的顺式元件,预测结果在去除家族成员必须包含的CAAT-box、TATA-box核心元件及功能未知及数量稀少的元件后,进一步利用Tbtools可视化(图5),11个基因中主要含有25种作用元件,其中和光相关响应(Gap-box等元件)、赤霉素响应(TATC-box,TCA-element等元件)、脱落酸响应(ABRE等元件)相关的元件数量最多,且在多数CsWOX基因家族成员的启动子区域中都有分布。其他的元件如生长素响应、水杨酸响应、厌氧响应、昼夜节律响应、低温和防御响应等不均匀分布于CsWOX基因家族成员启动子中。进一步,将25种启动子顺式作用元件分为4大类:其中激素响应元件(Phytohormone responsive)有6种,共计54个;光响应相关元件(Light responsive element)有13种,共计141个;胁迫响应相关元件(Biotic/abiotic stress)有种,共计33个;植株生长发育相关元件(Growth and development)有2种,共计7个。表明CsWOX基因家族在植物光响应,环境感知、激素响应、光合作用和代谢过程中受多种顺式作用元件调控。此外,CsWO3b含有的顺式元件数量最少,为7个;CsWOX11含有的顺式元件数量最多,为37个,暗示基因CsWOX11可能受到更多调控信号的影响,能够响应不同的环境或生理条件,通过多个顺式作用元件来调控表达,以应对特定的生理需求或环境变化。
2.7 工业大麻WOX转录因子GO富集分析
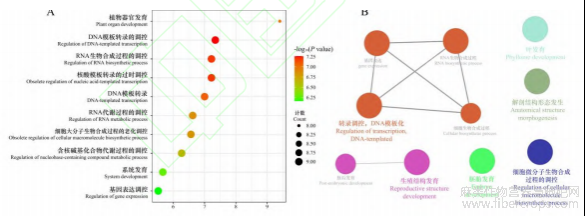
为了研究CsWOX基因家族在工业大麻中的生物学功能,我们对CsWOX基因家族成员进行GO生物过程富集分析,以P值小于0.05作为为显著性富集的阈值,选取最显著的10个Term,结果如(图6:A)所示,其生物过程主要注释到DNA模板转录的调控、核酸模板转录的过时调控、RNA生物合成过程的调控等途径。利用Cytoscape软件的Gluego插件对CsWOX基因家族的所有GO生物过程进行可视化,如(图6:B)所示,每个节点是代表性富集通路,共富集到10个节点,不同颜色表示该节点的富集情况分类,共聚集到6个分类,分为叶发育、转录调控及DNA模板化、生殖结构发育、细胞大分子生物合成过程的调控等。
图5 CsWOX 基因家族启动子区域的顺式元件分析
图6 工业大麻 CsWOX 基因家族成员 GO 富集分析
2.8 工业大麻WOX基因表达模式分析
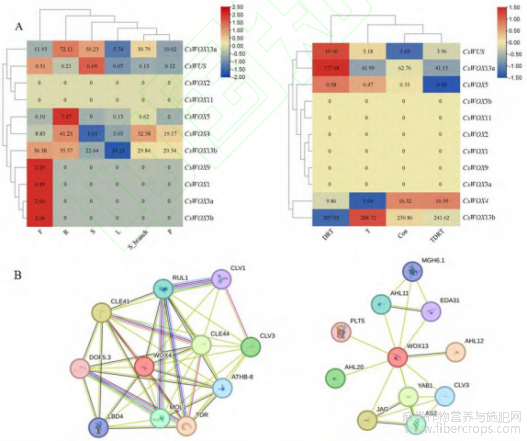
为了研究CsWOX基因在工业大麻中的表达模式,我们分析了CsWOX基因在工业大麻主干茎不同部位的表达量(图7:A)。结果表明,工业大麻CsWOX基因的表达具有组织特异性。进一步的分析发现,工业大麻CsWOX4及WOX13亚支的成员CsWOX13a和CsWOX13b基因在所有组织中均有较高的表达量,CsWOX1、CsWOX9、CsWOX3a、CsWOX3b这4个基因具有相似的表达模式,仅在花中特异性表达,CsWUS仅在茎与花中表达量表达,CsWOX5仅在根中差异高表达。此外,我们进一步分析了不同处理对CsWOX基因表达的影响,与对照组相比,CsWOX5基因在干旱处理及钩状木霉共处理中表达量上调,在干旱及钩状木霉共处理中表达量下降;与对照组相比,CsWUS基因表达量在所有处理中均有上调;CsWOX13a基因表达量仅在干旱处理中上调,CsWOX13b基因表达量仅在钩状木霉处理中上调,CsWOX4基因表达量在干旱处理及钩状木霉处理中均下调,其余CsWOX基因表达未检测出。值得注意的是,CsWOX4和CsWOX13a/b不仅在不同组织部位中表达量较高;且与对照组相比,CsWOX4和CsWOX13a/b在干旱处理、钩状木霉处理及干旱和钩状木霉共处理的表达量发生变化,推测其可能在工业大麻生长发育以及植株抗逆过程中发挥潜在作用。
进一步研究发现,CsWOX4、CsWOX13a、CsWOX13b不仅在工业大麻不同组织部位相对高表达,还在干旱处理及钩状木霉处理中差异表达,暗示CsWOX4、CsWOX13a、CsWOX13b基因可能在植株生长发育及逆境调控中发挥重要作用。为进一步明晰工业大麻CsWOX4、CsWOX13a、CsWOX13b基因在工业大麻生长发育及逆境调控中发挥的作用,本研究以拟南芥为参考,对CsWOX4、CsWOX13b基因在工业大麻生长发育及逆境调控中发挥的作用,本研究以拟南芥为参考,对CsWOX4、CsWOX13a、CsWOX13b基因编码的蛋白进行互作预测。结果显示工业大麻CsWOX4转录因子与拟南芥WOX4蛋白同源,后者被预测与TDR、CLV3、CLE41及CLE44等蛋白存在互作关系(图7:B)。工业大麻CsWOX13a及CsWOX13b转录因子与拟南芥WOX13蛋白同源,后者被预测与CLV3、EDA31、AHL11等蛋白存在互作关系(图7:B)。
图 A 为 CsWOX 基因表达聚类图。图 B 为 WOX4(左)及 WOX13(右)蛋白互作预测分析网络图。
F. 花;S.茎;R. 根;S-branch. 茎侧枝;P. 叶柄;L. 叶;Con. 对照组;DRT. 干旱处理;T. 钩状木霉处理;TDRT. 干旱与钩状木霉处理。
图7 CsWOX 基因表达量聚类分析及蛋白互作预测
3 讨论与结论
本研究基于生物信息学方法,从工业大麻中鉴定出11个GsWOX转录因子编码基因,亚细胞定位预测结果显示,大多数GsWOX蛋白定位于细胞核内,氨基酸数量、等电点及相对分子质量等理化性质存在一定程度的差异,这与李晓旭等(2016)对番茄WOX家族的研究结果相似,提示GsWOX基因家族可能参与细胞核内基因表达调控与遗传信息的传递,同时,不同成员可能在植物生长发育过程中发挥不同功能。系统发育分析表明,工业大麻与拟南芥、马铃薯WOX基因家族共同分为3个主要进化分支(现代,中间,古代)及9个亚支。然而,工业大麻(820 Mb)与马铃薯(844 Mb)和拟南芥(135 Mb)在基因组大小上存在显著差异,其WOX基因家族成员数量却较为接近,由此推测WOX基因分化时间要早于物种分化时间,值得注意的是,工业大麻WOX基因在系统进化树中的表达模式与植物存在一定差异:例如,古代型分支的CsWOX13a/b基因在工业大麻花器官发育中的表达模式与拟南芥显著不同,这可能反映了工业大麻作为雌雄异株植物的特殊进化适应(Costanzo et al.,2014)。例如,拟南芥AtWOX13基因主要调控叶片边缘发育(Vandenbussche et al.,2009),而CsWOX13a/b在工业大麻中则表现出更强的花器官特异性表达。这些发现为理解工业大麻WOX基因的功能分化提供了新的见解。
WOX转录因子具有时空表达特性,其转录因子的时空表达特性是生物体在不同生理条件下保持正常发育的关键(Kaufmann et al.,2010),这种模式在其他物种中广泛存在,如烟草WOX9在花器官中特异性表达、WOX5则在根中表达量较高(李晓旭等,2021),油桐WOX13在根和茎中的表达较高(王占军等,2024),柑橘WUS基因在茎中表达较高,WOX9则主要在花中表达(Shafique et al.,2021),马铃薯WOX1在花中表达量较高。与上述结果类似,本文CsWOX基因表达同样表现出组织表达特异性。通过启动子区域的顺式元件的预测分析表明,CsWOX基因家族成员的启动子含有丰富的顺式作用元件,这些元件不仅涉及光响应,还参与激素信号传导、环境胁迫应答及植物的生长发育调控。例如,ABRE是ABA信号通路的关键元件,当植物遭遇干旱、高盐、低温等非生物胁迫时,内源ABA水平升高,ABRE通过招募转录因子激活胁迫相关基因,提高植物抗逆性(Hossain et al.,2010);O2-site通过结合转录因子O2(Opaque-2)驱动胚乳中贮藏蛋白的合成,直接影响种子营养品质和农艺性状(Schmidt et al.,1992)。所有CsWOX基因的启动子区域中,参与光响应的顺式元件数量最多,符合前人研究结果(杨旭,2023),其中G-box元件数量最多,G-box为光响应调控的核心元件之一,光敏色素(phyB)通过感知光照以激活转录因子PIF7直接结合G-box元件,来调控植株光形态建成(Li et al.,2012)。综上,CsWOX基因可能参与工业大麻的组织生长、逆境响应以及器官发育调控过程,但其具体分子调控机制有待进一步开发。
系统进化树结果表明基因家族各成员之间基因结构差异体现出亲缘关系,本研究预测工业大麻WOX蛋白包含10个保守的结构基序(Motif),进化树各支系的Motif在数量上存在差异,但几乎均具有特异性的Motif特征。此外,WOX转录因子均含有由两个核心Motif(Motif 1和Motif 2)构成的同源盒结构的保守结构域,该保守结构是识别并结合下游DNA的关键结结构域,特别是在第三螺旋(α3)中的R/K残基为关键氨基酸位点,这些残基通过氢键或静电作用直接与DNA双螺旋的碱基或磷酸骨架结合(Sloan et al.,2020),若这些残基突变,可能破坏与DNA的相互作用,导致结合能力下降甚至丧失。跨物种比较分析揭示WOX转录因子关键功能位点的进化保守性:在拟南芥,工业大麻及马铃薯(叶明辉等,2021)、白桦(程大伟等,2025)、枳(马青龄等,2022)等植物中,WOX13同源异型结构域第49位氨基酸发生特异性置换(天冬氨酸替换为酪氨酸);而WOX4第37位甘氨酸则高度保守,未检测到37位甘氨酸位点突变。这些结构特征提示,WOX13的49位点可能参与物种特异性调控元件的识别,而WOX4的37位点则在所有分析物种中严格保守,提示其可能在维持DNA结合基序三维构象中具有不可替代性,在维持家族蛋白DNA结合功能中发挥核心作用。
K?pczyńska等(2020)研究发现,植物促生微生物通过合成生长素类似物,调控细胞周期及WOX5基因的表达,从而促进根系发育,优化根系构型。这一机制为微生物肥料的开发及植物-微生物互作研究提供了重要的理论依据。钩状木霉可能通过双重机制调控WOX基因:一方面通过分泌吲哚乙酸(IAA)等植物激素(Bae et al.,2009),直接激活WOX家族基因表达,促进根系构型优化和纤维发育;另一方面,其分泌的超氧化物歧化酶(SOD)、多酚氧化酶(PPO)等抗氧化物质(Abdelkhalek et al.,2022),可有效清除过氧化氢(H?O?)等活性氧组分,缓解干旱引发的氧化损伤。这一协同作用机制能够解释本研究的在单一干旱胁迫下,工业大麻通过下调WOX4表达收缩水分运输系统以降低蒸腾损耗;而当与钩状木霉共处理时,微生物介导的氧化压力缓解机制延迟了植物的胁迫响应进程,使WOX4表达量维持在接近对照组(图7:A)。此外,WOX13在植物生长发育和逆境响应中发挥潜在作用,具有一定的抗逆育种潜力,研究表明,水稻中的OsWOX13参与非生物胁迫耐受性和早期开花(Minh-Thu et al.,2018)。在干旱胁迫下WOX13被诱导表达(张玉等,2024)。CsWOX13a/b在所有组织中均有较高的表达量,且CsWOX13a/b在干旱和钩状木霉处理条件下的基因表达量均有显著的变化,对应的同源基因AtWOX13参与干细胞形成、激活细胞生长等生物学过程(王爽,2019),参与调节植株维管形成层活性和木材形成(Haghighat et al.,2024),调节植株抗旱性(Lv et al.,2023),推测工业大麻CsWOX13a/b基因可能具有相似的功能。上述结果表明,工业大麻CsWOX基因家族在组织表达特异性和非生物胁迫响应能力方面表现出相似的模式,暗示相应基因可能参与工业大麻生长发育过程和非生物胁迫响应。
综上,本研究系统分析了工业大麻CsWOX基因家族,鉴定得到11个CsWOX家族成员,并且所有CsWOX转录因子均含有Motif 1和Motif 2构成的螺旋-环-螺旋-转角-螺旋(同源异型)保守结构域,蛋白长度为202~435 aa;相对分子量为25398.3~48429.45 Da;等电点为4.97~9.38,分析和预测了CsWOX基因家族成员生物信息学功能,并根据基因表达量对其表达模式做出分析,本研究为工业大麻的分子育种提供了相关靶点,通过精准调控这些基因的表达,有望培育出具有多重抗逆性的工业大麻品种,为作物抗逆育种提供新的思路和方法。未来结合基因编辑技术和多组学分析,进一步挖掘其在作物改良中的应用价值,可以为进一步解析该家族功能解析提供一定的参考。
参考文献
ABDELKHALEK A,AL-ASKAR AA,ARISHI AA,et al.,2022.Trichoderma hamatum strain Th23 promotes tomato growth and induces systemic resistance against tobacco mosaic virus[J].Journal of Fungi,8(3):228.
AMADUCCI S,ZATTAA,PELATTI F,et al.,2008.Influence of agronomic factors on yield and quality of hemp(Cannabis sativa L.)fibre and implication for an innovative production system[J].Field Crops Research,107(2):161-169.
ANDRE CM,HAUSMAN JF,GUERRIERO G,2016.Cannabis sativa:the plant of the thousand and one molecules[J].Frontiers in Plant Science,7:19.
BAE H,SICHER RC,KIM MS,et al.,2009.The beneficial endophyte Trichoderma hamatum isolate DIS 219b promotes growth and delays the onset of the drought response in Theobroma cacao[J].Journal of experimental botany,60(11):3279-3295.
CHENG DW,WANG HH,SUN ZX,et al.,2025.Identification and expression analysis of BpWOX gene family in Betula platyphylla[J/OL].Molecular Plant Breeding:1-14.http://kns.cnki.net/kcms/detail/46.1068.s.20230301.1648.025.html.[程大伟,王晗慧,孙泽新,等.2025.白桦BpWOX基因家族鉴定与表达分析[J/OL].分子植物育种:1-14.http://kns.cnki.net/kcms/detail/46.1068.s.20230301.1648.025.html.]
CHENG S,ZHOU DX,ZHAO Y,2016.WUSCHEL-related homeobox gene WOX11 increases rice drought resistance by controlling root hair formation and root system development[J].Plant Signaling&Behavior,11(2):e1130198.
COSTANZO E,TREHIN C,VANDENBUSSCHE M,2014.The role of WOX genes in flower development[J].Annals of GEHRING WJ,MÜLLER M,AFFOLTER M,et al.,1990.The structure of the homeodomain and its functional implications[J].Trends in Genetics,6:323-329.
GEHRING WJ,QIAN YQ,BILLETER M,et al.,1994.Homeodomain-DNA recognition[J].Cell,78(2):211-223.
HAGHIGHAT M,ZHONG R,YE ZH,2024.WUSCHEL-RELATED HOMEOBOX genes are crucial for normal vascular organization and wood formation in poplar[J].Plant Science,346:112138.
HAO Q,ZHANG L,YANG Y,et al.,2019.Genome-wide analysis of the WOX gene family and function exploration of GmWOX18 in soybean[J].Plants,8(7):215.
HIRAKAWAY,KONDO Y,FUKUDA H,2010.TDIF peptide signaling regulates vascular stem cell proliferation via the WOX4 homeobox gene in Arabidopsis[J].The Plant Cell,22(8):2618-2629.
HOSSAIN MA,CHO JI,HAN M,et al.,2010.The ABRE-binding bZIP transcription factor OsABF2 is a positive regulator of abiotic stress and ABA signaling in rice[J].Journal of Plant Physiology,167(17):1512-1520.
KAUFMANN K,PAJORO A,ANGENENT GC,2010.Regulation of transcription in plants:mechanisms controlling developmental switches[J].Nature Reviews Genetics,11(12):830-842.
K?PCZY?SKA E,KARCZY?SKI P,2020.Medicago truncatula root developmental changes by growth-promoting microbes isolated from Fabaceae,growing on organic farms,involve cell cycle changes and WOX5 gene expression[J].Planta,251(1):25.
LI CX,CHEN L,JIANG T,et al.,2016.Identification and expression analysis of WUSCHEL-related homeobox(WOX)gene family in moso bamboo[J].Molecular Plant Breeding(8):2003-2011.[李晨曦,陈磊,江涛,等.2016.毛竹WUSCHEL-related homeobox(WOX)基因家族鉴定及表达分析[J].分子植物育种(8):2003-2011.]
LI L,LJUNG K,BRETON G,et al.,2012.Linking photoreceptor excitation to changes in plant architecture[J].Genes&development,26(8):785-790.
LI X X,GUO C,PU WX,et al.,2021.Genome-wide identification and systemic analysis of WOX family genes in tobacco[J].Acta Tabacaria Sinica,(1):90-100.[李晓旭,郭存,蒲文宣,等,2021.普通烟草WOX转录因子家族的全基因组鉴定及分析[J].中国烟草学报,(1):90-100.]
LIE C,KELSOM C,WU X,2012.WOX2 and STIMPY‐LIKE/WOX8 promote cotyledon boundary formation in Arabidopsis[J].The Plant Journal,72(4):674-682.
LUO K S,ZHANG MH,ZHAI ZD,et al.,2024.Identification and analysis of WOX gene family in lettuce[J].Molecular Plant Breeding(6):1795-1806.[罗康胜,张米欢,翟兆东,等,2024.生菜WOX基因家族的鉴定与分析[J].分子植物育种(6):1795-1806.]
LV J,FENG Y,JIANG L,et al.,2023.Genome-wide identification of WOX family members in nine Rosaceae species and a functional analysis of MdWOX13-1 in drought resistance[J].Plant Science,328:111564.
MA QL,LIANG BB,YANG L,Genome-wide identification and expression analysis of WOX gene family in trifoliate orange[J].Journal of Fruit Science,(5):712-729.[马青龄,梁贝贝,杨莉,等,2022.枳WOX基因家族全基因组鉴定及表达分析[J].果树学报,(5):712-729.]
MINH-THU PT,KIM JS,CHAE S,et al.,2018,A WUSCHEL homeobox transcription factor,OsWOX13,enhances drought tolerance and triggers early flowering in rice[J].Molecules and Cells,41(8):781-798.
MUKHERJEE K,BROCCHIERI L,BÜRGLIN TR,2009.A comprehensive classification and evolutionary analysis of plant homeobox genes[J].Molecular biology and evolution,26(12):2775-2794.
RASHEED H,SHI L,WINARSIH C,et al.,2024.Plant Growth Regulators:An Overview of WOX Gene Family[J].Plants,13(21):3108.
ROMERA‐BRANCHAT M,RIPOLL JJ,YANOFSKY MF,et al.,2013.The WOX 13 homeobox gene promotes replum formation in the Arabidopsis thaliana fruit[J].The Plant Journal,73(1):37-49.
SAJJAD M,WEI X,LIU L,et al.,2021.Transcriptome analysis revealed GhWOX4 intercedes myriad regulatory pathways to modulate drought tolerance and vascular growth in cotton[J].International Journal of Molecular Sciences,22(2):898.
SCHMIDT RJ,KETUDAT M,AUKERMAN MJ,et al.,1992.Opaque-2 is a transcriptional activator that recognizes a specific target site in 22-kD zein genes[J].The Plant Cell,4(6):689-700.
SHAFIQUE KHAN F,ZENG RF,GAN ZM,et al.,2021.Genome-wide identification and expression profiling of the WOX gene family in Citrus sinensis and functional analysis of a CsWUS member[J].International Journal of Molecular Sciences,22(9):4919.
SHAHZAD A,2012.Hemp fiber and its composites–a review[J].Journal of composite materials,46(8):973-986.
SHIMIZU R,JI J,KELSEY E,et al.,2009.Tissue specificity and evolution of meristematic WOX3 function[J].Plant physiology,149(2):841-850.
SLOAN J,HAKENJOS JP,GEBERT M,et al.,2020.Structural basis for the complex DNA binding behavior of the plant stem cell regulator WUSCHEL[J].Nature Communications,11(1):2223.
STAMENKOVI?S,BEŠKOSKI V,KARABEGOVI?I,et al.,2018.Microbial fertilizers:A comprehensive review of current findings and future perspectives[J].Spanish Journal of Agricultural Research,16(1):e09R01.
UEDA M,ZHANG Z,LAUX T,2011.Transcriptional activation of Arabidopsis axis patterning genes WOX8/9 links zygote polarity to embryo development[J].Developmental Cell,20(2):264-270.
VAN BAKEL H,STOUT JM,COTE AG,et al.,2011.The draft genome and transcriptome of Cannabis sativa[J].Genome Biology,12:1-18
VAN DER GRAAFF E,LAUX T,RENSING SA,2009.The WUS homeobox-containing(WOX)protein family[J].Genome Biology,10:1-9.
VANDENBUSSCHE M,HORSTMAN A,ZETHOF J,et al.,2009.Differential recruitment of WOX transcription factors for lateral development and organ fusion in Petunia and Arabidopsis[J].The Plant Cell,21(8):2269-2283.
WANG D,QIU Z,XU T,et al.,2024.Identification and expression patterns of WOX transcription factors under abiotic stresses in Pinus massoniana[J].International Journal of Molecular Sciences,25(3):1627.
WANG S,2019.Functional characterization of Populus PtrWOX13 during the secondary cell wall formation[D].Harbin:Northeast Forestry University:15-16.[王爽,2019.PtrWOX13在杨树次生细胞壁形成中的功能分析[D].哈尔滨:东北林业大学:15-16.]
WANG ZJ,LI LL,ZHANG M,et al.,2024.Genome-Wide identification and analysis of WOX transcription factor family in Vernicia fordii[J].Journal of Northwest Forestry University,39(5):111-117.[王占军,李玲丽,张萌,等.2024.油桐WOX转录因子家族的全基因组鉴定与分析[J].西北林学院学报,39(5):111-117.]
WU CC,LI FW,KRAMER EM,2019.Large-scale phylogenomic analysis suggests three ancient superclades of the WUSCHEL-RELATED HOMEOBOX transcription factor family in plants[J].PLoS ONE,14(10):e0223521.
XIAO XL,CHENG L,WEI L,et al.,2016.Genome-wide identification,phylogenetic analysis and expression profiling of the WOX family genes in Solanum[J].lycopersicum Hereditas(Beijing),38(5):444-460.[李晓旭,刘成,李伟,等.番茄WOX转录因子家族的鉴定及其进化、表达分析[J].遗传,38(5):444-460.]
XU L,2021.WUSCHEL:The versatile protein in the shoot apical meristem[J].Science China Life sciences,64(1):177-178.
YANG Z,GONG Q,QIN W,et al.,2017.Genome-wide analysis of WOX genes in upland cotton and their expression pattern under different stresses[J].BMC Plant Biology,17:1-17.
YE M H,ZHAO P,NIU Y,et al.,2021.Identification and expression analysis of homeobox gene family in potato(Solanum tuberosum)[J].Journal of Agricultural Biotechnology.29(2):224-239.[叶明辉,赵朋,牛洋,等.2021.马铃薯同源异形框基因家族的鉴定和表达分析[J].农业生物技术学报,29(2):224-239.]
YNNG X,2023.Genome-wide identification and expression characteristics analysis of WOX gene family members in woodland strawberry[D].Shenyang:Shenyang Agricultural University:35-36.[杨旭,2023.森林草莓WOX家族成员鉴定及表达特性分析[D].沈阳:沈阳农业大学:35-36.]
ZHANG Y,SHI L,GONG L,et al.,2024.Genome-wide identification of potato WOX gene family and its expression analysis in in vitro regeneration and abiotic stress[J].Biotechnology Bulletin,40(3):170-180[张玉,石磊,巩檑,等.2024.马铃薯WOX基因家族的鉴定及在离体再生和非生物胁迫中的表达分析[J].生物技术通报,40(3):170-180.]
ZHOU X,HAN H,CHEN J,et al.,2024.The emerging roles of WOX genes in development and stress responses in woody plants[J].Plant Science:112259.
?YDELIS R,HERBST M,WEIHERMÜLLER L,et al.,2022.Yield potential and factor influencing yield gap in industrial hemp cultivation under nemoral climate conditions[J].European Journal of Agronomy,139:126576.
文章摘自:田岳,黄紫樱,刘圆圆.工业大麻WOX基因家族的全基因组鉴定及表达分析[J/OL].广西植物,1-15[2025-07-05].http://kns.cnki.net/kcms/detail/45.1134.Q.20250619.1143.004.html.