摘 要:始花节位与纤维产量密切相关,然而有关始花节位的遗传机制仍然不清楚。以始花节位差异显著的红麻自交系K215和K89为亲本,构建了含有148个单株的F2群体。利用RAD-seq测序的方法进行bin标记基因分型,构建了红麻高密度遗传连锁图谱。该图谱含有3418个bin标记且不均匀的分布在18条染色体上,图谱总遗传距离为864.61cM,标记间平均遗传距离为0.25cM。结合始花节位的表型数据和遗传图谱,利用区间作图法,检测到1个与始花节位密切相关的QTL位点FFN6。该位点位于第6号染色体,LOD值为8.66,表型贡献率为13.77%,加性效应值为-19.97。结果表明该等位基因位点来自父本K215,对始花节位具有负调控作用。根据QTL定位区间内基因注释信息,初步筛选获得7个候选基因。qRT-PCR结果表明,短日照条件下Hc-bHLH6和Hc-MYB6基因的表达量显著增加,促进红麻植株开花,这2个候选基因可能与红麻植株始花节位密切相关。这一结果为一进步阐明红麻始花节位的遗传机制奠定了坚实的基础。
关键词:红麻;QTL;始花节位;遗传连锁图谱
红麻(Hibiscus cannabinus L.)是一年生短日照草本纤维作物。红麻纤维可以用于纺织、造纸、建筑材料等方面[1,2]。红麻纤维具有透气性、抗菌性和生物可降解性,是环境友好型天然纤维。近年来,随着人们环保意识的不断增强,世界对天然纤维的需求不断增加,提高红麻纤维产量,大力发展红麻产业是满足这一需求的重要途径。红麻的种植以收获韧皮纤维为主要目的,其纤维产量与第一朵花的开花节位密切相关。始花节位是从子叶节位到第一朵花开花所在位置的节位数。红麻植株开花后表示红麻植株的生长完全由营养生长转化为生殖生长。红麻植株进入生殖生长后纤维的积累速度和纤维质量急剧下降[3]。可见,始花节位越高,纤维产量就越高。因此,阐明始花节位的遗传规律并筛选、鉴定控制始花节位的有效基因将有助于红麻纤维产量育种。始花节位是多基因控制的数量性状,其遗传基础比较复杂[5]。高密度遗传图谱的构建和QTL定位分析是研究数量性状遗传规律的重要方法。Zhang等[4]以辣椒为研究材料,构建了包含9328个分子标记的高密度遗传连锁图谱,并定位到了2个与始花节位密切相关的主效QTL位点(Ffn2.1和Ffn2.2),分别能够解释28.62%和19.56%的表型遗传变异。Liu等[6]以番茄F2分离群体为材料,通过SSR分子标记构建遗传图谱并进行QTL分析,共定位到5个与始花节位密切相关的主效QTL位点,分别定位到2、3、5b、8b、11染色体上,分别能够解释7.2%~15.6%的表型遗传变异。近年来,很多研究人员构建了红麻高密度遗传图谱并对重要农艺性状进行了QTL定位。Zhang等[7]利用SNPs和InDels标记构建了红麻高密度遗传连锁图谱,定位到了112个与纤维产量和纤维质量密切相关的QTL位点。Li等[8]利用1997个SNP标记构建了红麻遗传图谱并定位到6个与株高、纤维产量、纤维质量相关的QTL位点。始花节位是红麻的重要农艺性状,然而到目前为止,有关红麻始花节位的遗传机制仍不清楚,也未见其相关的报道。本研究以红麻F2分离群体为材料,采用基于全基因组酶切位点的简化基因组测序(Restriction-site associated DNA sequencing,RAD-seq)方法,以SNP为分子标记构建高密度遗传连锁图谱,并对红麻始花节位进行QTL定位分析,对QTL位点相关基因进行筛选和鉴定,以期为阐明红麻始花节位的分子遗传机制及其精细定位,红麻纤维产量育种奠定坚实的基础。
1 材料和方法
1.1 供试材料
试验材料由湖南文理学院生命与环境科学学院红麻育种课题组提供。以始花节位为9的红麻自交系K215为母本,以始花节位为34红麻自交系K89为父本,于2019年5月18日种植于湖南长沙红麻育种基地,人工去雄杂交。2019年11月9日将人工杂交获得的F1种子种植于海南三亚,进行单株自交并收获F2种子。父母本和F2分离群体种植于海南三亚(2021年11月12日),单株自交并按单株收获种子。同时选取父母本和148个F2单株幼嫩的叶片液氮速冻后保存于超低温冰箱,用于高密度遗传图谱的构建和QTL定位分析。F2∶3群体按照株系于2022年11月15日种植于海南三亚。
为了进一步验证与始花节位QTL位点相关的基因,2023年6月13日选取中红麻16号健壮、饱满的种子播种于湖南长沙红麻育种基地。选取健壮整齐一致的红麻幼苗50株,然后分为5组。第一组幼苗作为对照,其余4组幼苗在3叶期接受8h的短日照处理,直到花蕾出现。采样时间从7月5日开始,每隔7d取样一次,直到8月4日(花蕾出现)。而对照组在处理组出现花蕾后30d才现出花蕾。每次取样都剪取所选植株的第一片真叶,液氮速冻后提取总RNA,并反转录为cDNA作为实时荧光定量PCR(qRT-PCR)的模板。
1.2 表型的测定和分析
分离群体F2种植于海南三亚(2021年11月12日),并统计F2分离群体始花节位数据。F2∶3群体于2022年11月15日种植于海南三亚。每个株系随机选取10株单株用于统计始花节位和纤维产量。所得数据采用Microsoft Excel 2019软件表进行统计,SPSS 22.0软件进行数据描述性和相关性分析。
1.3 简化基因组测序
用CTAB(2%)法分别提取父母本和F2分离群体单株幼嫩叶片的基因组DNA,然后将DNA样品运送到深圳华大基因科技有限公司进行简化基因组测序和基因分型。
1.4 高密度遗传连锁图谱的构建
将RAD测序结果进行过滤,去除接头序列和低质量序列,利用BWA软件将clean data比对到红麻参考基因组(https://ngdc.cncb.ac.cn/search/all?&q=kenaf)[7,9],同时利用GATK软件对多态性SNP位点进行检测。采用滑窗取bin的方式进行遗传图谱的构建[10],设置滑动窗口为10个SNP,将bin标记作为作图的标记,并通过R/qtl软件包(V1.7)构建高密度遗传连锁图谱[11]。
1.5 始花节位的QTL分析
采用R/qtl软件包(V1.7)的复合区间作图法(Composite interval mapping,CIM),结合高密度遗传连锁图谱和始花节位的表型数据进行QTL扫描。LOD阈值设定为3.0[12],然后采用R包qtl进行QTL定位分析。QTL位点的命名以“Q+性状英文缩写+QTL位点所在染色体”,如QFFN6。
1.6 始花节位候选基因的筛选和鉴定
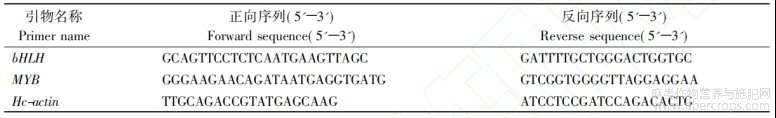
根据QTL定位检测获得的QTL候选区间,以红麻基因组序列[7]为参考,提取该区间内的基因序列,然后对该区间内的基因进行GO,KEEG和NR[13]数据库的功能注释以确定候选基因[14]。同时为了进一步确定候选基因,采用qRT-PCR的方法对其进行验证,以Hc-Actin为内参基因。qRT-PCR反应体系:正向和反向引物各0.8μL,qPCR mix 10μL,cDNA 20ng,ddH2O补足20μL。qRT-PCR扩增程序:95℃ 2min;95℃ 15s,60℃ 15,72℃ 60s,72℃ 2min,40个循环。引物序列见表1。
表1 实时荧光定量PCR引物序列
2 结果与分析
2.1 始花节位和纤维产量统计分析
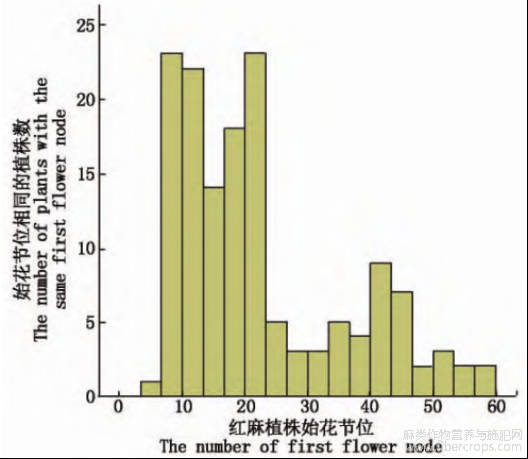
红麻F2∶3群体始花节位统计分析结果如图1所示。由图1可知,始花节位的表型数据呈现连续分布,具有数量性状的遗传特征。与亲本始花节位相比,F2∶3群体中出现了超亲分离现象,这表明控制始花节位的等位基因存在丰富的遗传变异。相关性分析结果表明,始花节位与纤维产量之间存在很强的线性关系,两者之间的相关系数为0.88,可见,始花节位越高,纤维产量也越高。红麻植株高度与纤维产量同样存在很强的线性关系,两者之间的相关系数为0.78。由此可知,红麻植株始花节位越高,植株高度越高,纤维产量就越高。
图1 红麻始花节位频率分布图
2.2 高质量遗传连锁图谱的构建
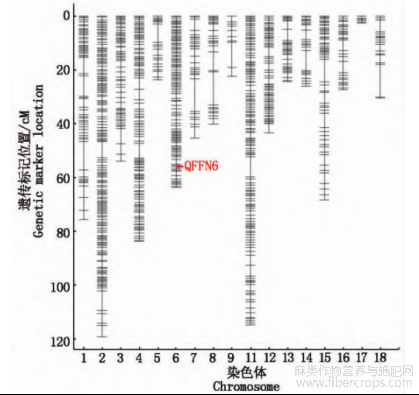
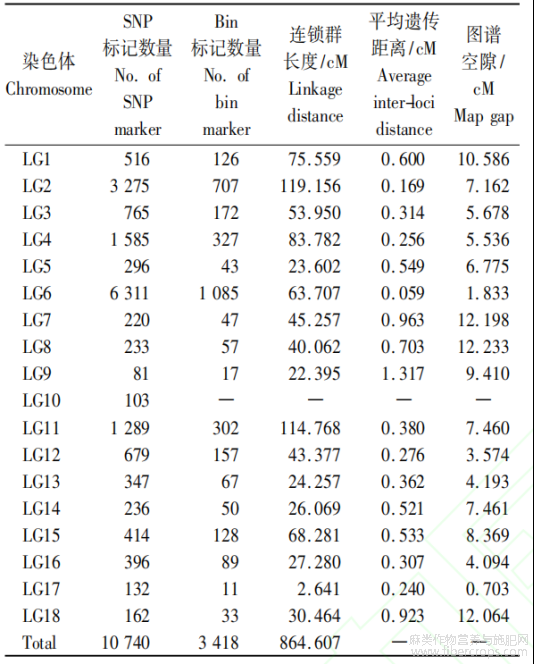
通过RAD测序获得多态性SNP位点17040个,且不均匀的分布在18条染色体上,结果如表2所示。通过滑动窗口基因分型合并冗余标记后获得3418个bin标记。根据3418个bin标记的基因型数据,利用R/qtl包(V1.7)构建红麻高密度遗传连锁图谱,结果如图2所示。由图2可知,3418个bin标记不均匀的分布在18条染色体。该图谱总遗传距离为864.61cM,平均每个bin标记间的遗传距离为0.25cM。在18条染色体中,遗传距离分布从2.64cM(17号染色体)到119.16cM(2号染色体),图谱中最大的空隙为12.06cM,位于18号染色体,最小的空隙为0.70cM,位于17号染色体。
图2 遗传连锁图和始花节位QTL定位
遗传标记最多的染色体是6号染色体,含有1085个bin标记,遗传标记最少的染色体是17号染色体,含有11个bin标记(图2,表2)。
表2 SNP和bin标记在红麻连锁图谱上的分布
利用构建的遗传连锁图谱,结合F2∶3群体植株始花节位的表型数据进行QTL位点扫描,在6号染色体上定位到1个QTL位点,其物理区间位于50.41~51.56Mb,将其命名为QFFN6(图2)。该QTL位点表型贡献率变为13.77%,LOD值为8.66,其等位基因位点来自父本K215,对始花节位具有负调控作用,其效应值为-19.97。
2.3 候选基因的筛选及其验证
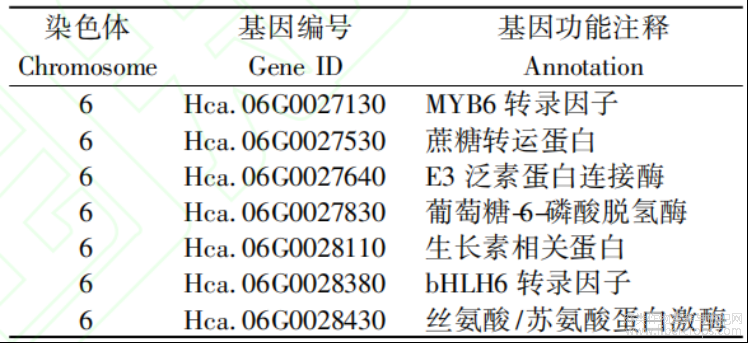
以红麻基因组序列为参考,根据QTL定位候选区间(50.41~51.56Mb)基因功能注释结果,初步筛选得到7个候选基因(表3)。根据前人的研究结果确定Hc-bHLH6[15]和Hc-MYB6[16]基因为目标候选基因,预测这2个基因通过调控红麻植株开花来影响第一朵花的位置。NR数据库功能注释结果表明Hc-bHLH6基因编码1个植物碱性螺旋-环-螺旋(Basic helix-loop-helix,bHLH)转录因子蛋白质,根据序列比对结果将其命名为Hc-bHLH6。bHLH转录因子是高等植物一类重要的转录因子,通过与靶基因的启动子结合,进而调控植物的生长发育和代谢进程,其中很多bHLH转录因子参与植物开花过程[17-18]。同时NR数据库功能注释结果表明Hc-MYB6基因编码1个MYB转录因子蛋白。MYB转录因子是植物中最大的转录因子家族之一,在调控植物开花过程具有重要作用[19]。
表3 QFFN6定位区间候选基因和功能注释
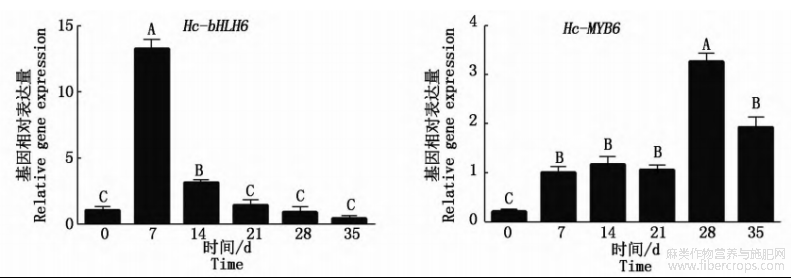
为了进一步验证QTL结果和始花节位相关的候选基因,利用qRT-PCR技术研究了短日照条件下Hc-bHLH6和Hc-MYB6的表达模式(图3)。由图3可知,基因Hc-bHLH6和Hc-MYB6在短日照条件处理7d后,基因表达量显著上调表达,且与对照相比具有极显著差异(以0d处理为对照),且Hc-MYB6基因在短日照处理期间始终处于高表达水平。综上所述,Hc-bHLH6和Hc-MYB6基因的表达受到短日照条件的诱导,推测这2个基因具有促进红麻植株开花的功能,且与始花节位的形成密切相关。
大写字母.与对照相比差异极显著(P<0.01);误差线为每组处理的标准误差(n=3)。
图3 短日照条件下Hc-bHLH6和Hc-MYB6的表达特征
3 结论与讨论
3.1 红麻高密度遗传连锁图谱的构建
高密度遗传连锁图谱的构建是重要农艺性状QTL定位和复杂数量性状遗传规律阐释的重要途径。Chen等[20]利用494个SRAP,60个ISSR和120个RAPD分子标记构建了第一张红麻遗传连锁图谱。由于该图谱分子标记数量有限,可重复差,很难用于QTL定位和数量性状遗传分析。本研究中利用SNP标记构建了一张红麻高密度遗传连锁图谱:
17040个多态性SNP分子标记整合为3418个bin marker。该图谱可以用于重要农艺性状的QTL定位,数量性状的遗传分析,分子标记辅助育种等方面。但该图谱还存在一定的局限性,比如17号染色体仅有11个bin标记,数量偏少;同时18号染色体的空隙有12.06cM,需要增加新的遗传标记来填补该空隙。因此,该图谱还需要添加更多的遗传标记,丰富其遗传标记密度,提高定位精度,从而使其更好的应用于重要农艺性状的定位研究。
3.2 始花节位的QTL定位分析
农作物植株的始花节位是一个重要农艺性状。在番茄中始花节位是由多基因控制的重要农艺数量性状,且与番茄的产量密切相关。Liu等[6]通过QTL定位分析发现了5个与始花节位密切相关的QTL位点。在辣椒中,辣椒植株主茎上始花节位是辣椒育种中评价果实成熟度的重要标准,且与开花时间密切相关。Zhang等[5]通过高密度遗传图谱的构建和QTL定位分析发现了与始花节位密切相关的2个主效QTL位点Ffn2.1和Ffn2.2。红麻始花节位与纤维产量密切相关。红麻开花后植株的生长由营养生长完全转变为生殖生长。此时红麻植株的生长主要集中在花序部分的生长,而韧皮部纤维的积累迅速下降。因此,红麻植株始花节位可以作为纤维产量育种的重要标志。本研究通过始花节位的表型数据结合高密度遗传连锁图谱,定位到了1个与始花节位密切相关的QTL位点QFFN6,该位点位于6号染色体。该位点的加性效应为-19.97,其遗传效应能够降低始花节位位数。在红麻中,QFFN6位点是发现的第一个与红麻始花节位密切相关的QTL位点。红麻始花节位是1个由多基因控制的数量性状,可能受到多个QTL位点的控制。本研究只鉴定到了1个QTL位点,这可能与所用定位的群体数量和遗传标记少有关。在今后的研究中,我们将进一步增加定位群体和遗传标记的数量,提高遗传图谱的密度和QTL定位的精确度,同时增加其在不同年份间的稳定性验证。
3.3 始花节位候选基因的鉴定
植物碱性螺旋-环-螺旋(Basic helix-loop-helix,bHLH)转录因子是植物重要的一类转录因子。bHLH家族成员通过结合调控靶基因的启动子,进而调控植物的生长发育和代谢过程,很多bHLH家族成员被验证参与植物开花调控。在拟南芥中短日照条件下AtbHLH193通过赤霉素途径促进拟南芥开花[21],AtbHLH113通过与SPL9在蛋白质水平上的互作调控开花相关基因(如SOC1、FT)的表达,从而影响拟南芥植株开花时间[22]。拟南芥bHLH家族中开花BHLH1(FBH1)、FBH2、FBH3和FBH4通过与CO基因启动子的E-box结合,从而激活该基因的表达。过表达4个FBH基因都能够提高CO基因表达水平,导致拟南芥植株提前开花[23]。在甘蓝中通过抑制bHLH家族中转录因子TT8-WD40的功能,提高开花基因SOC1和春化关键基因VIN3的表达量,从而促进甘蓝提早开花[24]。宋艳红等[25]通过农杆菌介导法将森林草莓FvbHLH130基因转化拟南芥,发现该基因使转基因拟南芥植株提7d开花。MYB转录因子在调控植物开花过程中具有重要作用。拟南芥中转录因子基因AtMYB30在韧皮部强启动子SUC2控制下表达,长日照和短日照条件下都能够促进拟南芥植株提前开花[26]。菊花转录因子CmMYB2通过与锌指蛋白CmBBX24相互作用影响赤霉素的合成,从而调控菊花的开花时间[27]。鸡爪槭转录因子基因ApMYB306通过抑制拟南芥FT基因的表达,从而延迟转基因植株的开花时间[28]。本研究结果表明,在短日照条件下Hc-bHLH6和Hc-MYB6基因的表达量显著上调,促进红麻植株开花。推测这2个基因与红麻始花节位密切相关,可以作为红麻纤维产量育种的候选基因。
本研究构建了1张含有3418个bin标记的高密度遗传连锁图谱并定位了1个与始花节位密切相关的QTL位点。该位点位于第6号染色体,其物理区间位于50.41~51.56Mb,LOD值为8.66,表型贡献率为13.77%,加性效应值为-19.97,对始花节位具有负调控作用。根据基因注释信息和qRT-PCR结果筛选到了2个与红麻植株第一朵花节位密切相关的候选基因,分别是Hc-bHLH6和Hc-MYB6基因。短日照条件会促进红麻植株提前开花,同时短日照处理显著提高了Hc-bHLH6和Hc-MYB6基因的表达量,因此推测这2个基因与红麻始花节位密切相关。
参考文献:
[1]李辉,李德芳,邓勇,潘根,陈安国,赵立宁,唐慧娟.红麻海藻糖生物合成关键酶基因HcTPPJ的克隆及响应逆境的表达分析[J].作物学报,2020,46(12):1914—1922.doi:10.3724/SP.J.1006.2020.04006.Li H,Li D F,Deng Y,Pan G,Chen A G,Zhao L N,Tang H J.Cloning of the key enzyme gene HcTPPJ in trehalose biosynthesis of kenaf and its expression in response to abiotic stress in kenaf[J].Acta Agronomica Sinica,2020,46(12):1914—1922.
[2]李辉,康泽培,邱财生,戴志刚,邱化蛟.红麻WRKY基因家族鉴定及其盐胁迫下的表达分析[J].华北农学报,2022,37(4):71—81.doi:10.7668/hbnxb.20192961.Li H,Kang Z P,Qiu C S,Dai Z G,Qiu H J.Identification of kenaf WRKY gene family and its expression analysis under salt stress[J].Acta Agriculturae Boreali-Sinica,2022,37(4):71—81.
[3]李辉,李德芳,陈安国,唐慧娟,李建军,黄思齐,邓勇.红麻短日条件下自然群体有效株高与始花节位的相关和回归分析[J].中国麻业科学,2017,39(1):7—11.Li H,Li D F,Chen A G,Tang H J,Li J J,Huang S Q,Deng Y.Correlation and regression analysis of kenaf natural population between first flower node and effective plant height under short day-light[J].Plant Fiber Sciences in China,2017,39(1):7—11.
[4]Zhang X F,Wang G Y,Chen B,Du H S,Zhang F L,Zhang H Y,Wang Q,Geng S S.Candidate genes for first flower node identified in pepper using combined SLAF-seq and BSA[J].PLoS One,2018,13(3):e0194071.doi:10.1371/journal.pone.0194071.
[5]Zhang X F,Wang G Y,Dong T T,Chen B,Du H S,Li C B,Zhang F L,Zhang H Y,Xu Y,Wang Q,Geng S S.High-density genetic map construction and QTL mapping of first flower node in pepper(Capsicum annuum L.)[J].BMC Plant Biology,2019,19(1):167.doi:10.1186/s12870-019-1753-7.
[6]Liu Y,Zhou X J,Zhang J H,Li H Z,Zhuang T M,Yang R Q,Chen H Y.Bayesian analysis of interacting quantitative trait loci(QTL) for yield traits in tomato[J].African Journal of Biotechnology,2011,10(63):13719—13723.doi:10.5897/ajb11.1779.
[7]Zhang L W,Xu Y,Zhang X T,Ma X K,Zhang L L,Liao Z Y,Zhang Q,Wan X B,Cheng Y,Zhang J S,Li D X,Zhang L M,Xu J T,Tao A F,Lin L H,Fang P P,Chen S,Qi R ,Xu X M,Qi J M,Ming R.The genome of kenaf( Hibiscus cannabinus L.)provides insights into bast fibre and leaf shape biogenesis[J].Plant Biotechnology Journal,2020,18(8):1796—1809.doi:10.1111/pbi.13341.
[8]Li H,Chang L,Tang H J,Luan M B,Pan G,Huang S Q,Jiang H,Wu Y N,Chen A G.High-resolution genetic map construction and QTL analysis of important fiber traits in ke-naf using RAD-seq[J].South African Journal of Botany,2023,153:46—52.doi:10.1016/j.sajb.2022.12.016.
[9]Li H,Durbin R.Fast and accurate short read alignment with Burrows Wheeler transform[J].Bioinformatics,2009,25(14):1754—1760.doi:10.1093/bioinformatics/btp324.
[10]McKenna A,Hanna M,Banks E,Sivachenko A,Cibul-skis K,Kernytsky A,Garimella K,Altshuler D,Gabriel S,Daly M,DePristo M A.The genome analysis toolkit: a MapReduce framework for analyzing next-generation DNA sequencing data[J].Genome Research,2010,20(9):1297—1303.doi:10.1101/gr.107524.110.
[11]Broman K W,Wu H,Sen S',Churchill G A.R/qtl: QTL mapping in experimental crosses[J].Bioinformatics,2003,19(7):889—890.doi:10.1093/bioinformatics/btg112.
[12]黄缓缓,安洪周,李魁英,王延兵,谷亦,乔亚科,高增玉.玉米花丝花青苷显色性状的QTL定位[J].华北农学报,2024,39(4):31—36.doi:10.7668/hbnxb.20194658.Huang H H,An H Z,Li K Y,Wang Y B,Gu Y,Qiao Y K,Gao Z Y.QTL mapping of silk coloring characteristics of anthocyanins in maize[J].Acta Agriculturae Boreali-Sinica,2024,39(4):31—36.
[13]Fei J B,Lu J Y,Jiang Q P,Liu Z B,Yao D,Qu J,Liu S Y,Guan S Y,Ma Y Y.Maize plant architecture trait QTL mapping and candidate gene identification based on multiple environments and double populations[J].BMC Plant Biology,2022,22(1):110.doi:10.1186/s12870-022-03470-7.
[14]张肖肖,殷学贵,陆建农,黄冠荣,张柳琴,刘朝裕,林海虹,左金鹰.蓖麻收获指数相关性状QTL定位及候选基因分析[J].华北农学报,2024,39(3):77—87.doi:10.7668/hbnxb.20194641.Zhang X X,Yin X G,Lu J N,Huang G R ,Zhang L Q,Liu C Y,Lin H H,Zuo J Y.QTL mapping and candidate gene analysis for harvest index and related traits in castor[J].Acta Agriculturae Boreali-Sinica,2024,39(3):77—87.
[15]Sanagi M,Aoyama S,Kubo A,Lu Y,Sato Y,Ito S,Abe M,Mitsuda N,Ohme-Takagi M,Kiba T,Nakagami H,Rolland F,Yamaguchi J,Imaizumi T,Sato T.Low nitro-gen conditions accelerate flowering by modulating the phosphorylation state of FLOWERING BHLH 4 in Arabidopsis[J].Proceedings of the National Academy of Sciences of the United States of America,2021,118(19):e2022942118.doi:10.1073/pnas.2022942118.
[16]Liu S D,Wang X P,Li E Y,Douglas C J,Chen J G,Wang S C.R2R3 MYB transcription factor PtrMYB192 regulates flowering time in Arabidopsis by activating FLOWERING LOCUS C[J].Journal of Plant Biology,2013,56(4):243—250.doi:10.1007/s12374-013-0135-1.
[17]Liu Y W,Li X,Li K W,Liu H T,Lin C T.Multiple bHLH proteins form heterodimers to mediate CRY2-dependent regulation of flowering-time in Arabidopsis[J].PLoS Genetics,2013,9(10):e1003861.doi:10.1371/journal.pgen.1003861.
[18]Yin J,Chang X X,Kasuga T,Bui M,Reid M S,Jiang C Z.A basic helix-loop-helix transcription factor,PhFBH4,regulates flower senescence by modulating ethylene biosynthesis pathway in petunia[J].Horticulture Research,2015,2:15059.doi:10.1038/hortres.2015.59.
[19]段怡红,阎媛媛,陈丽婷,李青,张冬梅,孙正文,张艳,马峙英,王省芬.陆地棉开花时间相关基因Gh-MYB44的克隆与功能验证[J].中国农业科技导报,2020,22(12):29—38.doi:10.13304/j.nykjdb.2020.0386.Duan Y H,Yan Y Y,Chen L T,Li Q,Zhang D M,Sun Z W,Zhang Y,Ma Z Y,Wang X F.Cloning and functional analysis of GhMYB44 related to flowering time in Gossypium hirsutum[J].Journal of Agricultural Science and Technology,2020,22(12):29—38.
[20]Chen M X,Wei C L,Qi J M,Chen X B,Su J G,Li A Q,Tao A F,Wu W R. Genetic linkage map construction for kenaf using SRAP,ISSR and RAPD markers[J].Plant Breeding,2011,130(6):679—687.doi:10.1111/j.1439-0523.2011.01879.x.
[21]Sharma N,Xin R J,Kim D H,Sung S,Lange T,Huq E.NO FLOWERING IN SHORT DAY(NFL) is a bHLH transcription factor that promotes flowering specifically under short-day conditions in Arabidopsis[J].Development,2016,143(4):682—690.doi:10.1242/dev.128595.
[22]宋建辉.bHLH113调控拟南芥开花和花青素合成的分子机制研究[D].杭州:浙江农林大学,2020.Song J H.The molecular regulatory mechanism of flowering and anthocyanin by bHLH113 in Arabidopsis[D].Hangzhou: Zhejiang A&F University,2020.
[23]Ito S,Song Y H,Josephson-Day A R,Miller R J,Breton G,Olmstead R G,Imaizumi T.FLOWERING BHLH transcriptional activators control expression of the photo-periodic flowering regulator CONSTANS in Arabidopsis[J].Proceedings of the National Academy of Sciences of the United States of America,2012,109:3582—3587.doi:10.1073/pnas.1118876109.
[24]周雯.甘蓝TT8及WD40转录因子影响花青素合成及开花时间的研究[D].重庆:西南大学,2017.Zhou W.Effects of TT8 and WD40 transcription factors on anthocyanin biosynthesis and flowering time in Brassi ca oleracea L.[D].Chongqing:Southwest University,2017.
[25]宋艳红,陈亚铎,张晓玉,宋盼,刘丽锋,李刚,赵霞,周厚成.森林草莓FvbHLH130转录因子调控植株提前开花[J].园艺学报,2023,50(2):295—306.doi:10.16420/j.issn.0513-353x.2021-1246.Song Y H,Chen Y D,Zhang X Y,Song P,Liu L F,Li G,Zhao X,Zhou H C.The transcription factor FvbHLH130 activates flowering in Fragaria vesca[J].Acta Horticulturae Sinica,2023,50(2):295—306.
[26]Liu L Y,Zhang J,Adrian J,Gissot L,Coupland G,Yu D Q,Turck F.Elevated levels of MYB30 in the phloem accelerate flowering in Arabidopsis through the regulation of FLOWERING LOCUS T[J].PLoS One,2014,9(2):e89799.doi:10.1371/journal.pone.0089799.
[27]Zhu L,Guan Y X,Liu Y N,Zhang Z H,Jaffar M A,Song A P,Chen S M,Jiang J F,Chen F D.Regulation of flowering time in chrysanthemum by the R2R3 MYB transcription factor CmMYB2 is associated with changes in gibberellin metabolism[J].Horticulture Research,2020,7:96.doi:10.1038/s41438-020-0317-1.
[28]朱璐,李淑顺,闻婧,马秋月,颜坤元,任杰,李倩中.鸡爪槭ApMYB306基因克隆及功能分析[J].江苏农业学报,2020,36(6):1512—1520.doi:10.3969/j.issn.1000-4440.2020.06.022.Zhu L,Li S S,Wen J,Ma Q Y,Yan K Y,Ren J,Li Q Z.Cloning and functional analysis of ApMYB306 gene in Acer palmatum[J].Jiangsu Journal of Agricultural Sciences,2020,36(6):1512—1520.
文章摘自:李辉,陈安国,唐慧娟,栾明宝.红麻始花节位QTL定位及候选基因分析[N].华北农学报,2025-08-06.doi:10.7668/hbnxb.20196050