摘 要:为丰富亚麻种质资源,了解资源遗传背景,选育优质亚麻新品种。本试验利用形态标记和分子标记(SSR)对387份亚麻种质资源的9个农艺性状及6个品质性状进行遗传多样性分析、聚类分析、主成分分析以及群体结构分析。农艺性状遗传多样性指数为0.70~2.08,变异系数在6.30%~1691.12%之间。品质性状遗传多样性指数在1.83~2.08之间,变异系数在0.07%~1.8%之间。试验共扩增出365个SSR位点,平均每对引物12.17个;有效等位基因数为1.2168~1.8320,引物PIC为0.2489~0.6257,遗传多样性指数为1.07~2.08;在遗传相似系数为0.66处可以将387份亚麻种质资源分为5个种群,农艺性状以及品质聚类分析均分为5个类群。本试验为呼和浩特地区的亚麻育种工作提供了理论依据。关键词亚麻;遗传多样性;表型性状
亚麻(LinumusitatissimumL.)栽培历史悠久,早在7000年前的新石器时代就有驯化栽培的记载[1],最新考古发现30000年前的旧石器时期人类就开始利用亚麻[2]。亚麻籽富含人体必须亚麻酸(ALA)和木酚素,同时还提供人体必须的食用纤维和蛋白质,是广受人们喜爱的功能食品[3]。
长期单一的传统育种方式使亚麻遗传基础越来越狭窄,不利于产量和品质的提高[4],因此需充分研究亚麻种质资源的遗传多样性和群体结构,以此扩大杂交亲本的选择范围和多样性,提高选育水平,选育出产量高、品质好、抗性强的优良品系。
遗传多样性代表作物进化过程中适应环境改变的能力[5],最初鉴定亚麻遗传多样性的方法是采用形态指标[6]和同工酶标记[7]。应用基于DNA分子标记技术来研究亚麻多样性的方法首先由Oh等提出[8],标记的方式主要有RAPD、AFLP、ISSR、SSR等[9-15]。简单重复序列标记(SSR)是串联重复的短序列,具有多态性丰富、拷贝多、覆盖面广、完全显性遗传等特点,更适合用于植物遗传变异的研究[15]。内蒙古农牧业科学院亚麻课题组“十二五”期间收集保存亚麻种质资源材料1078份,其中国外引种200份,内蒙古呼和浩特、鄂尔多斯、锡林郭勒、乌兰察布等盟市共503份,黑龙江40份,甘肃兰州、定西、张掖、平凉共97份,青海58份,山西大同86份,宁夏固原23份,新疆伊犁58份,河北张家口13份[16]。高凤云等对其进行了粗脂肪及脂肪酸组成成分的SSR关联分析[17];伊六喜对其进行了SRAP多样性分析[18]。本研究在前期研究的基础上,从收集保存的资源材料中,根据引种地区、生态类型及表型特征等筛选出符合育种目标的387份材料,采用形态指标与SSR分子标记的方法进行种质资源遗传多样性及群体结构分析,并对其进行主成分分析和聚类分析,根据亚麻种质资源基因型及表型变异范围,构建亚麻核心种质群体,为亚麻育种提供优质亲本材料。
1 材料与方法
1.1 材料的种植
2019年4月下旬在内蒙古农牧业科学院试验田种植供试种质资源材料,共计387份,每份材料种植在2m2小区,5行区种植,行长1m,行距20cm,2次重复。
1.2 农艺性及品质数据采集
根据《亚麻种质资源描述规范和数据标准》,调查并统计生育期,成熟后每小区随机选取20株进行株高、工艺长度、主茎分枝数、单株果数、每果粒数、千粒重、单株粒重等农艺性状的测量,并记录小区总产量。品质数据则采用DA7200型近红外仪测定。
1.3 亚麻基因组DNA的提取及检测
用CTAB法提取DNA,操作步骤如下:1)苗期取1g左右亚麻材料叶片置于2mL离心管中,加裂解液后用TissueLyser组织研磨机磨至粉状,65℃水浴裂解1h。2)加700μL氯仿:异戊醇(24:1)混匀后10000转/min离心5min。3)抽取上清液于1.5mL灭菌离心管中,加入约700μL无水乙醇,缓慢混匀至百色絮状DNA出现,-20℃冰箱中静置1h,12000转/min离心5min。4)去上清,DNA沉淀用70%乙醇冲洗2次,于超净台上干燥后加100μLTE溶解。采用1%的琼脂糖凝胶电泳和紫外分光光度计法检测DNA的纯度和浓度。
1.4 引物设计及PCR扩增
本试验所使用的248对引物序列是根据已有亚麻SSR标记设计所得,最终委托金斯瑞生物科技有限公司合成。PCR扩增产物经6%非变性聚丙烯酰胺凝胶电泳分离、银染、显色后运用人工读带的方式记录电泳图谱的多态性条带,同一位置出现清晰重复条带记为“1”,未出现条带记为“0”,从而生成由“1”和“0”组成的原始矩阵,据此计算多态性位点数、多态性位点百分率(P)等遗传系数。
1.5 数据处理
采用Excel计算各性状最大值、最小值、平均值、变异系数。用R对试验数据进行方差分析、相关性分析、主成分分析、聚类分析;用PopgenVer1.32软件进行基于Nei1973遗传距离的非加权组平均法(UPGMA)聚类分析;采用STRUCTURE2.2软件进行群体结构分析。遗传多样性指数采用ShannonWeaver法,计算公式为:H′=-∑PilnPi,式中Pi表示第i级别内材料份数占总份数的百分比。对数量性状进行分级处理。其中1级<X-2δ,10级≥X+2δ,中间每0.5δ间隔一级,X为平均值,δ则为标准差。
2 结果与分析
2.1 农艺性状的变异分析
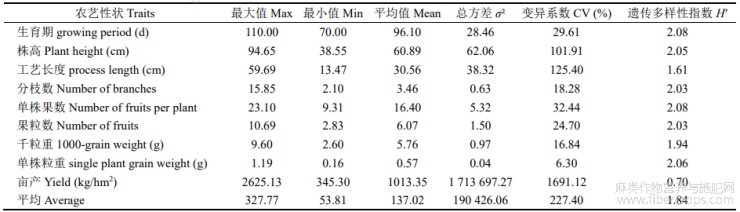
对亚麻种质资源的9个农艺学性状进行遗传多样性分析(表1),结果表明:各农艺性状的遗传多样性指数在0.70~2.08之间,平均为1.84,其中生育期和单株果数的遗传多样性指数最高,均达到2.08,亩产遗传多样性指数最低,说明387份亚麻种质资源的生育期和单株果数遗传丰富度较高,亩产遗传丰富度较低。对亚麻种质资源进行变异系数分析发现:亩产的变异系数最大为1691.12%,千粒重变异系数最小为6.30%,平均为227.40%。由此说明亚麻种质资源间的遗传差异明显,变异幅度大,遗传丰富度高。
表1 农艺性状的差异统计
2.2 农艺性状相关分析
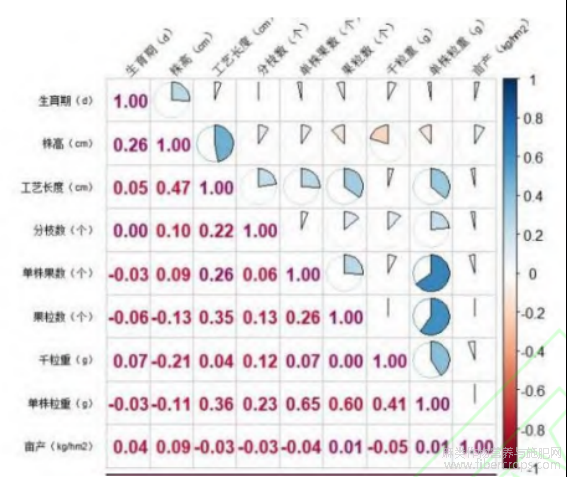
对亚麻种质资源的9个农艺性状进行相关性分析(图1),35对相关性状中有13对极显著,3对显著,19对不显著。其中株高分别与工艺长度、分枝数、单株果数、果粒数以及单株生产力呈极显著正相关。工艺长度与生育期呈极显著正相关,与千粒重呈极显著负相关与果粒数和单株生产力呈负相关。单株生产力与分枝数、单株果数、果粒数以及千粒重极显著正相关。分枝数分别与果粒数以及千粒重呈极显著正相关。综上所述,株高是影响产量的主要因素之一。
扇形的面积代表相关性大小,蓝色为正相关,颜色越深越趋近于1;红色为负相关,颜色越深越趋近于-1。下同。
图1 农艺性状的相关性分析
2.3 品质性状变异分析
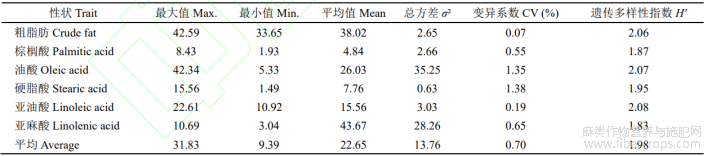
对387份亚麻种质资源的品质性状进行遗传多样性分析(表2),结果表明6个品质性状的遗传多样性指数在1.83~2.08之间,平均为1.98,其中亚油酸的遗传多样性指数最高,亚麻酸的遗传多样性指数最低,说明387份亚麻种质资源的亚油酸的遗传丰富度较高,亚麻酸的遗传丰富度较低。对亚麻种质资源进行变异系数分析发现:硬脂酸的变异系数最大为1.38%,粗脂肪变异系数最小为0.07%,平均为0.70%。由此说明亚麻种质资源间的遗传差异明显,变异幅度大,遗传丰富度高。
表2 品质性状的差异统计
2.4 品质性状相关性分析
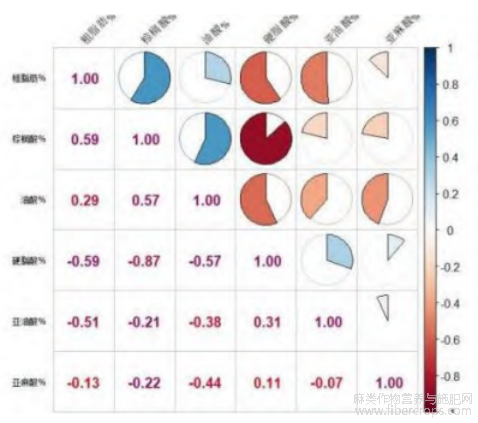
对387份亚麻种质资源的6种脂肪酸含量进行相关性分析(图2),15对相关性状中有2对极显著正相关,4对呈极显著负相关,1对正相关,1对负相关以及7对不显著。其中粗脂肪与棕榈酸呈极显著正相关,与硬脂酸和亚油酸成极显著负相关。棕榈酸与油酸呈极显著正相关,与硬脂酸呈极显著负相关;油酸与硬脂酸呈极显著负相关,与亚油酸以及亚麻酸成负相关;亚油酸和硬脂酸呈正相关;由此可知,在以含油率(粗脂肪含量)为育种目标是可以间接测量棕榈酸含量来辅助育种。同时棕榈酸与硬脂酸呈极显著负相关,这一指标也有待进一步应用到育种上。
图2 品质状的相关性分析
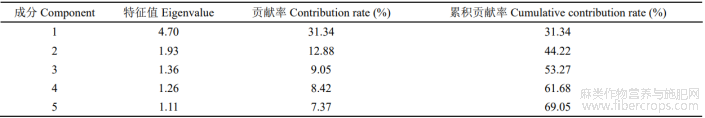
2.5 农艺性状及品质性状主成分分析
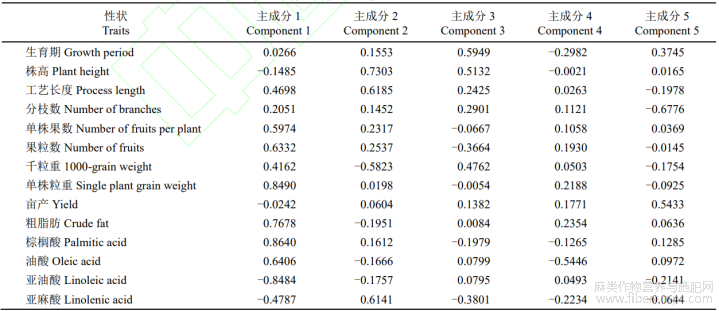
主成分分析通过线性变换或者舍弃一小部分数据的方式来展现作物各性状间起主导作用的综合指标。表3为387份亚麻种质资源农艺性状与品质性状主成分结果。由表4可知15个性状共计提取出5个特征值大于1的主成分。排名前五的主成分贡献率分别为:31.34%、12.88%、9.05%、8.42%以及7.37%,累积贡献率为69.05%。
第一主成分的特征值为4.70,其中棕榈酸的载荷最高且为正值。硬脂酸的载荷较高,工艺长度的载荷最低,由此推断第一主成分主要反映棕榈酸载荷。
第二主成分的特征值为1.93,其中株高载荷最大为0.7303,最低是千粒重-0.5823,由此推断第二主成分主要反映株高载荷。
第三主成分的特征值为1.36,生育期的载荷最高为0.5949由此推断第三主成分主要体现生育期载荷。
第四主成分的特征值为1.26,粗脂肪的载荷最高,为0.2354,由此推断第四主成分主要体现粗脂肪载荷。
第五主成分的特征值为1.11,亩产的载荷最高,为0.5433,由此推断第五主成分主要体现亩产载荷。
表3 各农艺性状主成分分析的特征向量
表4 各农艺性状主成分分析的特征值及贡献率
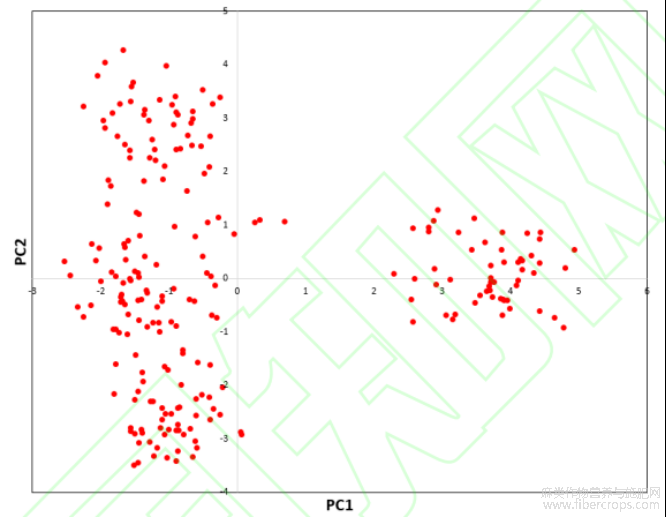
以第1主成分作横坐标,以第2主成分为纵坐标对387份种质资源分别进行散点分布作图(图3),结果表明:有56份种质资源材料第一主成分特征值较大,在产量及相关性状方面表现良好;第2主成分呈连续性分布,但产量性状表现良好的种质资源材料的第二主成分值在1和-1之间,可见要提高产量,在育种过程中就不能选择株高过高的材料。
图3 亚麻种质资源第1、2主成分二维排序图
2.6 农艺性状聚类分析
对供试材料的农艺性进行聚类分析(图4),387份亚麻种质资源被分为5个类群。
第I群体包含3份材料,该群体的平均株高(87.14cm)和平均工艺长度(56.23cm)明显高于其他材料,平均千粒重(4.73g)低于其它材料。通过田间观察发现,该群体可作为纤维亚麻的骨干亲本材料进行育种研究。
第II群体包含2份材料,该群体的生育期、株高、工艺长度、主茎分枝数、单株果数、每果粒数、千粒重以及单株粒重平均值分别为:80.50d、39.23cm、17.36cm、2.57个、11.61个、5.03粒、5.30g、0.26g,低于其他亚麻种质资源。以降低株高、适应机械化为育种目标时,可选用该群的材料作为父母本进行组配。
第III群体包含9份材料,该群体平均生育期76.67d,是生育期最短的材料。9份材料的平均主茎分枝数和平均单株果数居所有类群的第一位,分别为3.64个和16.85个。主茎分枝数和单株有效果数是影响产量的主要原因,结合生育期表现,该群体更能适应无霜期较短的地区。因此在无霜期短的的地区,以改良主茎分枝数和单株果数为育种目标可选用该群体的材料进行育种研究。
第IV群体包含43份材料,该群体的平均单株果数、平均千粒重以及平均单株粒重均为最高,分别为6.31个、5.88g、0.59g。该群体的平均生育期94.25d,为呼和浩特地区亚麻生长的正常周期。单株果数、千粒重以及单株粒重都是影响产量的重要性状,该群体结合第III群体可作为育种骨干亲本,通过轮回选择法选育高产、早熟的亚麻新品种。
第V群体包含330份材料,该群体的平均生育期最长,为96.89d,其他性状居中。该群体可作为常规材料进行亚麻的遗传改良。
图4 农艺性状的聚类分析图
2.7 品质性状聚类分析
对387份亚麻种质资源的6种脂肪酸含量进行了聚类分析(图5),被分为5个类群。
第I群体包含371份亚麻资源材料,该群体的平均油酸含量(26.33%)高于其他类群材料,硬脂酸含量7.82%,处于中等偏上水平,其他性状表现居中。油酸为单不饱和脂肪酸较多不饱和脂肪酸来说,更加耐储藏。因此选择该类群作为育种材料,可改善亚麻油的耐储时间。
第II群体有2份亚麻资源材料,该群体平均含油率(36.55%)处于中等偏下水平,但棕榈酸(7.28%)和亚油酸(22.38%)高于其他类群。同时油酸(23.43%)、硬脂酸(7.72%)、亚麻酸(45.27%)含量处于中等偏上水平。该类群可作为轮回亲本。
第III群体共有5份亚麻资源材料,该类群的平均硬脂酸含量(11.88%)较其他类群高,亚麻酸含量48.06%处于中等偏上水平,粗脂肪酸含量最低34.41%。因此该类群的材料适合作为非轮回亲本,或粗脂肪的精细定位试验低混池亲本。
第IV群体有4份亚麻资源材料,该类群的粗脂肪含量(41.17%)以及亚麻酸含量(55.85%)均为最高,同时该类群的油酸含量(11.89%)最低。该类群可作为品质改良骨干亲本。
第V群体有5份亚麻资源材料,该群体的粗脂肪含量(40.81%)、棕榈酸含量(7.87%)、油酸(24.55%)以及亚油酸(19.10%)的含量中等偏上,亚麻酸含量最低为13.52%。该类群可作为亚麻酸精细定位种低混池目标亲本。
图5 品质状的聚类分析图
2.8 SSR多态性分析
2.8.1 SSR位点多态性分析
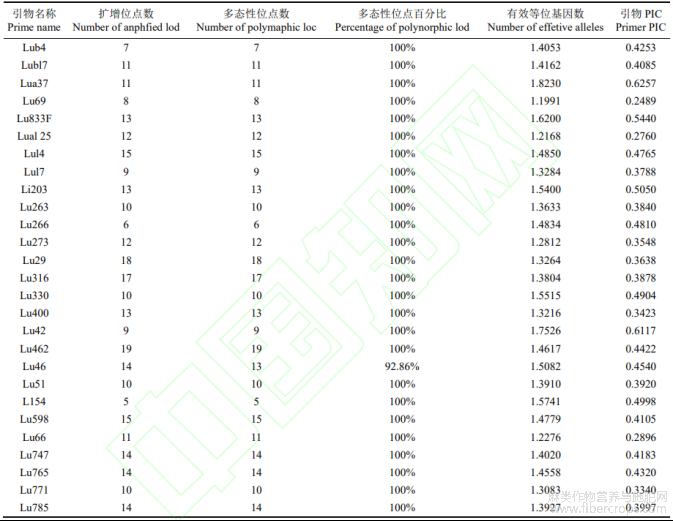
随机挑选8份(表5)地域、形态、遗传背景差异较大的亚麻种质资源为试验材料,对248对SSR引物进行初筛,最终筛选出30对条带清晰、多态性好的核心引物用于387份亚麻材料的遗传多样性分析。试验共扩增出365个位点,平均每对引物扩增出12.17个位点,平均扩增多态位点百分率为99.76%;有效等位基因数为1.2168~1.8320,引物PIC为0.2489~0.6257(表6)。由此说明此次筛选的引物多态性丰富、鉴别差异性强。其中引物Lua37有效等位基因数目以及引物的PIC值均最高,因此在今后的遗传多样性分析中可作为核心骨干引物。
表5 不同区域、形态、遗传背景的亚麻种质资源
表6 30对SSR引物在387份亚麻材料中的扩增结果
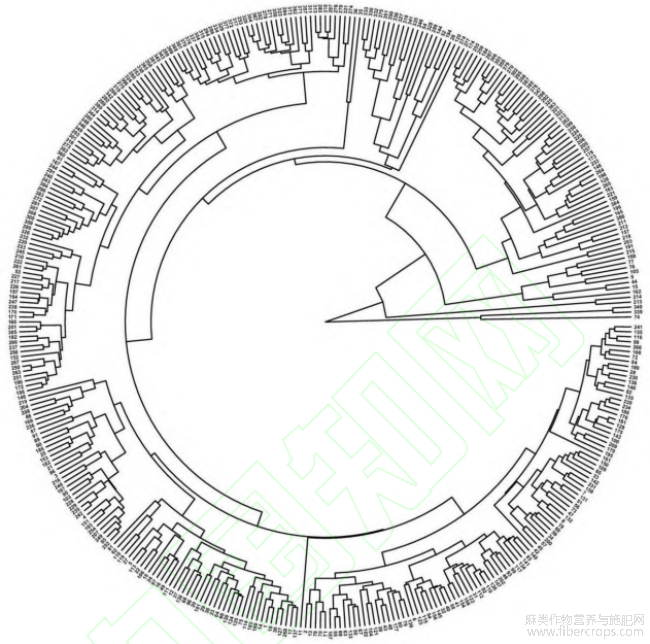
2.8.2 UPGMA聚类分析
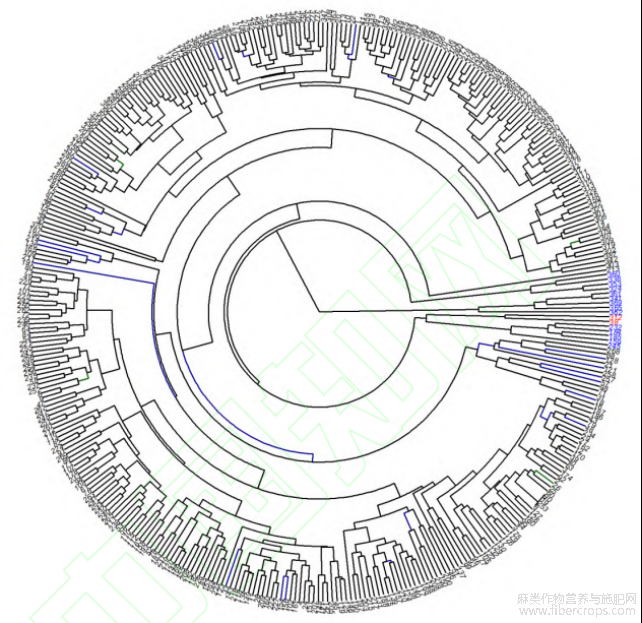
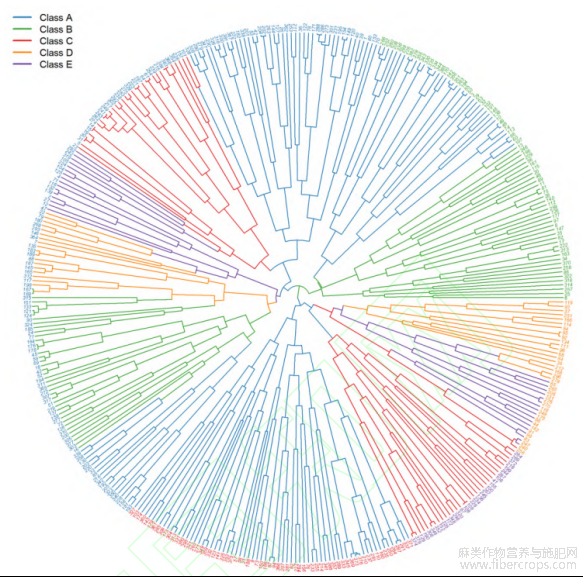
UPGMA是通过计算每一对簇的算数平方来展示数据间的层次关系,是遗传多样性研究中常用的分析方法。387份亚麻种质资源(图6)的相似系数为0.655~0.912,在遗传相似系数为0.66处可以将387份亚麻材料分为5个种群。种群1包含84份亚麻种质资源,种群2包括43份材料,种群3包含68份材料,种群4包含107份材料,种群5包含85份材料。
图6 聚类分析图
2.8.3 群体结构分析
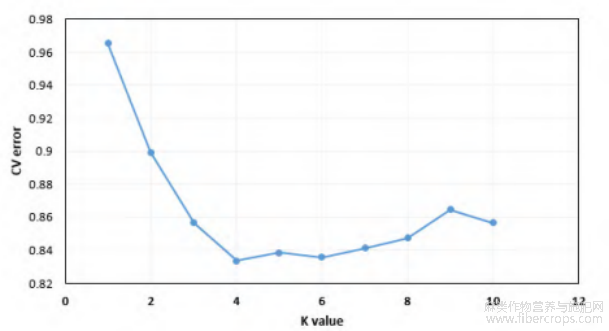
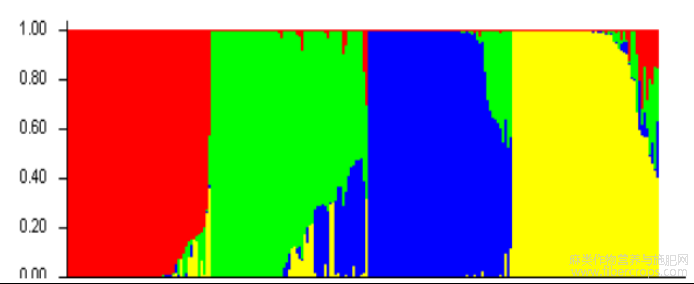
当K=4时出现拐点,以此为依据可将387份亚麻种质资源分为4个种群(图7),种群1和种群3都是97份种质资源,种群2为90份种质资源,种群4为96份种质资源;从图8可看出,种质资源材料群体间基因混杂程度不高,说明亚麻遗传背景丰富,群体构建合理,有利于不同育种目标亲本材料的选择。
图7 ΔK随K值的变化曲线
图8 群体结构分析图
3 讨论
3.1 基于形态学标记的遗传多样性分析
形态学标记是了解作物遗传背景最直接有效的方式,一般通过对花色等质量性状的观测以及株高、主茎分枝数、单株粒数等数量性状的测量,就可对植物进行分类。该方法操作简单,受外界因素(天气、人为)影响较大。在遗传多样性分析时,常因标记性状少,或数量性状和质量性状界定不清,造成试验数据不精确[19]。因此该方法一般只用于初步的遗传多样性分析。本次试验总共标记了9个农艺性状,基本涵盖了亚麻育种的目标性状。此次标记的性状与关虎等[20]人和李恭泽等[21]标记的基本吻合,但少于前人[22-23]标记的数目。因此在后续的试验中可继续增加花色、叶色以及茎秆粗度等易标记性状,使试验结果更加精准。
3.2 基于SSR分子标记的遗传多样性分析
简单重复序列(SSR)因其操作简单、经济实惠、等位基因呈现共显性的特点,一直被人们应用于的作物遗传多样性分析中[19]。引物扩增的多态性位点、有效等位基因数以及PIC的高低,可直接反应引物的好坏。本次试验对248对引物进行初筛最终筛选出30对核心引物。其中Lua37、Lu833F、Li203、Lu42的PIC高于0.5[24],说明引物多态性程度高,这与李恭泽[21]研究结果基本吻合。
3.3 亚麻种质资源群体结构分析
基于SSR标记位点的UPGMA聚类分析将387份亚麻种质资源大致分为5个群体,前4个种群中绝大部分为国内种质资源、少量国外种质资源,种群5全部为国外种质资源,各个群体组成相差不大。农艺性状聚类分析也分为5个群体,但各群体间在组成上存在较大差异,其中V群体包含330份种质资源,I、II、III群体分别只有3、2、9份种质资源,可见环境条件对亚麻农艺性状的影响巨大。从群体结构分析图可看出存在部分混杂,但总体上能聚类各个群体,有利于在各群体中挑选优质单株构建核心种质库。
4 结论
本试验利用形态标记和分子标记(SSR)的方法对387份亚麻种质资源进行多样性和群体结构分析。形态学结果显示性状间的遗传多样性指数在0.70~2.08,平均1.88,变异系数在6.30%~1691.12%。品质性状遗传多样性分析指数在1.83~2.08,变异系数在0.07%~1.38%之间。分子标记结果显示有效等位基因数为1.2168~1.8320,引物PIC为0.2489~0.6257,遗传多样性指数为1.07~2.08。由此说明387份种质资源间的遗传差异明显,变异幅度大,遗传丰富度高。同时筛选出了4对高多态性引物,可作为核心引物用于之后的遗传多样性研究。387份亚麻种质资源被分为5个类群,前4个类群主要为国内资源,第5个类群为国外资源。因此在今后的品种改良、新品种选育以及核心种质资源库的构建,应尽可能规避在相同类群中进行选择。
参考文献
[1]HarrisRBDR.Domesticationofplantsintheoldworld:TheoriginandspreadofcultivatedplantsinWestAsia,EuropeandtheNileValleybyDanielZohary;MariaHopf.TheAgriculturalHistoryReview,2001,49(2):226-227.
[2]KvavadzeE,Bar-YosefO,Belfer-CohenA,etal.30,000-year-oldwildflaxfibers.Science,2009,325(5946):1359-1359.
[3]BassettCMC,Rodriguez-LeyvaD,PierceGN.Experimentalandclinicalresearchfindingsonthecardiovascularbenefitsofconsumingflaxseed.PhysiologieAppliquéeNutritionEtMétabolisme,2009,34(5):965.
[4]RagupathyR,RathinaveluR,CloutierS.PhysicalmappingandBAC-endsequenceanalysisprovideinitialinsightsintotheflax(LinumusitatissimumL.)genome.BmcGenomics,2011,12(1):217.
[5]RandsMRW,AdamsWM,BennunL,etal.BiodiversityConservation:ChallengesBeyond2010[J].Science,2010,329(5997):1298-1303.
[6]DiederichsenA.Comparisonofgeneticdiversityofflax(LinumusitatissimumL.)betweenCanadiancultivarsandaworldcollection.PlantBreeding,2001,120(4):360-362.
[7]MånsbyE,O.Díaz,BothmerRV.PreliminarystudyofgeneticdiversityinSwedishflax(Linumusitatissimum).GeneticResources&CropEvolution,2000,47(4):417-424.
[8]OhTJ,GormanM,CullisCA.RFLPandRAPDmappinginflax(Linumusitatissimum).Theoretical&AppliedGenetics,2000,101(4):590-593.
[9]FuYB,DiederichsenA,RichardsKW,PetersonG.Geneticdiversitywithinarangeofcultivarsandlandracesofflax(LinumusitatissimumL)asrevealedbyRAPDs.GenetResourCropEvol,49(2):167-174
[10]FuYB,RowlandGG,DuguidSD,etal.RAPDAnalysisof54NorthAmericanFlaxCultivars.CropScience,2003,43(4):1510.
[11]DiederichsenA,FuYB.PhenotypicandMolecular(RAPD)DifferentiationofFourInfraspecificGroupsofCultivatedFlax(LinumusitatissimumL.subsp.usitatissimum).GeneticResources&CropEvolution,2006,53(1):77-90
[12]SpielmeyerW,GreenAG,BittisnichD,etal.IdentificationofquantitativetraitlocicontributingtoFusariumwiltresistanceonanAFLPlinkagemapofflax(Linumusitatissimum).Theoretical&AppliedGenetics,1998,97(4):633-641.
[13]RiekJD,LooseMD,WaesJV,etal.Mostsimilarvarietygroupingfordistinctnessevaluationofflaxandlinseed(LinumusitatissimumL.)varietiesbymeansofAFLPandmorphologicaldata.PlantVarieties&Seeds,2001,14(2):69-87.
[14]HüseyinU,FuYB,KurtO,etal.Geneticdiversityofcultivatedflax(LinumusitatissimumL.)anditswildprogenitorpaleflax(LinumbienneMill.)asrevealedbyISSRmarkers.GeneticResources&CropEvolution,2010,57(7):1109-1119.
[15]PowellW,MachrayGC,ProvanJ.Polymorphismrevealedbysimplesequencerepeats.TrendsinPlantScience,1996,1(7):215-222.
[16]党占海,赵伟.中国现代农业产业可持续发展战略研究-胡麻分册.北京:中国农业出版社,2016.
[17]高凤云,斯钦巴特尔,周宇,等.基于SSR标记的胡麻粗脂肪及脂肪酸组分的关联分析.作物杂志,2022(1):44-49.
[18]伊六喜.胡麻产量和品质相关性状的全基因组关联分析.呼和浩特:内蒙古农业大学,2018.
[19]何瑞超.绿豆遗传多样性研究及种子萌发期耐盐性评价.呼和浩特:内蒙古农业大学,2021.
[20]关虎,王振华,曹禹,等.亚麻品种主要农艺性状遗传多样性分析.新疆农业科学,2011,48(11):2035-2040.
[21]李恭泽,郭栋良,江海霞,等.379份亚麻种质白粉病抗性遗传多样性分析.分子植物育种,1-21.
[22]郭栋良,江海霞,张喻,等.国外引进亚麻种质资源遗传多样性分析.新疆农业科学,2019,56(11):2112-2122.
[23]左振兴,纪军建,付国庆,等.基于DUS测试性状的亚麻测试品种遗传多样性分析.中国农学通报,2021,37(24):48-53.
[24]SharmaP,MehtaG,ShefalI,etal.Developmentandvalidationofheat-responsivecandidategeneandmiRNAgenebasedSSRmarkerstoanalysisgeneticdiversityinwheatforheattolerancebreeding.MolBiolRep,2021,48(1):381-393.
文章摘自:高凤云,伊六喜,周宇,等.亚麻种质资源遗传多样性分析[J/OL].作物杂志,1-11[2025-03-03].http://kns.cnki.net/kcms/detail/11.1808.S.20250217.0952.002.html.