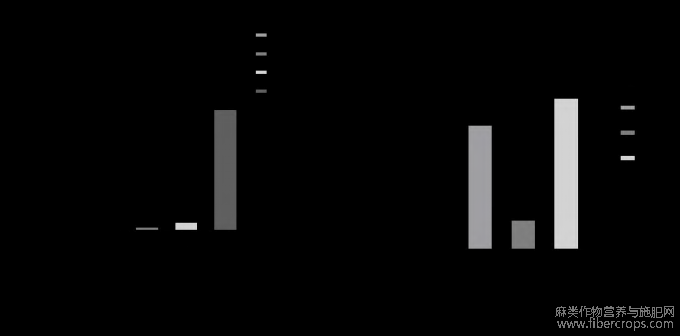摘 要:MYB类转录因子KAN4(KANADI4)在红麻类黄酮合成和纤维发育中发挥着重要作用。本研究以红麻品种‘福红952’为材料,对HcKAN4基因进行克隆和表达模式分析,探讨TRV-VIGS诱导该基因沉默对类黄酮合成途径中关键酶基因表达量改变的影响。基因克隆显示,HcKAN4基因开放阅读框(open reading frame,ORF)全长为966bp,编码322个氨基酸,包含一个MYB保守结构域。进化树分析发现,其与拟南芥和木槿KAN4s的亲缘关系较近。表达分析表明,该基因在植物不同组织中均有表达,且转录水平随着植物的生长递增。VIGS诱导基因沉默显示,6株HcKAN4的转录水平显著下调,达到基因沉默效果。进一步实时荧光定量PCR检测发现,类黄酮合成相关基因HcCHS?HcF3’5’H?HcANS?HcANR的转录水平显著下调,分别是对照组的0.51?0.14?0.23?0.11倍,表明HcKAN4基因可调控红麻类黄酮生物合成相关基因。这些结果为阐明红麻MYB转录因子调控类黄酮合成提供了依据,同时为改善纤维品质提供了研究思路。
关键词:红麻;HcKAN4;类黄酮合成;VIGS
红麻(Hibiscus cannabinus L.)为锦葵科木槿属的一年生常异花授粉纤维作物,属于短日照喜温类型,主要种植在亚洲的中国?印度?孟加拉?马来西亚以及非洲的马里?贝宁?赞比亚等国家。红麻属于韧皮部纤维作物,具有质地柔软?吸湿性强?透气性好?抑菌?易降解等优良特性。在建材?可降解塑料?吸污?饲料?汽车内衬和药用等领域都有广泛的应用。
类黄酮化合物是植物中具有高度活性的次生代谢产物,在植物抗氧化?抗病虫害等方面具有良好效果,原花青素(proanthocyanidins,PAs)是在植物中广泛存在的一种类黄酮化合物,能够决定植物器官颜色,并在应对生物和非生物胁迫中发挥重要作用。同时,花青素可以有效清除自由基,在抗氧化?抗衰老方面效果显著。因此深入了解类黄酮在红麻中的合成及调控过程,对于培育彩色纤维?增强抗逆性?提高纤维品质具有重要作用。
类黄酮生物合成途径已被广泛研究,参与其生物合成的结构基因己相继被克隆和鉴定。类黄酮生物合成通路错综复杂,其中先后涉及到十几个关键酶(PAL?C4H?4CL?CHS?CHI?F3H?F3’H?DFR\LDOX?ANS?LAR?ANR?LAC等)的催化反应。此外,类黄酮代谢的转录调控也被广泛研究,涉及到多种转录因子(WIP-ZF?MYB?bHLH?WD40?WRKY?MADS),其中以MYB?bHLH和WD40最为常见,这3类转录因子可以以MYB-bHLH-WD40(MBW)复合体的形式来调控植物PAs的生物合成,也可以单独发挥作用,例如Ding等发现苹果的转录因子MdMYB28可以负调控花青素的合成。MYB转录因子决定MBW复合体的特异性和被激活的基因,是复合体的核心成员,可通过过表达MYB就会明显促进PAs的生物合成。
在许多植物中,MYB转录因子均已被报道参与类黄酮代谢的调控。例如,在拟南芥(Arabidopsis thaliana)中,MYB 类转录因子 TT2/AtMYB123 通过调节ANR?DFR和AHA10的表达来特异调控种皮原花青素的积累。在水稻(Oryza sativa L.)中,MYB转录因子OsPL通过调控花青素代谢使种子耐热性增加。在苜蓿 (Medicago truncatula)中,MtMYB5和MtMYB14通过与MtTT8和MtWD40-1相互作用形成复合体,从而激活ANR和LAR的表达来协同调控种子外壳原花青素的积累。MYB转录因子不同成员除了正调控类黄酮的合成外,也存在对其负调控。例如,在苦荞(Fagopyrum tataricum)中,MYB转录因子基因FtMYB11通过与FtSAD2或FtJAZ1的相互作用抑制苯丙烷的生物合成从而抑制类黄酮的合成。拟南芥中AtKAN4基因过表达产生了一个显性突变系sk21-D,突变体中的CHS?CHI?F3’H?DFR?LDOX?ANR等类黄酮合成相关基因的表达量均下降,表明AtKAN4基因可广泛调控拟南芥PAs生物合成相关基因。
病毒诱导的基因沉默(virus induced gene silencing,VIGS)是一种植物用来抵御入侵病毒的自然防御机制,属于转录后基因沉默(post-transcriptional gene silencing,PTGS)。利用VIGS技术能够快速鉴定目标基因沉默后的表型和基因转录水平变化,不需要通过稳定遗传转化。因此它在功能基因组学研究中有显著的优势,能快速地鉴定目的基因功能。VIGS的沉默效率受多种因素的影响,病毒载体的选择对沉默效率有着较大影响,目前植物中应用范围较广的是烟草脆裂病毒(Tobacco rattle virus,TRV),与其他病毒载体相比,TRV具有沉默效率高和时间长等优点,TRV-VIGS体系目前已在棉花?亚麻等多种寄主植物中应用。
与黄麻等韧皮部纤维作物相比,红麻纤维纤维素含量较低而木质素含量较高。据报道,有317个淀粉和糖代谢途径基因可能参与纤维素生物合成,包括CesA?Csls基因家族;根据相关研究发现,PAL?HCT?C4H?COMT?CAD?4CL和F3’H在木质素生物合成起关键作用。同时类黄酮代谢途径不仅可以独自影响纤维的发育,其还可能和木质素代谢途径或者与苯丙烷代谢途径的其他分支代谢途径共同地来调控纤维的发育。植物MYB转录因子调控类黄酮代谢和纤维发育的机制复杂多样,而在红麻中鲜有报道。本研究以红麻优良品种‘福红952’为材料,基于其全基因组测序数据,克隆获得一个与拟南芥AtKAN4的同源基因HcKAN4,通过Gateway技术构建HcKAN4的VIGS载体,获得基因沉默植株后结合实时荧光定量PCR分析其对红麻类黄酮合成途径相关基因的表达影响。这将丰富MYB转录因子KAN4在红麻类黄酮合成途径中的研究,为红麻类黄酮合成及调控的分子机制研究和提高纤维品质提供理论依据。
1 材料与方法
1.1 试验材料
本研究以红麻‘福红 952’为试验材料,由福建农林大学麻类遗传育种与综合利用实验室保存提供。大肠杆菌菌株DB3.1?TOP10?农杆菌菌株GV3101?Gateway入门载体pDONR207?载体pTRV1和pTRV2等均由福建农林大学麻类遗传育种与综合利用实验室保存。RNA提取试剂盒和实时荧光定量PCR试剂盒购自北京天根生化技术有限公司。
1.2 试验方法
1.2.1 RNA提取和cDNA合成
使用RNA提取试剂盒提取‘福红952’红麻叶片的总RNA。用超微量分光光度计NanoDropND-1000(NanoDrop,Wilmington,DE,USA)检测RNA样品浓度,并用1%琼脂糖凝胶电泳检测其完整性。用RNA反转录试剂盒将提取的总RNA反转录为cDNA,反转录体系为(5× Fastking-RT SuperMix: 4 μL?Total RNA 50 ng: 2 μg?RNase-Free ddH2O 补足到 20 μL),反应程序为42℃反应15min?95℃反应3min,以cDNA为模板进行红麻HcKAN4基因的克隆及实时荧光定量PCR分析。
1.2.2 红麻HcKAN4基因克隆
实验室前期基础发现HcKAN4基因对类黄酮合成有着调控作用。从已报道文献和TAIR数据库(https://www.arabidopsis.org/)获得拟南芥AtKAN4(AT5G42630)蛋白序列。利用AtKAN4氨基酸序列进行BLASTP比对分析,获得相似性较高的同源基因,命名为HcKAN4(红麻数据库编号:Hc.03G039160)。以红麻cDNA为模板设计特异引物克隆HcKAN4基因(附表1)。PCR程序为:95℃预变性2min;95℃变性45s,55℃退火45s,72℃延伸30s,34个循环;72℃延伸10min。PCR反应结束后将产物用1%琼脂糖凝胶电泳检测,使用DNA纯化试剂盒(Vazyme,南京)回收目的条带,送尚亚生物技术公司(福州)测序,并通过DNAMAN软件进行序列比对。
1.2.3红麻HcKAN4蛋白系统进化树构建及序列比对分析
在中国国家基因组科学数据中心(National Genomics Data Center)基因组数据库Genome Warehouse (https://ngdc.cncb.ac.cn/gwh)中进行BLASTP 比对,得到‘福红952’红麻KAN4基因的蛋白质序列,用MEGA11.0软件绘制系统进化树。使用NCBI-Blast在线分析获得不同物种KAN4基因的同源蛋白序列,通过多序列比对软件DNAMAN对红麻HcKAN4蛋白序列进行比对分析。
1.2.4 红麻HcKAN4基因表达分析
根据实验室前期建立了红麻‘福红952’的根?茎?叶和花等不同发育时期不同组织的转录组数据库,用R语言中的pheatmap函数对‘福红952’红麻HcKAN4基因的电子表达量绘制热图。
1.2.5 TRV-VIGS载体构建
以克隆获得的HcKAN4基因为模板,通过primer5.0软件设计位于HcKAN4基因CDS上约250bp特异性片段的引物HcKAN4-SF和HcKAN4-SR(附表1)。PCR反应结束电泳检测并回收目标条带,测序后检查序列是否正确。
pTRV2-HcKAN4载体构建:将扩增得到的HcKAN4基因特异性片段?载体pDONR207和BP酶按BP反应体系(HcKAN4-250bp(20ngμL-1):0.5μL,pDONR207vector(40ngμL-1):0.5μL,BPClonasereactionbuffer:0.25μL)混匀后,25℃过夜连接,转化到DB3.1感受态细胞中,然后涂布在含有50μgmL-1庆大霉素的LB平板上。过夜培养后挑取单克隆,进行菌液PCR检测。将阳性菌液提取质粒,命名为pDONR207-HcKAN4。将质粒pDONR207-HcKAN4?载体pTRV2和LR酶按LR反应体系(pDONR207-HcKAN4(80~100ngμL-1):0.5μL,pTRV2(80~100ngμL-1):0.5μL,LR Clonase reaction buffer:0.25 μL)混匀后,过夜连接。转化TOP10感受态细胞,37℃培养,挑取单克隆,菌液PCR检测,菌液送尚亚生物技术公司进行测序,序列比对正确,提取质粒,将其命名为pTRV2-HcKAN4。
1.2.6 农杆菌转化侵染液制备
将pTRV1?pTRV2?pTRV2-HcKAN4质粒分别转化根瘤农杆菌GV3101感受态细胞,挑取单克隆,接种到含50μgmL-1的卡那霉素?50μgmL-1的庆大霉素和50μgmL-1的利福平的液体培养基,28℃培养12h,菌液PCR检测后验证阳性克隆。将验证为阳性的菌液,按1∶100的比例接种到含有卡那霉素?庆大霉素?利福平的150mLLB液体培养基中,28℃培养,以适当体积的重悬液(10mmolL-1MgCl2,10mmolL-1MES以及200μmolL-1乙酰丁香酮)重悬液浓度OD600=0.8。将携带有pTRV1载体重悬液与分别携带pTRV2?pTRV2-HcKAN4的重悬液按体积比1∶1混匀,用于侵染种植7d的红麻真叶。将侵染后的植株置于温度25℃?光/暗周期(14h/10h)的条件下生长,并用防虫网遮盖,根据生长情况浇营养液。
1.2.7 红麻类黄酮合成相关基因鉴定
利用拟南芥类黄酮生物合成相关基因与实验室组装的红麻蛋白质数据库进行BLASTP比对,鉴定得到红麻类黄酮生物合成基因,分别为HcPAL?HcCHS?HcF3’H?HcFLS?HcF3’5’H。通过NCBI-CDD分析红麻类黄酮合成候选基因的蛋白质结构域,再根据实验室前期建立的红麻‘福红952’在不同发育时期不同组织的转录组数据(种子萌发后10d下胚轴?种子萌发后60d茎皮?种子萌发后120d茎皮),分析类黄酮合成相关基因在红麻茎皮中的表达模式。
1.2.8 红麻细胞壁合成相关基因的筛选
根据已发表文献得到红麻木质素合成途径相关基因HcPAL?HcC4H?Hc4CL?HcCOMT?HcHCT?HcCCR?HcCAD?HcC3H?HcF5H,纤维素合成途径相关基因HcCSLD?HcCESA9?HcCESA5?HcCEL6,再根据实验室前期建立的红麻‘福红952’在不同发育时期不同组织的转录组数据,分析红麻发育过程中细胞壁合成相关基因的表达模式。
1.2.9 实时荧光定量PCR分析
以红麻Hc18sRNA为内参基因,使用荧光定量 PCR仪试剂盒(GO Taq qPCR Master Mix (Promega,美国)),在实时荧光定量PCR仪CFX96(BIO-RAD,美国)中进行qRT-PCR扩增。qRT-PCR反应体系为cDNA2.0μL?左引物和右引物各0.7μL?SYBR Green Ⅰ10.0 μL?ddH2O 6.6 μL,扩增程序为95℃预变性5s;95℃变性5s,退火温度为55℃,延伸30s,共40个循环。每个样品设置3个技术重复,采用2-DDCt计算基因的相对表达量。采用Microsoft Excel软件进行数据处理和作图,用 SPSS Statistics 25软件分析显著性。
2 结果与分析
2.1 红麻 HcKAN4 基因克隆与生物学信息分析
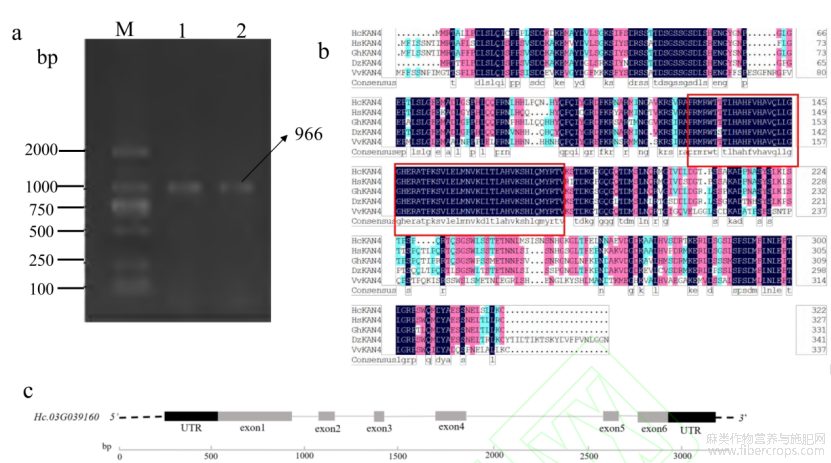
以红麻‘福红952’叶片cDNA为模板,以HcKAN4-F/R为引物克隆获得HcKAN4基因(图1-a),扩增结果和预期片段大小一致。基因结构分析表明(图1-b),HcKAN4基因包含6个外显子?521bp的5′非翻译区(non translated region,UTR)和226bp的3’UTR,编码322个氨基酸。预测蛋白分子量为35.38kD,等电点(pI)为6.90,分子式为C1523H2425N449O490S16。该蛋白氨基酸含量较多的有Ser(41个,12.7%)?Leu(39个,12.1%)?Gly(24个,7.5%)?Thr(20个,6.2%)?Asp(21个,6.5%),氨基酸不包含Pyl和Sec。通过DNAMAN进行蛋白质序列比对分析表明,红麻?棉花?木槿?榴莲和葡萄的KAN4蛋白序列比对都包含一段高度保守的氨基酸序列,属于MYB保守结构域,属于MYB家族(图1-c),其氨基酸序列与棉花?木槿?榴莲和葡萄比较发现都具有较高的同源性,其中与木槿同源性最高,达到87.42%,说明该基因在锦葵科植物中具有较高的保守性。使用MHMM软件对Hc.KAN4蛋白的跨膜结构区进行分析可知,Hc.KAN4整条肽链都存在细胞膜外侧,说明不存在跨膜螺旋(附图1)。通过MEGA7.0软件构建Hc.KAN4蛋白与其他植物KAN4蛋白的系统发育树,探究它们之间的同源关系(附图2)可知,Hc.KAN4与拟南芥和木槿的同源关系较近,与油菜?辣椒的同源关系较远。
图1 红麻HcKAN4基因克隆及序列分析
a:HcKAN4基因克隆电泳图。其中,M表示DL2000DNAMarker,1和2表示克隆产物。b:HcKAN4基因结构示意图。c:HcKAN4蛋白与其他物种同源蛋白序列比对图。其中红框表示MYB保守结构域,HcKAN4表示红麻KAN4蛋白,HsKAN4表示木槿KAN4蛋白,GhKAN4表示棉花KAN4蛋白,DzKAN4表示榴莲KAN4蛋白,VvKAN4表示葡萄KAN4蛋白。
2.2 HcKAN4基因在不同时期不同组织中的表达分析
由图2-a可知,Hc.KAN4基因在旺盛生长期(种子萌发后60d根?茎皮?茎骨和叶)及工艺成熟期(种子萌发后120d茎皮)的表达特征。Hc.KAN4在茎和叶中均有不同程度的表达,在旺盛生长期根和茎骨中表达低,而工艺成熟期(种子萌发后120d)茎皮表达量最高,说明Hc.KAN4基因具有组织特异性。在种子萌发后120d时茎皮中的表达量约为60d的12倍,说明Hc.KAN4基因随着植物生长在茎皮中发挥着重要作用。分析Hc.KAN4基因在不同发育时期的圆叶?三裂叶?五裂叶和七裂叶叶片的表达特征(图2-b)发现,Hc.KAN4基因在R7时具有最高表达量,是R?R3?R5时期的8.56倍?1.21倍?4.86倍,表明随着叶片发育基因表达量总体呈升高的趋势,推测Hc.KAN4基因可能参与红麻叶片的发育。
图2 红麻HcKAN4基因在不同时期不同组织中的表达分析
root-60d?stem-stick-60d?leaf-60d?stem-bark-60d?stem-bark-120d分别代表种子萌发60d后根?茎骨?叶?茎皮?和种子萌发120d后茎皮;R?B3?B5?B7分别代表种子萌发120d后圆叶?三裂叶?五裂叶和七裂叶。
2.3 红麻HcKAN4沉默载体的构建及农杆菌转化
为了沉默目的基因HcKAN4,以TRV作为病毒载体,构建TRV-HcKAN4并转化农杆菌GV3101。以红麻叶片cDNA为模板进行PCR,得到一条与预期大小一致的扩增片段。回收目的片段后通过Gateway技术构建pTRV2-KAN4载体,经冻融法获得转入pTRV1?pTRV2(空载)?pTRV2-KAN4载体的农杆菌GV3101,用特异引物进行菌液PCR鉴定,结果如图3所示,出现符合预期的条带,表明3个TRV-VIGS表达载体成功转入到农杆菌GV3101中。
图3 红麻Hc.KAN4基因重组载体质粒转化后菌液PCR结果
M:DL2000DNAmarker。a:P代表质粒,1~4代表pTRV2-KAN4待测农杆菌;b:1~5代表pTRV1待测农杆菌;c:1~5代表pTRV2(空载)待测农杆菌。
图4 红麻HcKAN4的cDNA和氨基酸序列
红色线标出沉默片段。
2.4 TRV-VIGS诱导HcKAN4基因沉默的效率检测
当沉默HcPDS基因的植株开始出现白化表型,随机选取6株沉默HcKAN4基因的红麻植株,以及长势与沉默植株一致的3株空载处理的红麻植株作为对照。本研究通过qRT-PCR检测TRV-VIGS对红麻叶中HcKAN4基因表达水平的影响(图5-a)发现,VIGS沉默后,与对照相比,6株沉默植株的相对表达量都达到显著性降低,沉默效率达到100%。为证实TRV-VIGS沉默效果是整株植物沉默,随机选取2株沉默HcKAN4植株,取其茎皮和2株空载处理植株作为对照(图5-b)。与对照相比,沉默植株茎皮的相对表达量也出现了降低,沉默效率100%,说明TRV-VIGS是整株沉默且沉默效率良好。
不定芽长至2~3cm,将其转接在附加TDZ2mg/L)?IAA(0.02mg/L)和NAA(0.05mg/L)的MS培养基上,10d后,6个种质全部生根。开始根少且短,植株矮小,1周后,整个三角瓶的底部都布满了根,植株也随着长高长大,从而形成了完整的再生植株。达到显著性降低,沉默效率达到100%。为证实TRV-VIGS沉默效果是整株植物沉默,随机选取2株沉默HcKAN4植株,取其茎皮和2株空载处理植株作为对照(图5-b)。与对照相比,沉默植株茎皮的相对表达量也出现了降低,沉默效率100%,说明TRV-VIGS是整株沉默且沉默效率良好。
图5 HcKAN4基因沉默后红麻植株的沉默效果检测
a:VIGS沉默后叶片HcKAN4基因的qRT-PCR分析;b:VIGS沉默后茎皮HcKAN4基因的qRT-PCR分析。柱上不同小写字母表示同一基因在不同植株表达量差异达到显著水平
2.5 TRV-VIGS诱导HcKAN4基因沉默的效率检测
2.5.1 类黄酮合成相关基因在不同发育时期茎皮中的表达分析
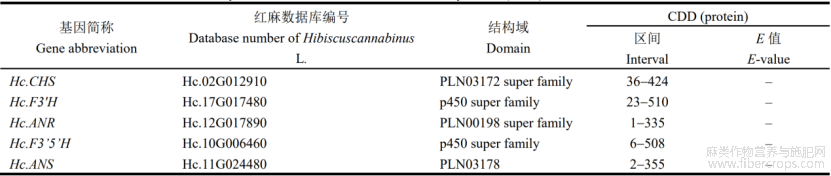
利用拟南芥类黄酮合成相关基因蛋白质序列,通过与红麻蛋白质数据blastp鉴定得到红麻类黄酮合成相关基因,通过NCBI-CDD对这些基因的结构域进一步分析(表1)。由表1可知,Hc.CHS蛋白具有PLN03172superfamily结构域,Hc.F3'H具有p450 super family结构域,Hc.ANS具有PLN03178结构域,Hc.ANR具有PLN 00198 super family结构域,Hc.F3’5’H具有p450 super family结构域。
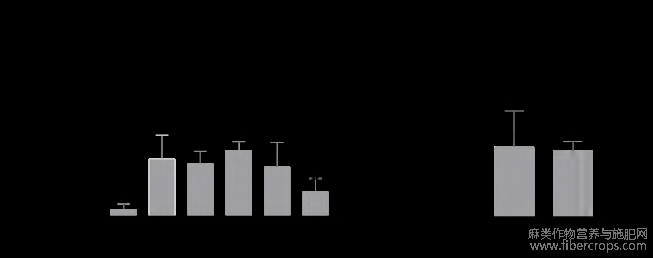
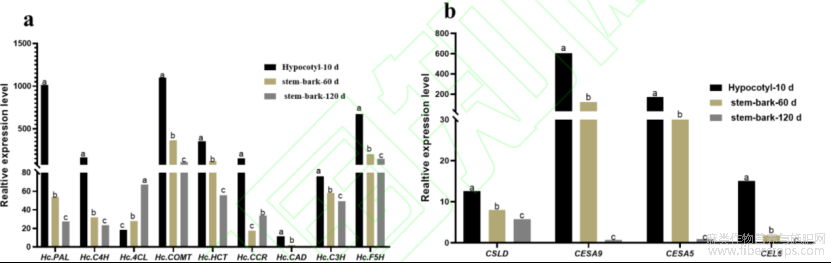
通过对鉴定得到的类黄酮合成基因在红麻不同时期茎皮的电子表达量进行表达模式分析(图6)发现,5个类黄酮合成基因的电子表达量都随着生长时间增加在茎皮中的表达量呈显著性增加的趋势,说明类黄酮在植物茎皮的生长发育中起着重要作用。由图7-a可知,红麻木质素合成相关基因Hc.PAL?Hc.C4H?Hc.COMT?Hc.HCT?Hc.CCR?Hc.CAD?Hc.C3H?Hc.F5H的电子表达量随着植株生长时间的增加呈显著性降低的趋势,只有木质素合成相关基因Hc.CCT的电子表达量先降低后增加的变化;由图7-b可知,红麻纤维素合成相关基因Hc.CSLD?Hc.CESA9?Hc.CESA6?Hc.CEL6的电子表达量随着植物生长时间增加呈显著性降低的趋势。说明随着植株生长时间增加,茎皮中类黄酮合成基因的电子表达量呈显著性增加趋势,而茎皮中的木质素和纤维素合成相关基因呈显著性降低趋势,类黄酮表达量的增加导致了木质素基因和纤维素合成酶基因表达量的减少,由此可说明类黄酮相关基因高表达可能不利于红麻纤维品质的提高。
表1 NCBI(CDD)鉴定红麻类黄酮合成基因及其结构
图6 红麻类黄酮合成相关基因在不同时期茎皮的表达分析
Hypocotyl-10d:种子萌发后10d的下胚轴;stem-bark-60d:种子萌发后60d的茎皮;stem-bark-120d:种子萌发后120d的茎皮。柱上不同小写字母表示同一基因在不同时期表达量差异达到显著水平。
图7 红麻纤维素和木质素合成相关基因在不同时期茎皮的表达量
a:表示木质素合成相关基因在不同时期茎皮的表达量;b:表示纤维素合成相关基因在不同时期茎皮的表达量;Hypocotyl-10d:种子萌发后10d的下胚轴;stem-bark-60d:种子萌发后60d的茎皮;stem-bark-120d:种子萌发后120d的茎皮。柱上不同小写字母表示同一基因在不同时期表达量差异达到显著水平。
2.5.2 HcKAN4基因沉默植株类黄酮合成相关基因的表达分析
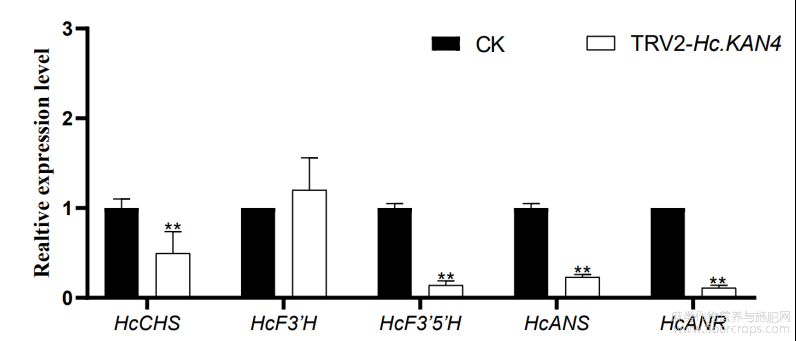
对HcKAN4基因VIGS沉默处理的和对照组的红麻茎皮中的类黄酮合成相关基因进行qRT-PCR检测(图8)发现,HcKAN4基因VIGS沉默植株中,类黄酮合成基因HcF3’H?HcCHS?HcF3’5’H?HcANS?HcANR在茎皮的表达量下调达到极显著水平,分别是对照组的0.51?0.14?0.23?0.11倍。表明HcKAN4基因能够影响红麻类黄酮合成基因的表达,从而影响类黄酮的生物合成。
图8 红麻HcKAN4基因VIGS沉默后类黄酮合成相关基因的qRT-PCR分析
每组数据代表3次技术重复的平均值,误差以SD表示,*表示在0.05概率水平差异显著,**表示在0.01概率水平差异显著。HcCHS:查尔酮合成酶基因;HcF3’H:类黄酮3’-羟化酶基因;HcF3’5’H:类黄酮3’5’-羟化酶基因;HcANS:花青素合成酶基因;HcANR:花青素还原酶基因。
3 讨论
3.1 MYB转录因子在类黄酮和纤维发育中的作用机制
MYB转录因子在类黄酮合成和纤维发育中发挥着重要作用,本研究利用TRV-VIGS技术鉴定了红麻HcKAN4基因在类黄酮合成中的作用。结果显示,在HcKAN4基因沉默后,类黄酮合成基因表达量均显著减少,说明HcKAN4是红麻类黄酮代谢途径中部分酶基因的关键调控因子。进一步分析发现,该基因在红麻中主要在茎?叶中表达而在根中不表达。在油菜中,KAN4基因在根茎叶中都有表达,在根中的表达量显著高于茎?叶,表明该基因同源基因在不同植物存在不同表达模式,具有组织特异性。
多种转录因子对类黄酮生物合成通路中的基因有调控作用,MYB转录因子调控类黄酮的途径也有相关报道。Zhai等发现PbMYB10b可以调控PAs的生物合成,但其功能可以被其他MYB转录因子补充。
Li等发现FhMYB5和bHLH转录因子基因FhTT8L和FhGL3L共同作用时,类黄酮合成基因被激活。Zhu等从菊花中发现R2R3-MYB转录因子的CmMYB8基因对木质素和类黄酮化合物的合成起到负调控作用。在棉花中发现R2R3-MYB基因与纤维品质有遗传相关性。本研究发现,沉默HcKAN4基因显著下调红麻类黄酮化合物合成酶基因的转录水平,而正向调控木质素合成相关酶基因的表达。这些结果表明,红麻HcKAN4沉默后可能抑制了黄酮类化合物的合成而促进纤维素含量积累。而提高纤维素含量是改良纤维品质的关键途径,因此,这为改善红麻纤维品质的分子育种提供了新的研究思路。
研究发现,基关于HcKAN4基因与类黄酮合成相关基因HcF3’H?HcCHS?HcF3’5’H?HcANS?HcANR之间的调控关系,是通过转录调控还是蛋白结合等方式实现,目前还没有相关文献报道。生物信息学分析结果显示,红麻类黄酮合成相关基因HcF3’5’H(Hc.10G006460)启动子上游1959bp处存在1个HcKAN4的结合元件(TTTTTACGGTTA),其功能是MYB binding site involved in flavonoid biosynthetic genes regulation。多项研究显示,该元件是MYB转录因子家族调控下游靶基因的核心元件。因此,转录因子HcKAN4可能结合HcF3’5’H启动子进而调控类黄酮的合成。具体调控机制有待进一步深入研究。
3.2 TRV-VIGS的影响因素
VIGS具有不通过稳定遗传转化就降低目的基因表达水平的特征,从而可以研究目的基因表达水平降低后对作物表型的影响。VIGS的沉默效率受多种因素影响,如载体种类?侵染方法?侵染时间等。VIGS在不同的植物中沉默效率也有较大差别,Jia等发现草莓CHS基因的沉默效率达80%左右,而本研究中沉默Hc.KAN4基因沉默效率为100%。现阶段VIGS研究通常沉默一个基因,从而研究基因功能作用。李玉霞等通过共沉默GbCHI?GbDFR和GbF3’H来研究在海岛棉中的抗枯萎病发现,共沉默相对于单独沉默GbF3’H的作用效果有显著提升。通过共沉默来探讨红麻HcKAN4基因与类黄酮合成相关基因HcF3’H?HcCHS?HcF3’5’H?HcANS?HcANR之间的调控关系,有待进一步研究。
4 结论
HcKAN4基因表达具有组织特异性,在茎?叶中的表达量显著高于根中。TRV-KAN4的沉默效率良好,且沉默植株中类黄酮合成相关基因的表达量与对照相比显著下降,通过对植株在相同组织不同时期的表达量发现,类黄酮合成相关基因的表达量呈上升趋势,而纤维素和木质素相关基因的表达量呈下降趋势。推测该转录因子可以影响类黄酮的生物合成,进而影响红麻的纤维发育。
参考文献
[1]Afzal M Z, Ibrahim A K, Xu Y, Niyitanga S, Li Y, Li D, Yang X, Zhang L. Kenaf (Hibiscus Cannabinus L.) breeding. J Nat Fibers, 2022, 19: 4063–4081.
[2]Sreenivas H T, Krishnamurthy N, Arpitha G R. A comprehensive review on light weight kenaf fiber for automobiles. Intl J Lightweight Mater Manufact, 2020, 3: 328–337.
[3]Saba N, Paridah M T, Jawaid M. Mechanical properties of kenaf fiber reinforced polymer composite: a review. Constr Build Mater, 2015, 76: 87– 96.
[4]Mahmood A, Sapuan S M, Karmegam K, Abu A S. Design and development of kenaf fiber-reinforced polymer composite polytechnic chairs.Asian J Agric Biol, 2018, 6: 62–65.
[5]张迪,赵文军,马丽娟,柴友荣.原花青素的性质、功能、纯化和利用.安徽农学通报, 2009, 15(1): 35–39.Zhang D, Zhao W J, Ma L J, Chai Y R. Properties, functions, purification and utilization of proanthocyanidins. Anhui Agric Sci Bull, 2009, 15(1): 35–39 (in Chinese with English abstract).
[6]刘淑华,臧丹丹,孙燕,李金霞,赵恒田.花青素生物合成途径及关键酶研究进展.土壤与作物, 2022, 11: 336–346.Liu S H, Zang D D, Sun Y, Li J X, Zhao H T. Research advances on biosynthesis pathway of anthocyanins and relevant key enzymes. Soil Crop, 2022, 11: 336–346 (in Chinese with English abstract).
[7]赵文军,张迪,马丽娟.原花青素的生物合成途径、功能基因和代谢工程.植物生理学通讯, 2009, 45: 509–519.Zhao W J, Zhang D, Ma L J. Biosynthetic pathway, functional genes and metabolic engineering of proanthocyanidins. Plant Physiol J, 2009, 45: 509–519 (in Chinese with English abstract).
[8]苏全胜,王爽,孙玉强,梅俊,柯丽萍.植物原花青素生物合成及调控研究进展.中国细胞生物学学报, 2021, 43(1): 219–229.Su Q S, Wang S, Sun Y Q, Mei J, He L P. Advances in biosynthesis and regulation of plant proanthocyanidins. Chin J Cell Biol, 2021, 43(1): 219–229 (in Chinese with English abstract).
[9]Chen L H, Hu B, Qin Y H, Hu G B, Zhao J T. Advance of the negative regulation of anthocyanin biosynthesis by MYB transcription factors.Plant Physiol Biochem, 2019, 136: 178–187.
[10]安秀红,张修德,陈可钦,刘肖娟,郝玉金,程存刚.苹果MdMYB9、MdMYB11表达及其蛋白互作分析. 中国农业科学, 2015, 11: 2208– 2216.An X H, Zhang X D, Chen K Q, Liu X J, Hao Y J, Cheng C G. Expression and protein interaction analysis of MdMYB9 and MdMYB11 in apple.Sci Agric Sin, 2015, 11: 2208–2216 (in Chinese with English abstract).
[11]Peter C P. Molecular controls of proanthocyanidin synthesis and structure: Prospects for genetic engineering in crop plants. J Agric Food Chem, 2018, 66: 9882–9888.
[12]Nesi N, Jond C, Debeaujon I, Caboche M, Lepiniec L. The Arabidopsis TT2 gene encodes an R2R3 MYB domain protein that acts as a key determinant for proanthocyanidin accumulation in developing seed. Plant Cell, 2001, 13: 2009–2114.
[13]Akhter D, Qin R, Nath U K, Jin X L, Shi C H. A rice gene, OsPL, encoding a MYB family transcription factor confers anthocyanin synthesis, heat stress response and hormonal signaling. Gene, 2019, 699: 62–72.
[14]Liu C C, Jun J H, Dixon R A. MYB5 and MYB14 play pivotal roles in seed coat polymer biosynthesis in Medicago truncatula. Plant Physiol, 2014, 165: 1424–1439.
[15]Zhou M L, Sun Z M, Ding M Q, Logacheva M D, Kreft I, Wang D, Yan M L, Shao J R, Tang Y X, Wu Y M, Zhu X M. FtSAD2 and FtJAZ1 regulate activity of the FtMYB11 transcription repressor of the phenylpropanoid pathway in Fagopyrum tataricum. New Phytol, 2017, 216: 814– 828.
[16]Gao P, Li X A, Cui D J , Wu L M, Parkin I, Gruber M Y. Margaret Y G. A new dominant Arabidopsis transparent testa mutant, sk21-D, and modulation of seed flavonoid biosynthesis by KAN4. Plant Biotechnol J, 2010, 8: 979–993.
[17]Purkayastha A, Dasgupta I. Virus-induced gene silencing: a versatile tool for discovery of gene functions in plants. Plant Physiol Biochem, 2009, 47: 967–976.
[18]Chantreau M, Chabbert B, Billiard S, Billiard S, Hawkins S, Neutelings G. Functional analyses of cellulose synthase genes in flax (Linum usitatissimum) by virus-induced gene silencing. Plant Biotechnol J, 2015, 13: 1312–1324.
[19]王心宇,吕坤,蔡彩平,徐军,郭旺珍. TRV 病毒介导的基因沉默体系在棉花中的建立及应用. 作物学报, 2014, 40: 1356–1363.Wang X Y, Lü K, Cai C P, Xu J, Guo W Z. Establishment and application of TRV-Mediated virus-induced gene silencing in cotton. Acta Agron Sin, 2014, 40: 1356–1363 (in Chinese with English abstract).
[20]Liu Y L, Schiff M, Dinesh-Kumar S P. Virus-induced gene silencing in tomato. Plant J, 2002, 31: 777–786.
[21]Zhang L W, Wan X B, Xu J T, Lin L H, Qi J M. De novo assembly of kenaf (Hibiscus cannabinus) transcriptome using Illumina sequencing for gene discovery and marker identification. Mol Breed, 2015, 35: 192.
[22]Ryu J, Kwon S J, Sung S Y, Kim W J, Kim D, Ahn J W, Kim J B, Kin S, Ha B K, Kang S Y. Molecular cloning, characterization, and expression analysis of lignin biosynthesis genes from kenaf (Hibiscus cannabinus L.). Genes Genomics, 2015, 38: 59–67.
[23]涂礼莉,谭家福,郭凯,李中华,张献龙.类黄酮代谢途径与棉花纤维发育. 中国科学: 生命科学, 2014, 44: 758–765.Tu L L, Tan J Q, Guo K, Li Z H, Zhang X L. Flavonoid pathway in cotton fiber development. Sci Sin Vitae, 2014, 44: 758–765 (in Chinese with English abstract).
[24]郭光艳,柏峰,刘伟,秘彩莉. 转录因子对木质素生物合成调控的研究进展. 中国农业科学, 2015, 48: 1277–1287.Guo G Y, Bai F, Liu W, Mi C L. Advances in research of the regulation of transcription factors of lignin biosynthesis. Sci Agric Sin, 2015, 48: 1277–1287(in Chinese with English abstract).
[25]Zhang L W, Xu Y, Zhang X T, Ma X K, Zhang L L, Liao Z Y, Zhang Q, Wan X B, Chen Y, Zhang J S, Li D X, Zhang L M, Xu J T, Tao A F, Lin L H, Fang P P, Chen S, Qi R, Xu X M, Qi J M, Ming R. The genome of kenaf (Hibiscus cannabinus L.) provides insights into bast fiber and leaf shape biogenesis. Plant Biotechnol J, 2020, 18: 1796–1809.
[26]Qin G J, Gu H Y, Ma L G, Peng Y B, Deng X W, Chen Z L, Qu L J. Disruption of phytoene desaturase gene results in albino and dwarf phenotypes in Arabidopsis by impairing chlorophyll, carotenoid, and gibberellin biosynthesis. Cell Res, 2007, 17: 471–482.
[27]高国应,伍小方,黄伟,周定港,张大为,周美亮,张凯旋,严明理.芥菜型油菜 BjuB.KAN4 基因调控类黄酮的途径. 作物学报, 2020,46: 1322–1331.Gao G Y, Wu X F, Huang W, Zhou D G, Zhang D W, Zhou M L, Zhang K X, Yan M L. Regulation of flavonoid pathway by BjuB.KAN4 gene in Brassica juncea. Acta Agron Sin, 2020, 46: 1322–1331 (in Chinese with English abstract).
[28]Zhai R, Wang Z M, Zhang S W, Meng G, Song L Y, Wang Z G, Li P M, Ma F W, Xu L F. Two MYB transcription factors regulate flavonoid biosynthesis in pear fruit (Pyrus bretschneideri Rehd.). J Exp Bot, 2016, 67: 1275–1284.
[29]Li Y Q, Shan X T, Zhou L D, Gao R F, Yang S, Wang S C, Wang L, Gao X. The R2R3-MYB factor FhMYB5 from freesia hybrid a contributes to the regulation of anthocyanin and proanthocyanidin biosynthesis. Front Plant Sci, 2018, 9: 1935.
[30]Zhu L, Guan Y X, Zhang Z H, Song A P, Chen S M, Jiang J F, Chen F D. CmMYB8 encodes an R2R3 MYB transcription factor which represses lignin and flavonoid synthesis in chrysanthemum. Plant Physiol Biochem, 2020, 149: 217–224.
[31]Wang N H, Ma Q, Ma J J, Pei W F, Liu G Y, Cui Y P, Wu M, Zang X S, Zhang J F, Yu S X, Ma L J, Yu J W. A comparative genome-wide analysis of the R2R3-MYB gene family among four gossypium species and their sequence variation and association with fiber quality traits in an interspecific G. hirsutum × G. barbadense population. Front Genet, 2019, 10: 741.
[32]Chapman S, Kavanagh T, Baulcombe D. Potato virus X as a vector for gene expression in plants. Plant J, 1992, 2: 549–557.
[33]Liu Y L, Schiff M, Dinesh-Kumar S P. Virus-induced gene silencing in tomato. Plant J, 2002, 31: 777–786.
[34]李萌晗. 油棕病毒诱导的基因沉默(VIGS)体系的建立,优化和验证研究. 海南大学硕士学位论文,海南海口, 2022.Li M H. Establishment, Optimization and Validation of Oil Palm Virus-induced Gene Silencing (VIGS) System. MS Thesis of Hainan University, Haikou, Hainan, China, 2022 (in Chinese with English abstract).
[35]Jia H F, Lu D, Sun J H, Li C L, Xing Y, Qin L, Shen Y Y. Type 2C protein phosphatase ABI1 is a negative regulator of strawberry fruit ripening.J Exp Bot, 2013, 64: 1677–1687.
[36]李玉霞,曲延英,艾海提·艾合买提,王慧敏,黄启秀,陈琴,陈全家. 通过GbF3'H基因单独沉默及其与GbCHI和GbDFR基因共沉默研究其在海岛棉中抗枯萎病功能. 棉花学报, 2020, 32(1): 1–10.Li Y X, Qu Y Y, Aihaiti A, Wang H M, Huang Q X, Chen Q, Chen Q J. Through single silencing GbF3'H gene and its co-silencing with GbCHI and GbDFR genes to study their function in resistance to fusarium wilt in Gossypium barbadense. Cotton Sci, 2020, 32(1): 1–10 (in Chinese with English abstract).
文章摘自:吴法轩,李秦,杨昕,李新根,徐建堂,陶爱芬,方平平,祁建民,张立武.红麻HcKAN4基因克隆、表达及在类黄酮合成中的功能.植物学报,20230928.0957.002