摘 要:工业大麻miR156基因家族在碱性盐胁迫响应过程中发挥重要的调控作用,为探究工业大麻miR156基因家族响应NaHCO3胁迫的分子机制,以盐碱耐受型工业大麻品种火麻一号为供试材料,利用生物信息学和qRT-PCR方法,对miR156基因家族进行分析。结果表明,工业大麻miR156家族基因与芦笋、野草莓、苹果和大豆的亲缘关系较近。2条工业大麻pre-miR156序列,产生一条相同的miR156成熟体,且成熟体的保守性较高。在NaHCO3胁迫后的24h内,工业大麻miR156基因表达量随胁迫时间的延长呈先升高后降低再升高的趋势。该结果为进一步研究工业大麻miR156基因家族及其靶基因的调控网络提供理论依据。
关键词:工业大麻;miR156基因家族;NaHCO3胁迫
MicroRNAs(miRNA)是真核生物中发现的一类内源性具有调控功能的非编码RNA,长约20~25个核苷酸。作为一种进化上保守的miRNA,在增强植物对逆境胁迫耐受性方面起着重要作用[1]。随着高通量测序(Highthroughtputsequencing)技术发展,可找出数以千万条miRNA。在草本植物中,Akdogan等[2]鉴定了小麦根和叶中响应干旱的miRNA,干旱处理后,miR159、miR160、miR166、miR169、miR172、miR395、miR396、miR408、miR472、miR477、miR482、miR1858、miR2118和miR5049在小麦中差异表达显著。Sun等[3]鉴定了萝卜中响应NaCl胁迫的miRNA,发现136个已知miRNA(属于43个miRNA家族)和68个潜在的新miRNA(属于51个miRNA家族),49个已知miRNA和22个新miRNA在盐胁迫下差异表达,这些miRNA的靶基因主要注释在信号感知和转导、离子稳态调控、基本代谢过程、次生胁迫响应以及对植物生长发育调控等通路。在木本植物中,Pan等[4]在桦树中鉴定了响应热胁迫的miRNA,发现206个miRNA,其中98个(57个已知miRNA和41个新miRNA)在高温胁迫下差异表达。上述研究从组学角度揭示了miRNA在非生物胁迫过程中的调控作用,并未针对某个特定的miRNA展开研究。
miRNA156最先发现于拟南芥中,约由20个核苷酸组成,结构保守,通过靶向SPL基因参与植物生长发育[5]。越来越多证据表明,高度保守的miRNA156/SPL模式似乎平衡了植物生长发育和胁迫应答。过表达miRNA156可以增加苜蓿和水稻根部生物量,进而提高其盐碱耐受性[6];最终,还可以提高苜蓿、柳枝稷等饲料用作物产量和品质[7-8]。因此,有人认为组成型表达miRNA156可提高植物对非生物胁迫耐受性[9-10]。当植物受到逆境胁迫时,盐胁迫后1h是柽柳(Tamarixchinensis)中miR156响应盐胁迫调控的关键时间点[11]。诱导ZmmiR156表达能提高烟草对干旱和盐胁迫耐受性[12]。盐胁迫早期,菊花miRNA156表达量先升高后降低,进而可能通过调控CmSPL13或者其他靶基因参与盐胁迫早期应答[13]。但在白桦树、苹果树等木本植物中,过表达miRNA156材料的耐盐性呈降低趋势[14-15]。故工业大麻miRNA156在响应碱性盐胁迫早期应答过程中的表达模式仍有待进一步探究。
以上研究报道了miRNA156对植物盐碱耐受性的调控作用,但对其分子调控机制研究尚不深入。尤其是在工业大麻中,miRNA156如何感知外界胁迫信号,并通过调控下游相关基因响应NaHCO3胁迫的分子机制研究鲜有报道。为此,本研究采用生物信息学和qRT-PCR方法,对工业大麻miR156家族的基因前体序列和成熟体序列的保守性、二级结构等进行预测,并对其在NaHCO3胁迫下的表达模式进行分析,旨在丰富miRNA156基因家族的分子调控网络和基因功能研究,为工业大麻的分子育种提供基因资源和理论参考。
1.材料与方法
1.1材料
以耐受型品种火麻一号为试验材料(火麻一号种子由黑龙江省科学院大庆分院提供)。待幼苗生长到3对真叶时,在培养液中加入NaHCO3,使其浓度达到100mmol/L,对照组不加NaHCO3。分别在胁迫0、0.5、1、6、12、24h采集长势一致的幼苗根部样品,于-80℃保存备用[16]。植物根部总RNA提取、cDNA合成和定量PCR试剂盒均购自天根生化科技有限公司。
1.2方法
1.2.1miRNA156成熟体和前体序列分析
采用数据库miRBase(http://www.miRbase.org)对工业大麻中新发现的miRNA进行比对,获取相似度高的已知miRNA156家族前体和成熟体序列。采用MEGA5软件进行前体和成熟体序列的进化关系分析,使用miRDeep2软件预测新miRNA的前体序列和结构,采用WebLogo3(http://weblogo.threeplusone.com/)绘制miR156家族序列保守性Logo图。
1.2.2工业大麻miRNA156成熟体qRT-PCR
以cDNA作为模板,使用基因特异性引物进行PCR扩增,使用U6作为miRNA的内参基因,采用PowerqPCRPreMix(Genecopoeia)试剂盒,样品设置3次重复。反应条件:95℃,10min;(95℃,10s;60℃,40s)×40循环;(95℃,15s;60℃,60s;95℃,30s;60℃,15s)。用2-△△Ct法评价基因的相对表达量[17]。
表1qRT-PCR引物序列
2.结果与分析
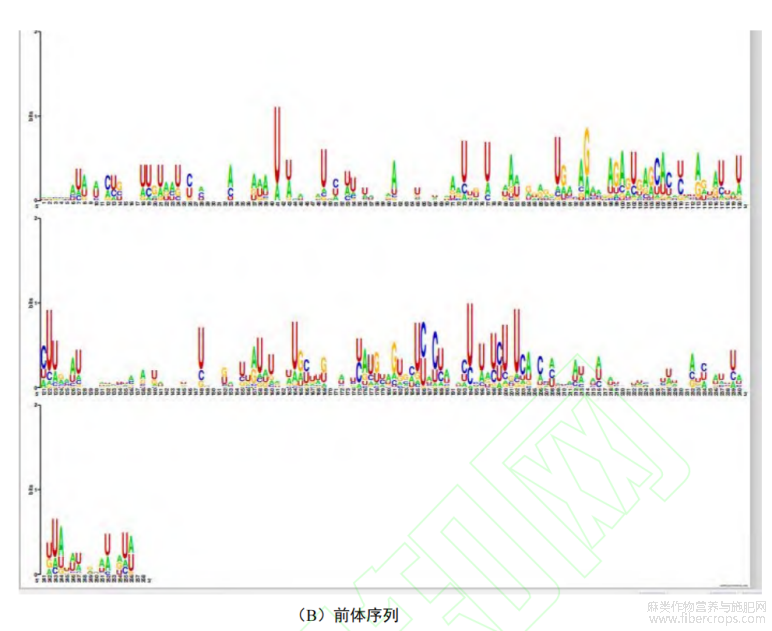
利用软件miRDeep2对novel_miR_75和novel_miR_179的候选前体序列进行预测,红色为成熟序列,黄色为环状结构,紫色为star序列(图1(A)),前体序列均可形成较为稳定的二级茎环结构,且成熟体产生于前体的5’端上臂,保守性较强。从图1(B)中可以看出,成熟序列相同或高度相似,花椒、烟草、刺头朝鲜蓟、大豆的miR156成熟体序列非常保守,而工业大麻miR156与其他植物miR156相比,在3’端有2个核苷酸不同。
图1工业大麻pre-miR156的二级结构预测和成熟体序列比对
由图2可知,前体序列的89~110和175~216位点的碱基保守性较高,其他位点碱基保守性较低。成熟体序列基本产生于前体序列的89~110位点。12个miR156成熟体中12个碱基完全保守,8个保守性较高,3个碱基保守性较低。
图2miRNA156家族成熟序列保守性分析
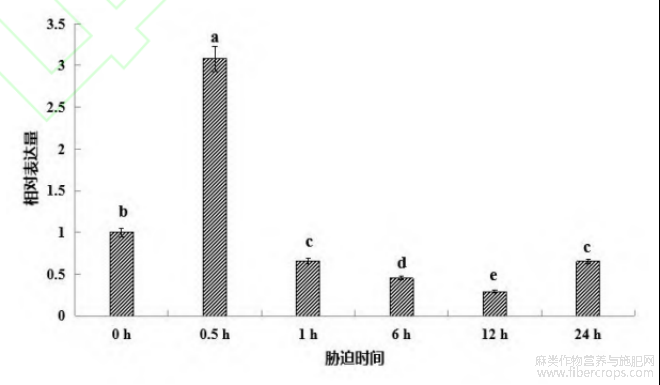
工业大麻受NaHCO3胁迫后,根部不同时间点miRNA156表达变化规律显示(图3),miRNA156的表达量随胁迫时间的延长呈先升高后降低再升高的趋势。0.5h时工业大麻根部miRNA156的表达量最高,较对照增加了2.1倍。
图3NaHCO3胁迫下工业大麻miRNA156表达模式
注:不同小写字母代表差异显著(p<0.05)。
系统进化树结果显示:成熟体miRNA中,novel_miR_179和苹果(Malusdomestica)的miR156m聚在一起,novel_miR_75与大豆(Glycinemax)的miR156u聚在一起(图4(A));miRNA前体序列中,novel_miR_179和芦笋(Asparagusofficinalis)的miR156a聚在一起,novel_miR_75与野草莓(Fragariavesca)的miR156h聚在一起(图4(B))。
图4工业大麻miR156家族系统进化树分析
3.讨论
在植物miRNA的研究中,已在72种植物中鉴定出7385种成熟miRNA和6150种前体miRNA[18]。其中,miR156家族是植物中数量最多且高度保守的成员之一,在根、茎、叶的 形态发生、花的发育等方面起着重要作用,同时在抗逆性等方面起着至关重要的调控作用[19]。在水稻[20]、萱草[21]、苜蓿[22]、花生[23]等多种植物受到逆境胁迫时,均发现miR156具有重要的调控作用,且过表达miR156增加了植物对逆境胁迫的耐受性[10]。
通常miRNA的表达水平与外界环境改变有一定相关性[24-25],受NaHCO3胁迫,工业大麻miR156的表达量随胁迫时间的延长呈先升高后降低再升高的趋势。NaHCO3胁迫0.5h时工业大麻根部miR156的表达量最高,较对照增加了2.1倍,该规律在柽柳中也有类似报道[11]。通过系统进化树分析,发现工业大麻novel_miR_179和苹果的miR156m聚在一起,novel_miR_75与大豆miR156u聚在一起,这些已知的miR156基因在植物生长发育方面具有重要的调控作用[26-27]。在pre-miRNA序列的进化树中,novel_miR_179和芦笋miR156a聚在一起,novel_miR_75与野草莓miR156h聚在一起,这些已知的miR156基因在调控开花与性别分化方面具有重要作用[28-29]。可见。miR156在植物生长发育和抗逆性方面具有重要的调控作用[7,30]。故推测工业大麻novel_miR_75和novel_miR_179在抗逆性调控方面也具有重要的作用。
相同或者相似的成熟体miRNA可能来源于不同的前体miRNA[31]。本研究中,novel_miR_75和novel_miR_179的候选前体来源于不同的染色体,且候选前体具有不同的二级结构,说明工业大麻miR156基因家族前体序列具有较大差异。novel_miR_75和novel_miR_179的成熟体均产生于前体的5’端上臂,且成熟体序列的同源性较高,说明不同的前体miRNA可能产生了相同的成熟体miR156,该规律在辣椒中也有类似报道[32]。
目前,从生理特性、农艺性状及产量性状等角度揭示了工业大麻品种间的盐碱耐受性差异[33],但从miRNA角度的研究鲜有报道。miR156及其靶基因SPL转录因子可用于改良植物性状,进而影响抗逆性和产量[6,34-35]。本研究通过生物信息学和qRT-PCR方法分析了工业大麻miR156基因对NaHCO3胁迫的响应,为后续研究miR156及其靶基因间的调控网络和工业大麻盐碱耐受性提供了理论参考。
参考文献
[1]Wani S H, Kumar V, Khare T, et al. miRNA applications for engineering abiotic stress tolerance in plants[J]. Biologia, 2020, 75(7): 1063- 1081.
[2]Akdogan G, Tufekci E D, Uranbey S, et al. miRNA-based drought regulation in wheat[J]. Funct Integr Genomics, 2016, 16(3): 221-233.
[3]SUN X C, XU L, WANG Y, et al. Identification of novel and salt-responsive miRNAs to explore miRNA-mediated regulatory network of salt stress response in radish (Raphanus sativus L.)[J]. BMC Genomics,2015, 16(1): 1- 16.
[4]PAN Y, NIU M Y, LIANG J S, et al. Identification of heat-responsive miRNAs to reveal the miRNA- mediated regulatory network of heat stress response in Betula luminifera[J]. Trees, 2017, 31(5): 1635- 1652.
[5]Reinhart B J, Weinstein E G, Rhoades M W, et al. MicroRNAs in plants[J]. Genes & development, 2002, 16(13): 1616- 1626.
[6]Jerome Jeyakumar J M, AliA, Wang W-M, et al. Characterizing the role of the miR156-SPL network in plant development and stress response[J]. Plants, 2020, 9(9): 1206.
[7]Arshad M, Gruber M Y, Wall K, et al. An insight into microRNA156 role in salinity stress responses of alfalfa[J]. Frontiers in Plant Science, 2017, 8: 356.
[8]FU C X, Sunkar R, ZHOU C E, et al. Overexpression of miR156 in switchgrass (Panicum virgatum L.) results in various morphological alterations and leads to improved biomass production[J]. Plant Biotechnol Journal, 2012, 10(4): 443-452.
[9]SUN Z X, SU C, YUN J X, et al. Genetic improvement of the shoot architecture and yield in soya bean plants via the manipulation of GmmiR156b[J]. Plant BiotechnolJ, 2019, 17(1): 50-62.
[10]CUI LG, SHAN JX, SHIM, et al. The miR156‐ SPL 9‐DFR pathway coordinates the relationship between development and abiotic stress tolerance in plants[J]. The Plant Journal, 2014, 80(6): 1108- 1117.
[11]WANG J W, YE YY, XU M, et al. Roles of the SPL gene family and miR156 in the salt stress responses of tamarisk (Tamarix chinensis)[J]. BMC Plant Biology, 2019, 19(1): 1- 11.
[12]KANG T, YU CY, LIU Y, et al. Subtly manipulated expression of ZmmiR156 in tobacco improves drought and salt tolerance without changing the architecture of transgenic plants[J]. Frontiers in Plant Science,2020, 10: 1664.
[13]秦梅, 田心宇, 于凯丽, 等. miR156 调控菊花逆境响应与开花的表达特性研究[J]. 西北植物学报,2023, 43(3): 374-381.
[14]MA Y, XUE H, ZHANG F, et al. The miR156/SPL module regulates apple salt stress tolerance by activating MdWRKY100 expression[J]. Plant Biotechnol Journal, 2021, 19(2): 311-323.
[15]颜斌. BpmiR156 对白桦生长发育的影响及抗逆性分析[D]. 哈尔滨: 东北林业大学, 2019.
[16]CAO K, SUN Y F, HAN C W, et al. The transcriptome of saline-alkaline resistant industrial hemp (Cannabis sativa L.) exposed to NaHCO3 stress[J]. Industrial Crops and Products, 2021, 170: 113766.
[17]Livak KJ, SchmittgenT D. Analysis of relative gene expression data using real-time quantitative PCR and the 2− ΔΔCT method[J]. Methods, 2001, 25(4): 402-408.
[18]WANG Y J, LUO Z, ZHAO X A, et al. Superstar microRNA, miR156, involved in plant biological processes and stress response: A review[J]. Scientia Horticulturae, 2023, 316: 112010.
[19]刘宝宝, 孟桂智, 刘祖江, 等. 苜蓿 miR156 及其靶基因生物信息学分析[J]. 西南农业学报: 2023,36(7): 1385- 1392.
[20]Nguyen D Q, Nguyen N L, Nguyen V T, et al. Comparative analysis of microRNA expression profiles in shoot and root tissues of contrasting rice cultivars (Oryza sativa L.) with different salt stress tolerance [J].PLoS One, 2023, 18(5): e0286140.
[21]ZHOU B, GAO X, ZHAO F. Integration of mRNA and miRNA Analysis Reveals the Post-Transcriptional Regulation of Salt Stress Response in Hemerocallis fulva[J]. International Journal of Molecular Sciences,2023, 24(8): 7290.
[22]Sarri E, Kapazoglou A, Gerakari M, et al. Expression Profile of Selected Genes Involved in Na+Homeostasis and In Silico miRNA Identification in Medicago sativa and Medicago arborea under SalinityStress[J]. Stresses, 2023, 3(1): 331-339.
[23]徐媛, 陈锦玲, 陈玉梅, 等. 干旱胁迫下花生转录组与 miRNA 测序及相关基因的表达[J]. 贵州农业科学, 2021, 49(1): 1-9.
[24]Ma Z R, Coruh C, Axtell M J. Arabidopsis lyrata small RNAs: transient MIRNA and small interfering RNA loci within the Arabidopsis genus[J]. The Plant Cell, 2010, 22(4): 1090- 1103.
[25]Tang G L. Plant microRNAs: an insight into their gene structures and evolution[C]. Seminars in cell & developmental biology. Academic Press, 2010, 21(8): 782-789.
[26]Turner M, Yu O, Subramanian S. Genome organization and characteristics of soybean microRNAs[J].BMC Genomics, 2012, 13: 1- 16.
[27]XIA R, ZHU H, AnY Q, et al. Apple miRNAs and tasiRNAs with novel regulatory networks[J]. Genome biology, 2012, 13(6): 1- 18.
[28]Harkess A, ZHOU J S, XU C Y, et al. The asparagus genome sheds light on the origin and evolution of a young Y chromosome[J]. Nature communications, 2017, 8(1): 1279.
[29]XIA R, YE S Q, LIU ZR, et al. Novel and recently evolved microRNA clusters regulate expansive F-BOX gene networks through phased small interfering RNAs in wild diploid strawberry[J]. Plant Physiology, 2015, 169(1): 594-610.
[30]ZHENG C F, YE M X, SANG M M, et al. A regulatory network for miR156-SPL module in Arabidopsis thaliana [J]. International Journal of Molecular Sciences, 2019, 20(24): 6166.
[31]Baker C C, Sieber P, Wellmer F, et al. The early extra petals1 mutant uncovers a role for microRNAmiR164c in regulating petal number in Arabidopsis [J]. Current Biology, 2005, 15(4): 303-315.
[32]黄渺, 宋泽洋, 蔡骏一, 等. 辣椒 miR169 家族在非生物胁迫下的表达及启动子顺式作用元件分析[J]. 西部林业科学, 2023, 52(1): 165- 174.
[33]曹焜, 孙宇峰, 张晓艳, 等. 盐碱胁迫对工业大麻生长发育的影响[J]. 种子, 2022, 41(11): 37-46.
[34]WU G, Poethig R S. Temporal regulation of shoot development in Arabidopsis thaliana by miR156 and its
target SPL3 [J]. 2006, 133 (18): 3539–3547.
[35] 张昱, 张莉, 张静, 等. miR156-SPL 分子模块调控植物生长发育[J]. 分子植物育种: 2022, 5: 1-22.
文章摘自:曹焜, 孙宇峰, 张晓艳, 赵越, 边境, 朱浩, 王盼, 韩承伟, 孙凯旋, 王晓楠. 工业大麻miR156基因家族在NaHCO_(3)胁迫下的表达模式及生物信息学分析[J]. 中国麻业科学, 1-9.